Lección 7 Comparación de infectadas anteparto y periparto
Clasificamos en infección anteparto y periparto comparando las variables “EG_TOTAL_PARTO” (edad gestacional en el momento del parto) y “EDAD.GEST.TOTAL” (edad gestacional en el momento del diagnóstico): diagnósticos realizados 3 o más días antes del parto se consideran anteparto, los diagnósticos realizados 2 días antes del parto en adelante se consideran periparto. Las diferencias se calculan usando las edades gestacionales.
Casos$Dif=round((Casos$EG_TOTAL_PARTO-Casos$EDAD.GEST.TOTAL)*7)
Casos$PreP=rep(NA,n_I)
Casos$PreP[Casos$Dif>2]=1 #Diagnóstico de covid anteparto
Casos$PreP[Casos$Dif<=2]=0
n_IAP=table(Casos$PreP)[2]
n_IPP=table(Casos$PreP)[1]
ColumnesP=c("Infectadas anteparto (N)", "Infectadas anteparto (%)","Infectadas periparto (N)", "Infectadas periparto (%)","No infectadas (N)", "No infectadas (%)")
CasosGM$Dif=round((CasosGM$EG_TOTAL_PARTO-CasosGM$EDAD.GEST.TOTAL)*7)
CasosGM$PreP=rep(NA,dim(CasosGM)[1])
CasosGM$PreP[CasosGM$Dif>2]=1 #Diagnóstico de covid anteparto
CasosGM$PreP[CasosGM$Dif<=2]=07.1 Diferencias entre edades gestacionales en el momento del parto y edades gestacionales en el momento del diagnóstico
data =data.frame(
name=rep("Infectadas",length(as.numeric(Casos$Dif))),
Diferencias=as.numeric(Casos$Dif)
)
data %>%
ggplot( aes(x=name, y=Diferencias, fill=name)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
#theme_ipsum() +
theme(
legend.position="none",
plot.title = element_text(size=11)
) +
xlab("")+
ylab("Diferencias (días)")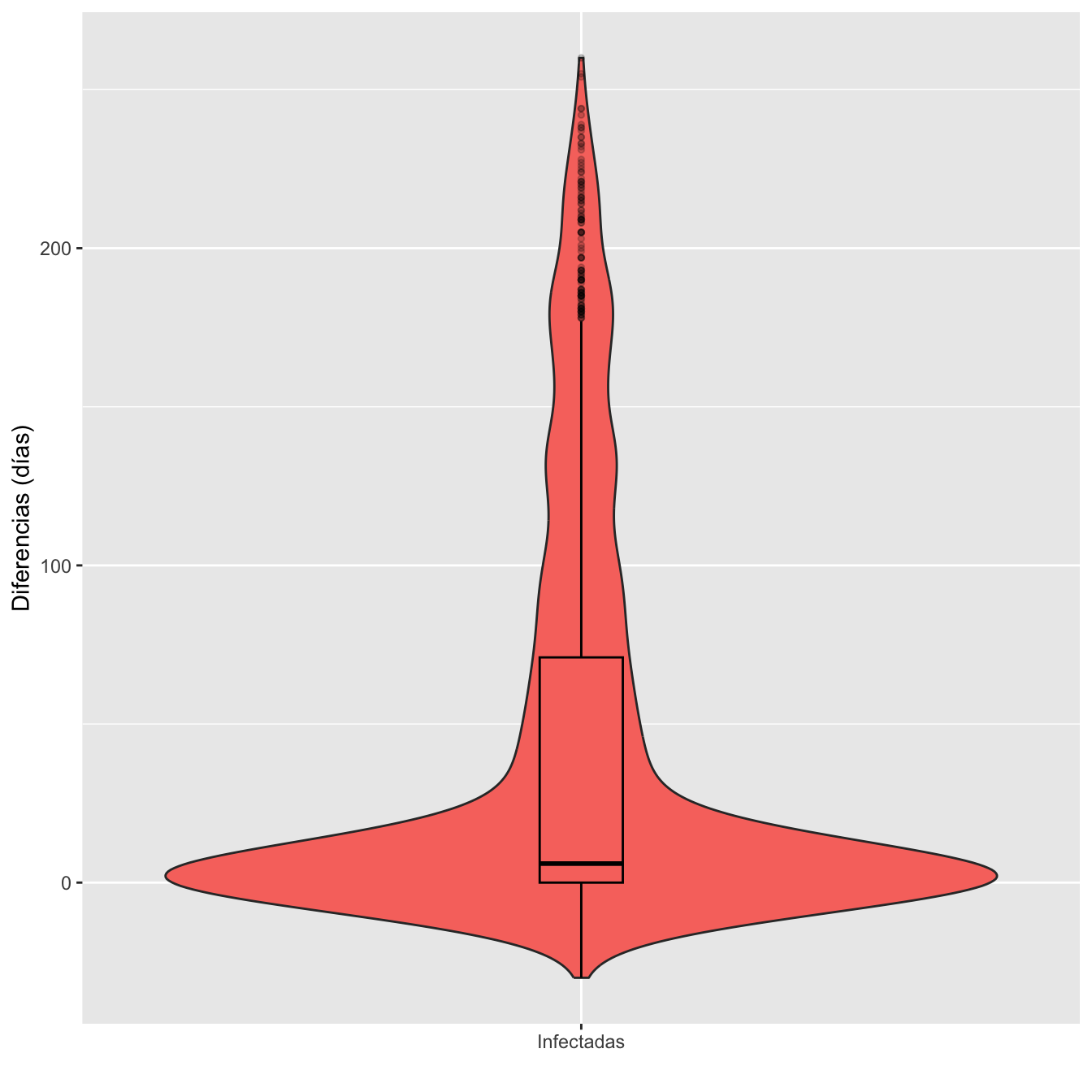
I=as.numeric(Casos$Dif)
Dades=rbind(c(min(I,na.rm=TRUE),max(I,na.rm=TRUE), round(mean(I,na.rm=TRUE),1),round(median(I,na.rm=TRUE),1),round(quantile(I,c(0.25,0.75),na.rm=TRUE),1), round(sd(I,na.rm=TRUE),1)))
colnames(Dades)=c("Mínima","Máxima","Media","Mediana", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Diferencias (días)")
Dades %>%
kbl() %>%
kable_styling()| Mínima | Máxima | Media | Mediana | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Diferencias (días) | -30 | 260 | 44 | 6 | 0 | 71 | 63.8 |
Hay:
933 casos diagnosticadas anteparto (al menos 3 días antes del parto),
735 diagnosticadas periparto (de 2 días antes del parto en adelante)
0 pérdidas (la edad gestacional de diagnóstico o de parto son desconocidas)
7.2 Comparación de antecedentes según momento de infección
7.2.1 Edades
data =data.frame(
name=c(rep("Inf. Anteparto",length(I.AP)), rep("Inf. Periparto",length(I.PP))),
Edades=c(I.AP,I.PP)
)
sample_size = data %>% group_by(name) %>% summarize(num=n())
data %>%
left_join(sample_size) %>%
mutate(myaxis = paste0(name, "\n", "n=", num)) %>%
ggplot( aes(x=myaxis, y=Edades, fill=name)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title = element_text(size=11)
) +
xlab("")+
ylab("Edades (años)")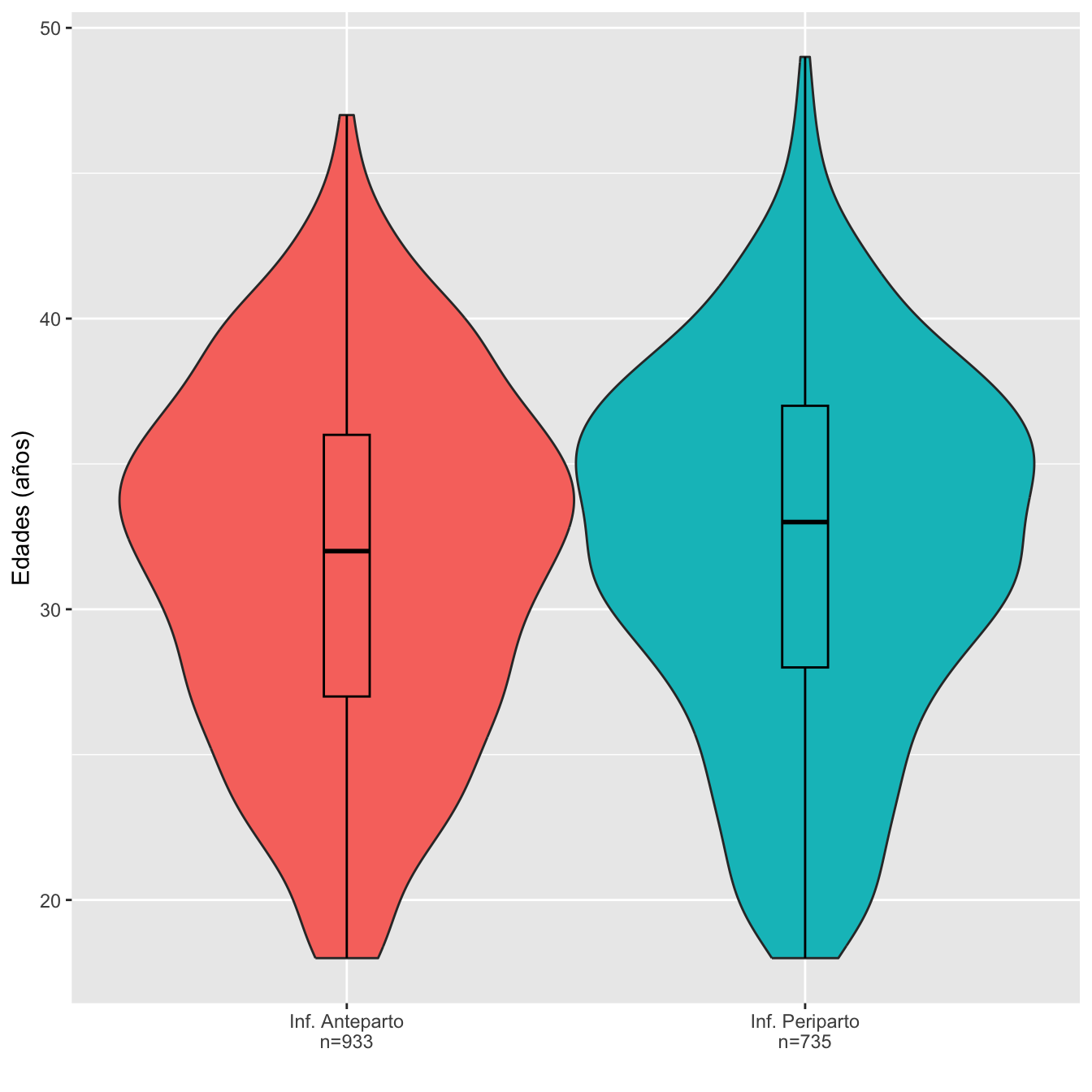
Figura 7.1:
Dades=rbind(c(min(I.AP,na.rm=TRUE),max(I.AP,na.rm=TRUE), round(mean(I.AP,na.rm=TRUE),1),round(median(I.AP,na.rm=TRUE),1),round(quantile(I.AP,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.AP,na.rm=TRUE),1)),
c(min(I.PP,na.rm=TRUE),max(I.PP,na.rm=TRUE), round(mean(I.PP,na.rm=TRUE),1),round(median(I.PP,na.rm=TRUE),1),round(quantile(I.PP,c(0.25,0.75),na.rm=TRUE),1),round(sd(I.PP,na.rm=TRUE),1)))
colnames(Dades)=c("Edad mínima","Edad máxima","Edad media", "Edad mediana", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Inf. Anteparto","Inf. Periparto")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Edad mínima | Edad máxima | Edad media | Edad mediana | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Inf. Anteparto | 18 | 47 | 31.7 | 32 | 27 | 36 | 6.1 |
| Inf. Periparto | 18 | 49 | 32.1 | 33 | 28 | 37 | 6.2 |
Ajuste de las edades de infectadas y no infectadas a distribuciones normales: test de Shapiro-Wilks, p-valores \(3\times 10^{-7}\) y \(8\times 10^{-7}\), respectivamente
Igualdad de edades medias: test t, p-valor 0.24, IC del 95% para la diferencia de medias [-0.96, 0.24] años
Igualdad de desviaciones típicas: test de Fligner-Killeen, p-valor \(0.84912\)
En las tablas como la que sigue:
- Los porcentajes se han calculado en la muestra sin pérdidas
- OR: la odds ratio univariante estimada de infectarse periparto (entre las infectadas) relativa a la franja de edad
- IC: el intervalo de confianza del 95% para la OR
- p-valor ajustado: p-valores de tests de Fisher bilaterales ajustados por Bonferroni calculados para la muestra sin pérdidas
I.cut=cut(I,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
TablaAntePeriOR.m(I.cut,c("18-30","31-40",">40"))| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| 18-30 | 376 | 40.3 | 271 | 37.1 | 1.14 | 0.93 | 1.40 | 0.564525 |
| 31-40 | 497 | 53.3 | 404 | 55.3 | 0.92 | 0.75 | 1.12 | 1.000000 |
| >40 | 60 | 6.4 | 55 | 7.5 | 0.84 | 0.57 | 1.26 | 1.000000 |
| Datos perdidos | 0 | 5 |
df =data.frame(
Factor=c(rep("Inf. Anteparto",length(I.AP)), rep("Inf. Periparto",length(I.PP))),
Edades=c( I.cut[Casos$PreP==1],I.cut[Casos$PreP==0] )
)
base=ordered(rep(c("18-30","31-40",">40"), each=2),levels=c("18-30", "31-40", ">40"))
Grupo=rep(c("Inf. Anteparto","Inf. Periparto") , length(levels(I.cut)))
valor=as.vector(prop.table(table(df), margin=1))
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=base , y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
ylab("")+
scale_fill_brewer(palette = "Accent")+
labs(fill = "Grupo de edad")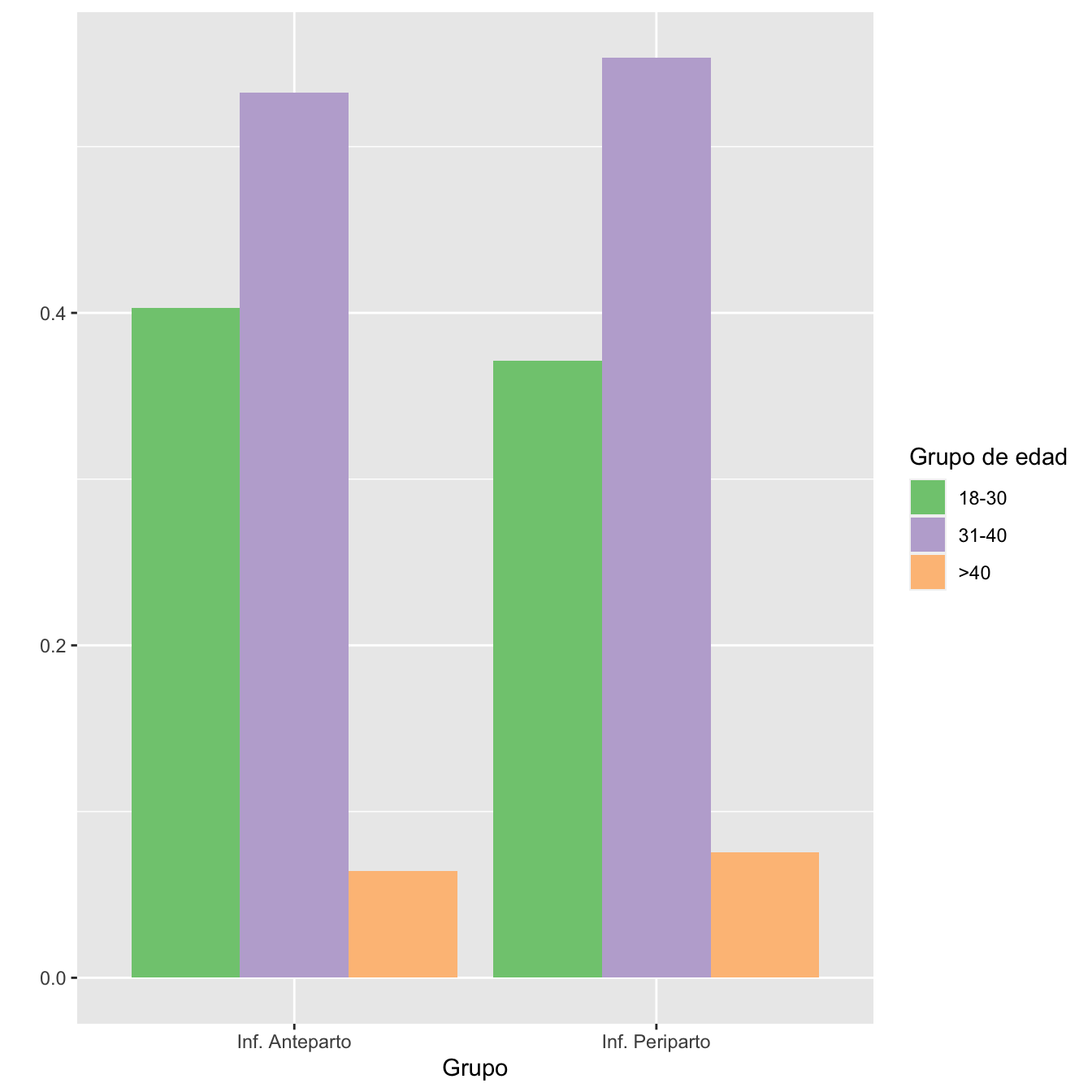
Figura 7.2:
df =data.frame(
Factor=c(rep("Inf. Anteparto",length(I.AP)), rep("Inf. Periparto",length(I.PP))),
Edades=c( I.cut[Casos$PreP==1],I.cut[Casos$PreP==0] )
)
base=ordered(rep(c("18-30","31-40",">40"), each=2),levels=c("18-30", "31-40", ">40"))
Grupo=rep(c("Inf. Anteparto","Inf. Periparto") , length(levels(I.cut)))
valor=as.vector(prop.table(table(df), margin=2))
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=Grupo, y=valor, x=base)) +
geom_bar(position="dodge", stat="identity")+
xlab("Grupos de edad")+
ylab("")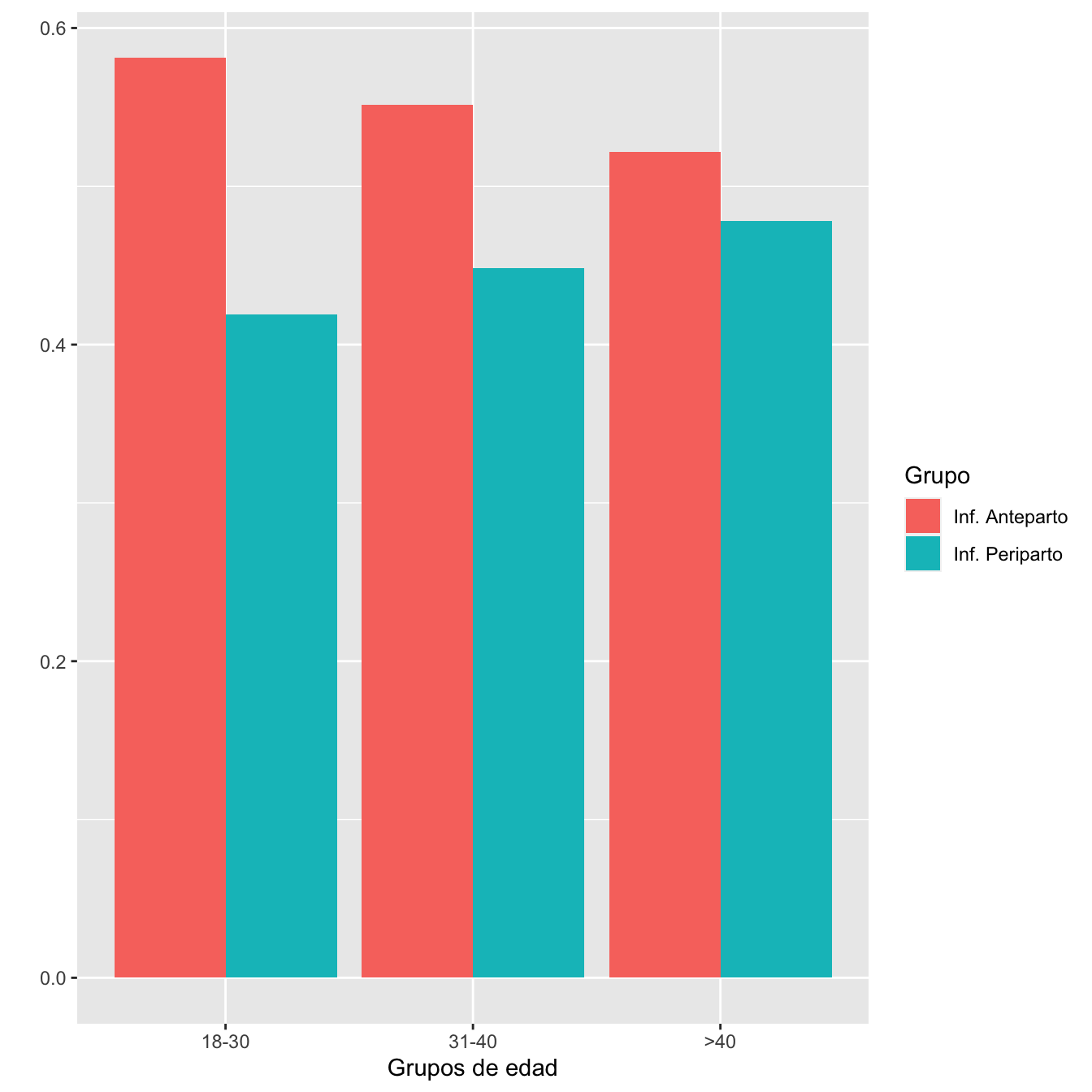
Figura 7.3:
- Igualdad de composiciones por edades de los grupos de infectadas anteparto y periparto: test \(\chi^2\), p-valor 0.3485.
7.2.2 Etnias
I=Casos$Etnia
I[I=="Asia"]="Asiática"
I=factor(I)
TablaAntePeriOR.m(I,c("Árabe", "Asiática", "Blanca", "Latinoamericana","Negra"),r=6)| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| Árabe | 70 | 7.5 | 71 | 9.7 | 0.76 | 0.53 | 1.09 | 0.658182 |
| Asiática | 26 | 2.8 | 23 | 3.1 | 0.89 | 0.48 | 1.64 | 1.000000 |
| Blanca | 470 | 50.5 | 454 | 61.9 | 0.63 | 0.51 | 0.77 | 0.000019 |
| Latinoamericana | 339 | 36.5 | 166 | 22.6 | 1.96 | 1.57 | 2.45 | 0.000000 |
| Negra | 25 | 2.7 | 19 | 2.6 | 1.04 | 0.54 | 2.01 | 1.000000 |
| Datos perdidos | 3 | 2 |
df =data.frame(
Factor=c(rep("Inf. Anteparto",length(I.AP)), rep("Inf. Periparto",length(I.PP))),
Etnias=c(I[Casos$PreP==1],I[Casos$PreP==0] )
)
base=rep(levels(I) , each=2)
Grupo=rep(c("Inf. Anteparto","Inf. Periparto") , length(levels(I)))
valor=as.vector(prop.table(table(df), margin=1))
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=base , y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
ylab("")+
scale_fill_brewer(palette = "Accent")+
labs(fill = "Etnia")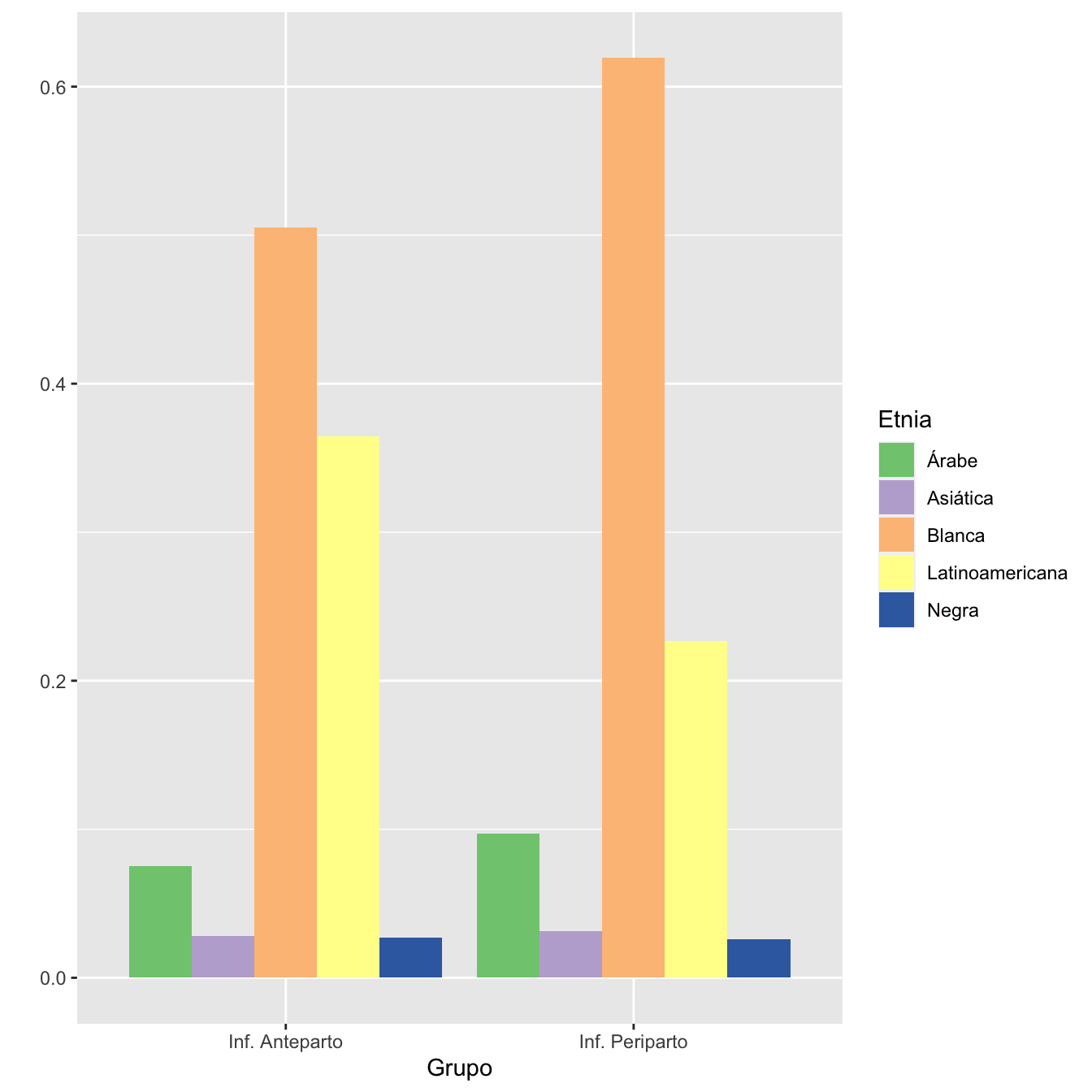
Figura 7.4:
df =data.frame(
Factor=c(rep("Inf. Anteparto",length(I.AP)), rep("Inf. Periparto",length(I.PP))),
Etnias=c(I[Casos$PreP==1],I[Casos$PreP==0] )
)
base=rep(levels(I) , each=2)
Grupo=rep(c("Inf. Anteparto","Inf. Periparto") , length(levels(I)))
valor=as.vector(prop.table(table(df), margin=2))
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=Grupo, y=valor, x=base)) +
geom_bar(position="dodge", stat="identity")+
xlab("Etnias")+
ylab("")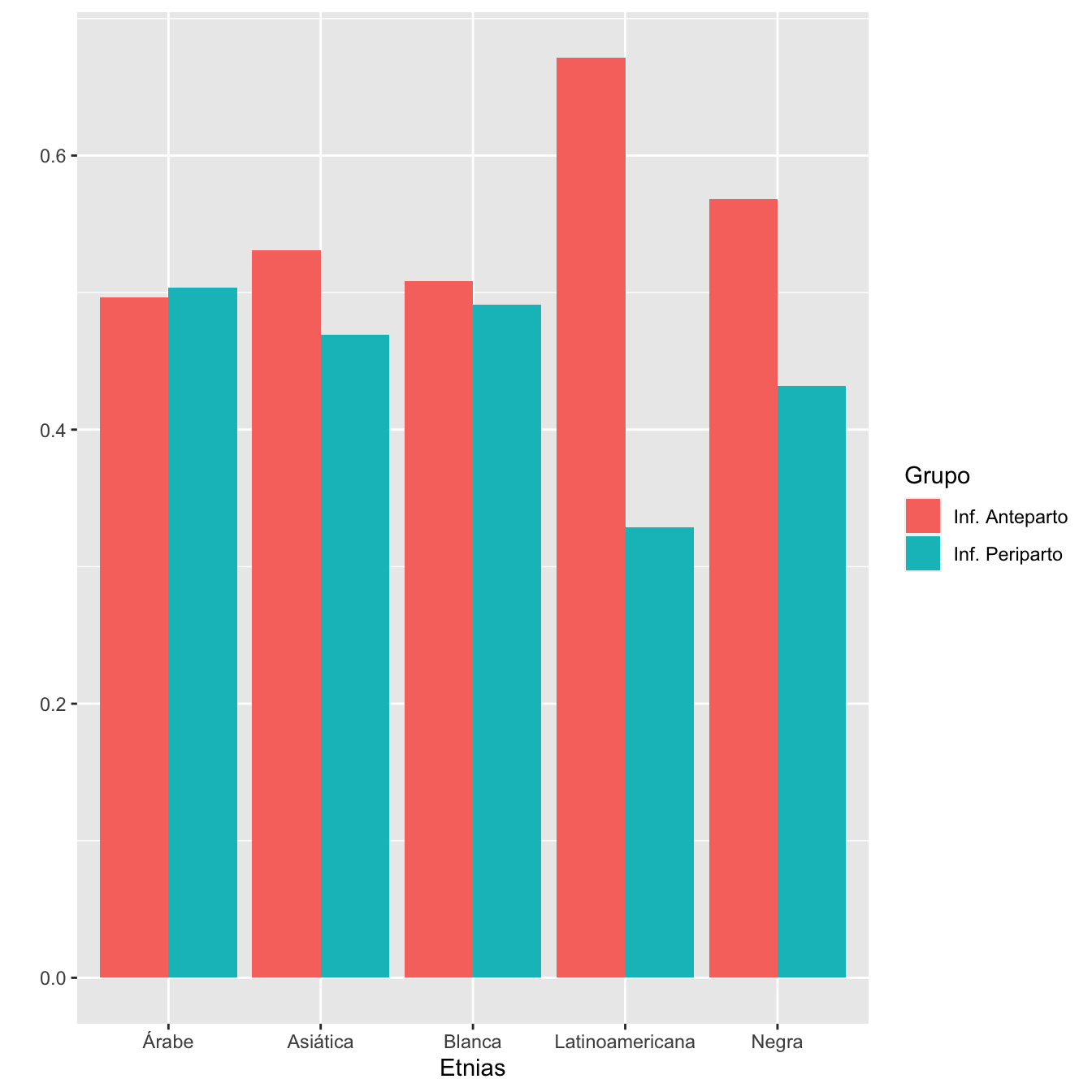
Figura 7.5:
- Igualdad de composiciones por edades de los grupos de infectadas anteparto y periparto: test \(\chi^2\), p-valor 0.
7.2.3 Hábito tabáquico
En las tablas como la que sigue (para antecedentes):
- Los porcentajes se calculan para la muestra sin pérdidas
- La OR es la de infección periparto (entre las infectadas) relativa al antecedente
- El IC es el IC 95% para la OR
- El p-valor es el del test de Fisher bilateral sin tener en cuenta los datos perdidos
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| Fumadora | 82 | 9 | 71 | 10.2 | 0.87 | 0.61 | 1.24 | 0.440591 |
| No fumadora | 829 | 91 | 625 | 89.8 | ||||
| Datos perdidos | 22 | 39 |
7.2.4 Obesidad
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| Obesa | 179 | 19.2 | 126 | 17.1 | 1.15 | 0.89 | 1.49 | 0.307449 |
| No obesa | 754 | 80.8 | 609 | 82.9 | ||||
| Datos perdidos | 0 | 0 |
7.2.5 Hipertensión pregestacional
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| HTA | 19 | 2 | 6 | 0.8 | 2.52 | 0.96 | 7.76 | 0.043945 |
| No HTA | 914 | 98 | 729 | 99.2 | ||||
| Datos perdidos | 0 | 0 |
7.2.6 Diabetes Mellitus
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| DM | 25 | 2.7 | 10 | 1.4 | 2 | 0.92 | 4.69 | 0.083988 |
| No DM | 908 | 97.3 | 725 | 98.6 | ||||
| Datos perdidos | 0 | 0 |
7.2.7 Enfermedades cardíacas crónicas
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| ECC | 12 | 1.3 | 7 | 1 | 1.35 | 0.49 | 4.08 | 0.644426 |
| No ECC | 921 | 98.7 | 728 | 99 | ||||
| Datos perdidos | 0 | 0 |
7.2.8 Enfermedades pulmonares crónicas (incluyendo asma)
INA=Casos$Enfermedad.pulmonar.crónica.no.asma
IA=Casos$Diagnóstico.clínico.de.Asma
I=rep(NA,n_I)
for (i in 1:n_I){I[i]=max(INA[i],IA[i],na.rm=TRUE)}
TablaAntePeriOR(I,"No EPC","EPC")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| EPC | 52 | 5.6 | 22 | 3 | 1.91 | 1.13 | 3.34 | 0.011689 |
| No EPC | 881 | 94.4 | 713 | 97 | ||||
| Datos perdidos | 0 | 0 |
7.2.9 Paridad
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | OR | Extr. Inf. IC | Extr. Sup. IC | p-valor | |
|---|---|---|---|---|---|---|---|---|
| Nulípara | 308 | 33.2 | 298 | 41.1 | 0.71 | 0.58 | 0.88 | 0.001002 |
| Multípara | 619 | 66.8 | 427 | 58.9 | ||||
| Datos perdidos | 6 | 10 |
7.3 Comparación de desenlaces según momento de infección
7.3.1 Preeclampsia
En las tablas como la que sigue:
RA: la proporción de desenlaces entre las infectadas anteparto menos la proporción de desenlaces entre las infectadas periparto
RR: la proporción de desenlaces entre las infectadas anteparto dividida por la proporción de desenlaces entre las infectadas periparto
p-valor: p-valor del test de Fisher de igualdad de proporciones de desenlaces entre las infectadas anteparto y periparto
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| PE | 49 | 5.3 | 37 | 5 | 0.0022 | -0.0204 | 0.0247 | 1.04 | 0.69 | 1.58 | 0.911337 |
| No PE | 884 | 94.7 | 698 | 95 | |||||||
| Datos perdidos | 0 | 0 |
7.3.2 Preeclampsia con CG
I.AP=Casos$Preeclampsia.grave_HELLP_ECLAMPSIA[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1 & Casos$PreP==1]
I.PP=Casos$Preeclampsia.grave_HELLP_ECLAMPSIA[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1 & Casos$PreP==0]
TablaAntePeri.r(I.AP, I.PP, "PE con CG", "PE sin CG")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| PE con CG | 15 | 30.6 | 15 | 40.5 | -0.0993 | -0.3272 | 0.1286 | 0.76 | 0.43 | 1.34 | 0.368505 |
| PE sin CG | 34 | 69.4 | 22 | 59.5 | |||||||
| Datos perdidos | 0 | 0 |
7.3.3 Anomalías congénitas
I=Casos$Diagnóstico.de.malformación.ecográfica._.semana.20._
TablaAntePeri(I,"Anomalías congénitas", "No anomalías congénitas")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Anomalías congénitas | 16 | 1.8 | 12 | 1.7 | 8e-04 | -0.0128 | 0.0144 | 1.05 | 0.51 | 2.17 | 1 |
| No anomalías congénitas | 887 | 98.2 | 698 | 98.3 | |||||||
| Datos perdidos | 30 | 25 |
7.3.4 Retraso del crecimiento intrauterino
I=Casos$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.
TablaAntePeri(I,"RCIU","No RCIU")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| RCIU | 39 | 4.2 | 20 | 2.7 | 0.0146 | -0.004 | 0.0332 | 1.54 | 0.91 | 2.6 | 0.141336 |
| No RCIU | 894 | 95.8 | 715 | 97.3 | |||||||
| Datos perdidos | 0 | 0 |
7.3.5 Diabetes gestacional
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| DG | 73 | 7.8 | 52 | 7.1 | 0.0075 | -0.019 | 0.034 | 1.11 | 0.79 | 1.56 | 0.575736 |
| No DG | 860 | 92.2 | 683 | 92.9 | |||||||
| Datos perdidos | 0 | 0 |
7.3.6 Hipertensión gestacional
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| HG | 22 | 2.4 | 20 | 2.7 | -0.0036 | -0.0201 | 0.0129 | 0.87 | 0.48 | 1.56 | 0.640659 |
| No HG | 911 | 97.6 | 715 | 97.3 | |||||||
| Datos perdidos | 0 | 0 |
7.3.7 Rotura prematura de membranas
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| RPM | 120 | 12.9 | 115 | 15.6 | -0.0278 | -0.063 | 0.0073 | 0.82 | 0.65 | 1.04 | 0.118773 |
| No RPM | 813 | 87.1 | 620 | 84.4 | |||||||
| Datos perdidos | 0 | 0 |
7.3.8 Ingreso materno en UCI
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| UCI | 19 | 2 | 18 | 2.4 | -0.0041 | -0.0197 | 0.0115 | 0.83 | 0.44 | 1.56 | 0.617234 |
| No UCI | 914 | 98 | 717 | 97.6 | |||||||
| Datos perdidos | 0 | 0 |
7.3.9 Ingreso materno en UCI según el momento del parto o cesárea
I.AP=Casos$UCI_ANTES.DESPUES.DEL.PARTO[Casos$UCI==1 & Casos$PreP==1]
I.PP=Casos$UCI_ANTES.DESPUES.DEL.PARTO[Casos$UCI==1 & Casos$PreP==0]
TablaAntePeri.r(I.AP, I.PP,"Antes del parto", "Después del parto", )| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Antes del parto | 11 | 57.9 | 16 | 88.9 | -0.3099 | -0.6293 | 0.0094 | 0.65 | 0.4 | 0.96 | 0.062496 |
| Después del parto | 8 | 42.1 | 2 | 11.1 | |||||||
| Datos perdidos | 0 | 0 |
7.3.10 Feto muerto anteparto
I1.AP=Casos$Feto.muerto.intraútero[Casos$PreP==1]
I1.AP[I1.AP=="Sí"]=1
I1.AP[I1.AP=="No"]=0
I1.PP=Casos$Feto.muerto.intraútero[Casos$PreP==0]
I1.PP[I1.PP=="Sí"]=1
I1.PP[I1.PP=="No"]=0
I2.AP=CasosGM$Feto.vivo[CasosGM$PreP==1]
I2.AP[I2.AP=="Sí"]=0
I2.AP[I2.AP=="No"]=1
I2.PP=CasosGM$Feto.vivo[CasosGM$PreP==0]
I2.PP[I2.PP=="Sí"]=0
I2.PP[I2.PP=="No"]=1
I.AP=c(I1.AP,I2.AP)
I.PP=c(I1.PP,I2.PP)
TablaAntePeri.r(I.AP, I.PP,"Ningún feto muerto anteparto","Algún feto muerto anteparto")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Ningún feto muerto anteparto | 6 | 0.6 | 13 | 1.7 | -0.0112 | -0.023 | 7e-04 | 0.36 | 0.14 | 0.91 | 0.036298 |
| Algún feto muerto anteparto | 947 | 99.4 | 732 | 98.3 | |||||||
| Datos perdidos | 1 | 0 |
7.3.11 Prematurez
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Prematuro | 89 | 9.5 | 80 | 10.9 | -0.0135 | -0.044 | 0.0171 | 0.88 | 0.66 | 1.17 | 0.369921 |
| A término | 844 | 90.5 | 655 | 89.1 | |||||||
| Datos perdidos | 0 | 0 |
7.3.12 Eventos trombóticos
| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos trombóticos | 4 | 0.4 | 8 | 1.1 | -0.0066 | -0.0164 | 0.0032 | 0.39 | 0.13 | 1.22 | 0.146395 |
| No eventos trombóticos | 929 | 99.6 | 727 | 98.9 | |||||||
| Datos perdidos | 0 | 0 |
7.3.13 Eventos hemorrágicos
I=Casos$EVENTOS_HEMORRAGICOS_TOTAL
TablaAntePeri(I,"Eventos hemorrágicos","No eventos hemorrágicos")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos hemorrágicos | 45 | 4.8 | 46 | 6.3 | -0.0144 | -0.0378 | 0.0091 | 0.77 | 0.52 | 1.15 | 0.232208 |
| No eventos hemorrágicos | 888 | 95.2 | 689 | 93.7 | |||||||
| Datos perdidos | 0 | 0 |
7.3.14 Hemorragias potsparto
I=Casos$Hemorragia.postparto
I.sino=I
I.sino[I.sino!="No" & !is.na(I.sino)]="Sí"
TablaAntePeri(I.sino,"HPP","No HPP")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| HPP | 35 | 3.8 | 38 | 5.3 | -0.0152 | -0.037 | 0.0067 | 0.71 | 0.46 | 1.12 | 0.148691 |
| No HPP | 885 | 96.2 | 676 | 94.7 | |||||||
| Datos perdidos | 13 | 21 |
I=ordered(I,levels=names(table(I))[c(2,3,1,4)])
TablaAntePeri.m(I,L=c("Tto. médico",
"Tto. Qx. conservador","Histerectomía",
"No HPP"))| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Tto. médico | 30 | 3.3 | 29 | 4.1 | -0.0080 | -0.0277 | 0.0117 | 0.803 | 0.489 | 1.319 | 1.000000 |
| Tto. Qx. conservador | 4 | 0.4 | 5 | 0.7 | -0.0027 | -0.0113 | 0.0060 | 0.621 | 0.179 | 2.150 | 1.000000 |
| Histerectomía | 1 | 0.1 | 4 | 0.6 | -0.0045 | -0.0116 | 0.0026 | 0.194 | 0.031 | 1.228 | 0.698069 |
| No HPP | 885 | 96.2 | 676 | 94.7 | 0.0152 | -0.0067 | 0.0370 | 1.016 | 0.994 | 1.038 | 0.594763 |
| Datos perdidos | 13 | 21 |
- Diferencia entre infectadas ante y periparto: test \(\chi^2\) de Montecarlo, p-valor NaN
7.3.15 Inicio del parto
I=Casos$Inicio.de.parto
I=factor(Casos$Inicio.de.parto,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
TablaAntePeri.m(I,c("Espontáneo", "Inducido", "Cesárea"))| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Espontáneo | 467 | 50.1 | 404 | 55.1 | -0.0506 | -0.1001 | -0.0012 | 0.908 | 0.829 | 0.995 | 0.128420 |
| Inducido | 365 | 39.1 | 263 | 35.9 | 0.0324 | -0.0156 | 0.0804 | 1.090 | 0.962 | 1.236 | 0.556730 |
| Cesárea | 101 | 10.8 | 66 | 9.0 | 0.0182 | -0.0118 | 0.0482 | 1.202 | 0.896 | 1.613 | 0.749737 |
| Datos perdidos | 0 | 2 |
I.AP=Casos$Inicio.de.parto[Casos$PreP==1]
I.PP=Casos$Inicio.de.parto[Casos$PreP==0]
NI=Controls$Inicio.de.parto
NI[NI=="Cesárea programada"]="Cesárea"
df =data.frame(
Factor=c( rep("Inf. anteparto",length(I.AP)), rep("Inf. periparto",length(I.PP)), rep("No infectadas",length(NI))),
Inicios=ordered(c( I.AP,I.PP,NI ),levels=c("Espontáneo","Inducido","Cesárea"))
)
base=ordered(rep(c("Espontáneo","Inducido","Cesárea"), each=3),levels=c("Espontáneo","Inducido","Cesárea"))
Grupo=rep(c("Inf. anteparto","Inf. periparto","No infectada") , 3)
valor=c(as.vector(prop.table(table(df),margin=1)) )
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=base, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("")+
scale_fill_brewer(palette = "Set3") +
labs(fill = "Inicio")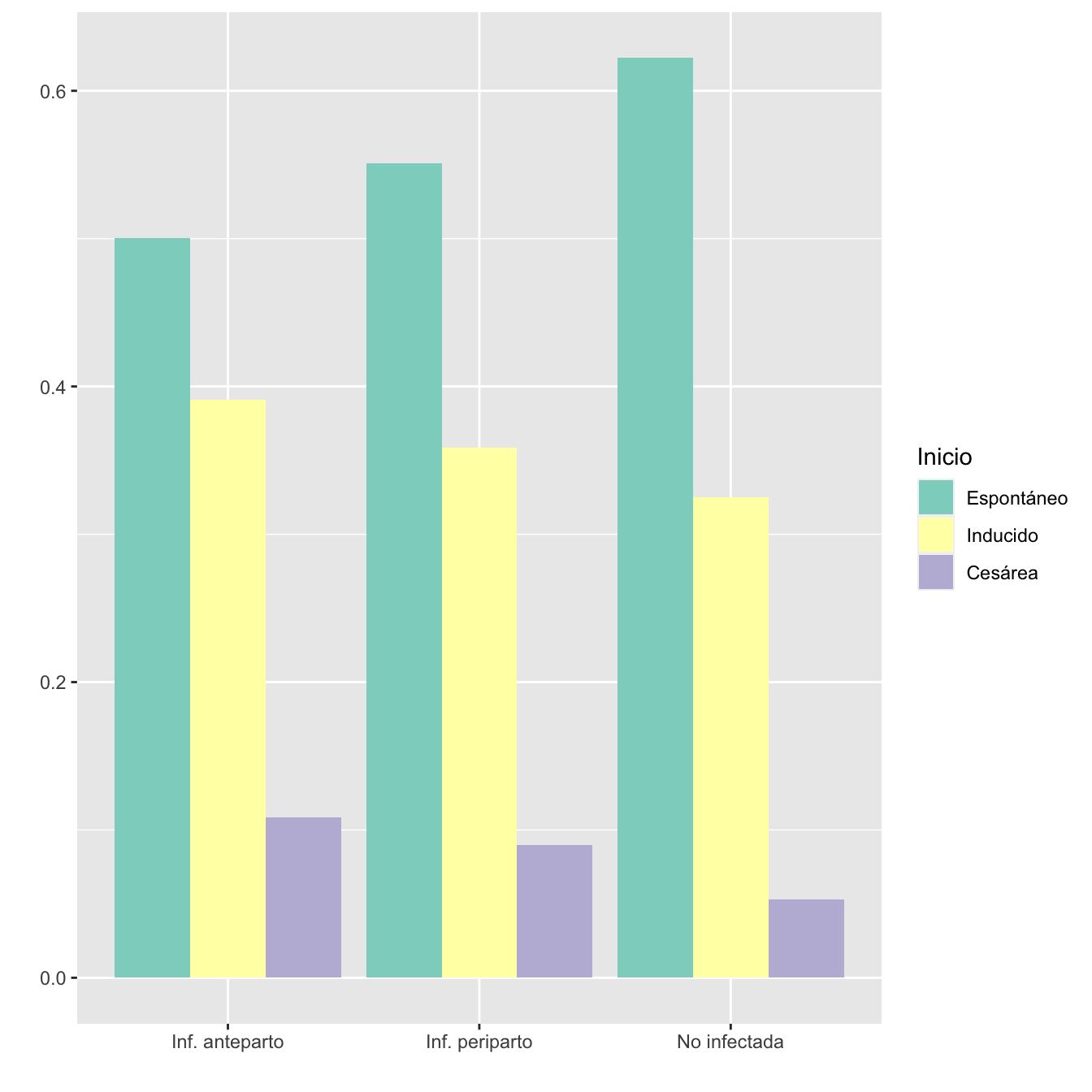
- Diferencia entre infectadas ante y periparto: test \(\chi^2\), p-valor NaN
7.3.16 Tipo de parto
I.In=Casos$Inicio.de.parto
I.In[I.In=="Cesárea"]="Cesárea programada"
I=Casos$Tipo.de.parto
I[I.In=="Cesárea programada"]="Cesárea programada"
I[I=="Cesárea"]="Cesárea urgente"
I=factor(I,levels=c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
TablaAntePeri.m(I,c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"))| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Eutócico | 606 | 65.0 | 449 | 61.1 | 0.0386 | -0.0093 | 0.0865 | 1.063 | 0.987 | 1.145 | 0.451299 |
| Instrumental | 93 | 10.0 | 84 | 11.4 | -0.0146 | -0.0458 | 0.0166 | 0.872 | 0.661 | 1.152 | 1.000000 |
| Cesárea programada | 101 | 10.8 | 66 | 9.0 | 0.0185 | -0.0115 | 0.0484 | 1.206 | 0.899 | 1.617 | 0.875604 |
| Cesárea urgente | 133 | 14.3 | 136 | 18.5 | -0.0425 | -0.0796 | -0.0053 | 0.770 | 0.619 | 0.958 | 0.090011 |
| Datos perdidos | 0 | 0 |
I.AP.In=Casos$Inicio.de.parto[Casos$PreP==1]
I.AP.In[I.AP.In=="Cesárea"]="Cesárea programada"
I.AP=Casos$Tipo.de.parto[Casos$PreP==1]
I.AP[I.AP.In=="Cesárea programada"]="Cesárea programada"
I.AP[I.AP=="Cesárea"]="Cesárea urgente"
I.PP.In=Casos$Inicio.de.parto[Casos$PreP==0]
I.PP.In[I.PP.In=="Cesárea"]="Cesárea programada"
I.PP=Casos$Tipo.de.parto[Casos$PreP==0]
I.PP[I.PP.In=="Cesárea programada"]="Cesárea programada"
I.PP[I.PP=="Cesárea"]="Cesárea urgente"
NI.In=Controls$Inicio.de.parto
NI=Controls$Tipo.de.parto
NI[NI.In=="Cesárea programada"]="Cesárea programada"
NI[NI=="Cesárea"]="Cesárea urgente"
df =data.frame(
Factor=c( rep("Inf. anteparto",length(I.AP)), rep("Inf. periparto",length(I.PP)), rep("No infectadas",length(NI))),
Inicios=ordered(c( I.AP,I.PP,NI ),levels=c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"))
)
base=ordered(rep(c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"), each=3),levels=c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"))
Grupo=rep(c("Inf. anteparto","Inf. periparto","No infectada") , 4)
valor=c(as.vector(prop.table(table(df),margin=1)) )
data <- data.frame(base,Grupo,valor)
ggplot(data, aes(fill=base, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("")+
scale_fill_brewer(palette = "Set3") +
labs(fill = "Tipo")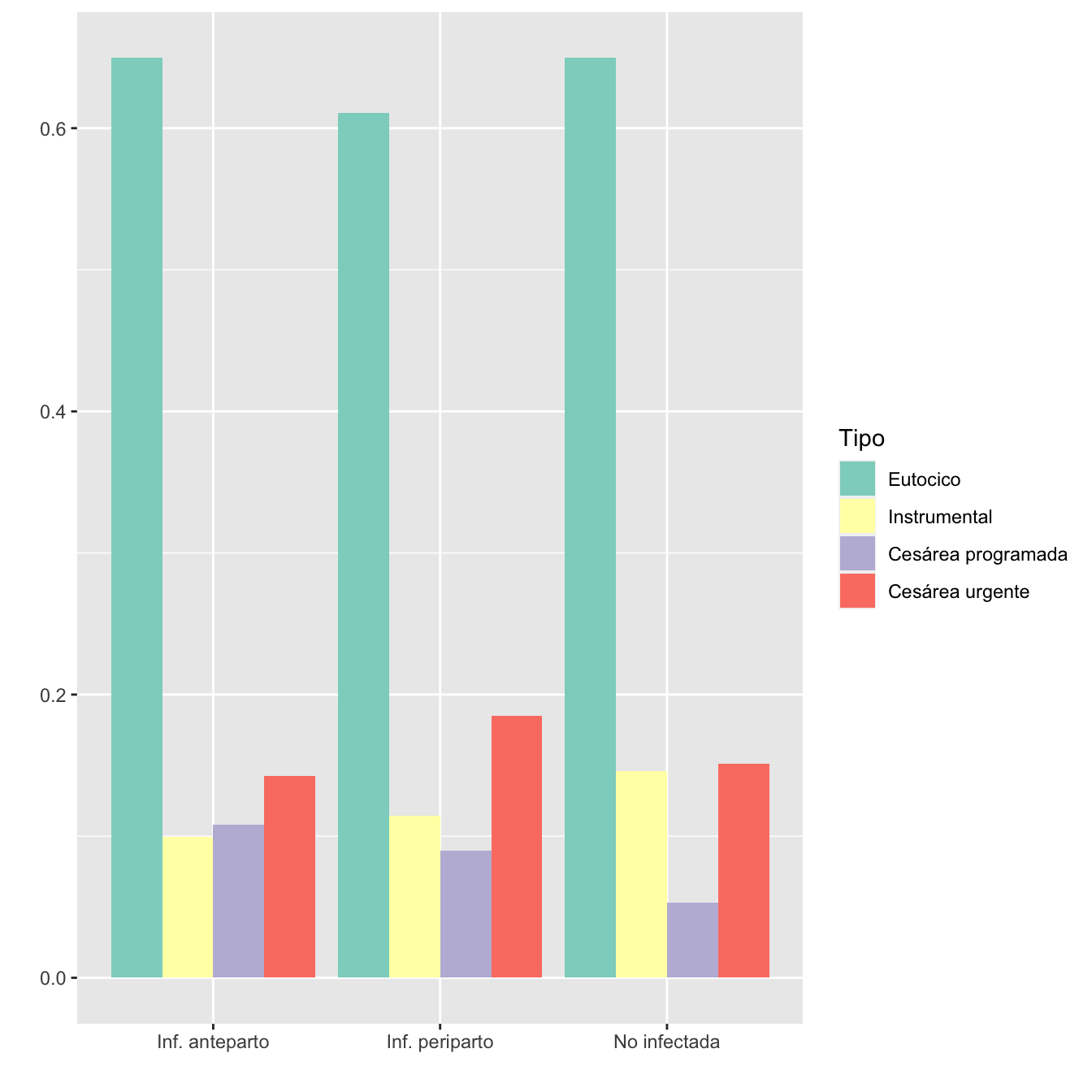
- Diferencia entre infectadas ante y periparto: test \(\chi^2\), p-valor NaN
7.3.17 Test de Apgar
I.AP=c(Casos[Casos$PreP==1,]$APGAR.5...126,CasosGM[CasosGM$PreP==1,]$APGAR.5...150)
I.PP=c(Casos[Casos$PreP==0,]$APGAR.5...126,CasosGM[CasosGM$PreP==0,]$APGAR.5...150)
I.AP=cut(I.AP,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I.PP=cut(I.PP,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I.AP=ordered(I.AP,levels=c("8-10","0-7"))
I.PP=ordered(I.PP,levels=c("8-10","0-7"))
TablaAntePeri.r(I.AP,I.PP,"Apgar.5≤7", "Apgar.5≥8")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Apgar.5≤7 | 20 | 2.1 | 30 | 4.1 | -0.0195 | -0.0376 | -0.0013 | 0.52 | 0.3 | 0.91 | 0.029026 |
| Apgar.5≥8 | 921 | 97.9 | 707 | 95.9 | |||||||
| Datos perdidos | 13 | 8 |
7.3.18 Ingreso del neonato en UCI
I.AP=c(Casos[Casos$Feto.muerto.intraútero=="No" & Casos$PreP==1,]$Ingreso.en.UCIN,CasosGM[CasosGM$Feto.vivo=="Sí" & CasosGM$PreP==1,]$Ingreso.en.UCI)
I.PP=c(Casos[Casos$Feto.muerto.intraútero=="No" & Casos$PreP==0,]$Ingreso.en.UCIN,CasosGM[CasosGM$Feto.vivo=="Sí" & CasosGM$PreP==0,]$Ingreso.en.UCI)
TablaAntePeri.r(I.AP,I.PP, "UCIN", "No UCIN")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| UCIN | 71 | 7.6 | 86 | 11.9 | -0.043 | -0.0733 | -0.0127 | 0.64 | 0.47 | 0.86 | 0.003916 |
| No UCIN | 864 | 92.4 | 637 | 88.1 | |||||||
| Datos perdidos | 13 | 9 |
7.3.19 Peso de neonatos
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí",]$Peso._gramos_...247)
I.AP=I[Casos$PreP==1]
I.PP=I[Casos$PreP==0]Dades=rbind(c(round(min(I.AP,na.rm=TRUE),1),round(max(I.AP,na.rm=TRUE),1), round(mean(I.AP,na.rm=TRUE),1),round(median(I.AP,na.rm=TRUE),1),round(quantile(I.AP,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.AP,na.rm=TRUE),1)),
c(round(min(I.PP,na.rm=TRUE),1),round(max(I.PP,na.rm=TRUE),1), round(mean(I.PP,na.rm=TRUE),1),round(median(I.PP,na.rm=TRUE),1),round(quantile(I.PP,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.PP,na.rm=TRUE),1)))
colnames(Dades)=c("Peso mínimo","Peso máximo","Peso medio", "Peso mediano", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Infectadas anteparto", "Infectadas periparto")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Peso mínimo | Peso máximo | Peso medio | Peso mediano | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Infectadas anteparto | 315 | 4750 | 3210.4 | 3275 | 2920 | 3576.2 | 589.0 |
| Infectadas periparto | 680 | 5190 | 3206.4 | 3250 | 2910 | 3560.0 | 590.9 |
data =data.frame(
name=c( rep("Anteparto",length(I.AP)), rep("Periparto",length(I.PP))),
Pesos=c( I.AP,I.PP))
data %>%
ggplot( aes(x=name, y=Pesos, fill=name)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title=element_text(size=11)
) +
xlab("")+
ylab("Pesos (gramos)")+
scale_fill_brewer(palette = "Dark2") 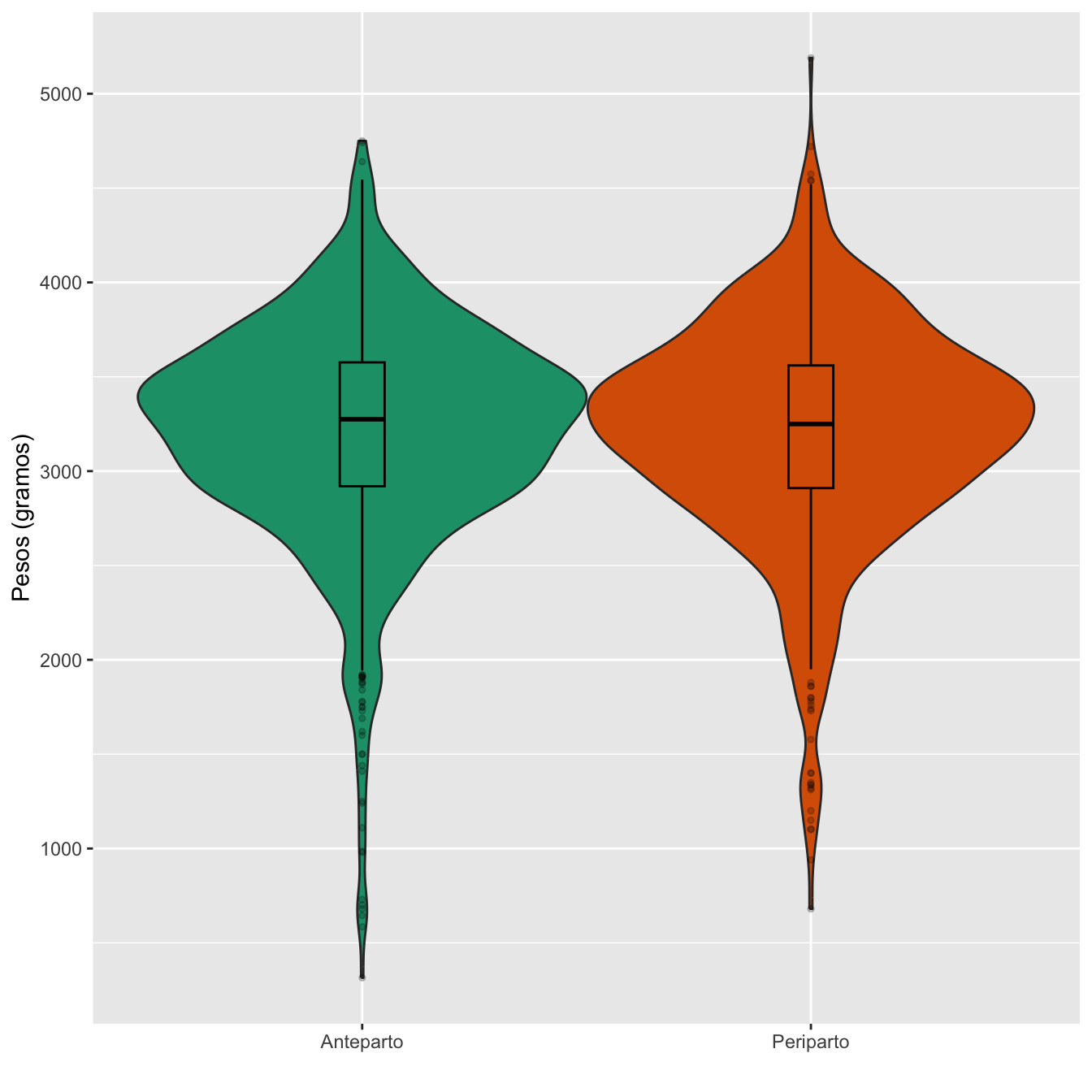
Figura 7.6:
- p-valor del test t para los pesos medios de infectadas ante y periparto: p-valor 0.89441
Bajo peso
I.cut=cut(I,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
I.cut=ordered(I.cut,levels=c(0,1))
TablaAntePeri(I.cut,"Bajo peso","No bajo peso")| Inf. Anteparto (N) | Inf. Anteparto (%) | Inf. Periparto (N) | Inf. Periparto (%) | RA | Extr. Inf. IC RA | Extr. Sup. IC RA | RR | Extr. Inf. IC RA | Extr. Sup. IC RR | p-valor | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Bajo peso | 85 | 9.3 | 67 | 9.6 | -0.0032 | -0.0333 | 0.0269 | 0.97 | 0.71 | 1.31 | 0.863541 |
| No bajo peso | 831 | 90.7 | 631 | 90.4 | |||||||
| Datos perdidos | 17 | 37 |
7.4 Comparación de desenlaces según momento de infección y sintomatología
Porcentajes calculados por filas
I.AP=Casos$SINTOMAS_DIAGNOSTICO[Casos$PreP==1]
I.PP=Casos$SINTOMAS_DIAGNOSTICO[Casos$PreP==0]
NA_IAP=length(I.AP[is.na(I.AP)])
NA_IPP=length(I.PP[is.na(I.PP)])
EEExt=rbind(c(table(I.AP)[1],round(100*prop.table(table(I.AP)),2)[1],
table(I.AP)[2],round(100*prop.table(table(I.AP)),2)[2],
table(I.AP)[3],round(100*prop.table(table(I.AP)),2)[3],
length(I.AP)),
c(table(I.PP)[1],round(100*prop.table(table(I.PP)),2)[1],
table(I.PP)[2],round(100*prop.table(table(I.PP)),2)[2],
table(I.PP)[3],round(100*prop.table(table(I.PP)),2)[3],
length(I.PP))
)
EEE=EEExt[,c(1,3,5)]
colnames(EEE)=c("Asintomáticas", "Síntomas leves", "Neumonía")
colnames(EEExt)= c("Asintomáticas (N)", "Asintomáticas (%)","Síntomas leves (N)", "Síntomas leves (%)", "Neumonía (N)", "Neumonía (%)","Total")
rownames(EEExt)=c("Infectadas anteparto", "Infectadas periparto")
EEExt %>%
kbl() %>%
kable_styling()| Asintomáticas (N) | Asintomáticas (%) | Síntomas leves (N) | Síntomas leves (%) | Neumonía (N) | Neumonía (%) | Total | |
|---|---|---|---|---|---|---|---|
| Infectadas anteparto | 223 | 23.90 | 505 | 54.13 | 205 | 21.97 | 933 |
| Infectadas periparto | 522 | 71.02 | 138 | 18.78 | 75 | 10.20 | 735 |
Porcentajes calculados por columnas
I.AP=Casos$SINTOMAS_DIAGNOSTICO[Casos$PreP==1]
I.PP=Casos$SINTOMAS_DIAGNOSTICO[Casos$PreP==0]
TT=table(Casos$PreP,Casos$SINTOMAS_DIAGNOSTICO)
p.TT=round(100*prop.table(TT,margin=2)[2:1,],1)
NA_IAP=length(I.AP[is.na(I.AP)])
NA_IPP=length(I.PP[is.na(I.PP)])
#table(I.AP)
#table(I.PP)
EEExt=rbind(c(TT[2,1],p.TT[1,1],
TT[2,2],p.TT[1,2],
TT[2,3],p.TT[1,3],
length(I.AP)),
c(TT[1,1],p.TT[2,1],
TT[1,2],p.TT[2,2],
TT[1,3],p.TT[2,3],
length(I.PP))
)
EEE=EEExt[,c(1,3,5)]
colnames(EEE)=c("Asintomáticas", "Síntomas leves", "Neumonía")
colnames(EEExt)= c("Asintomáticas (N)", "Asintomáticas (%)","Síntomas leves (N)", "Síntomas leves (%)", "Neumonía (N)", "Neumonía (%)","Total")
rownames(EEExt)=c("Infectadas anteparto", "Infectadas periparto")
EEExt %>%
kbl() %>%
kable_styling()| Asintomáticas (N) | Asintomáticas (%) | Síntomas leves (N) | Síntomas leves (%) | Neumonía (N) | Neumonía (%) | Total | |
|---|---|---|---|---|---|---|---|
| Infectadas anteparto | 223 | 29.9 | 505 | 78.5 | 205 | 73.2 | 933 |
| Infectadas periparto | 522 | 70.1 | 138 | 21.5 | 75 | 26.8 | 735 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=2),levels=c("Asintomática", "Leve", "Grave"))
Grupo=rep(c("Anteparto","Periparto") , 3)
valor1=as.vector(prop.table(EEE, margin=1))
data1 <- data.frame(Grupo,Síntomas,valor1)
valor2=as.vector(prop.table(EEE, margin=2))
data2 <- data.frame(Grupo,Síntomas,valor2)
ggplot(data1, aes(fill=Síntomas, y=valor1, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("Momento de diagnóstico")+
ylab("")+
scale_fill_brewer(palette = "Dark2") 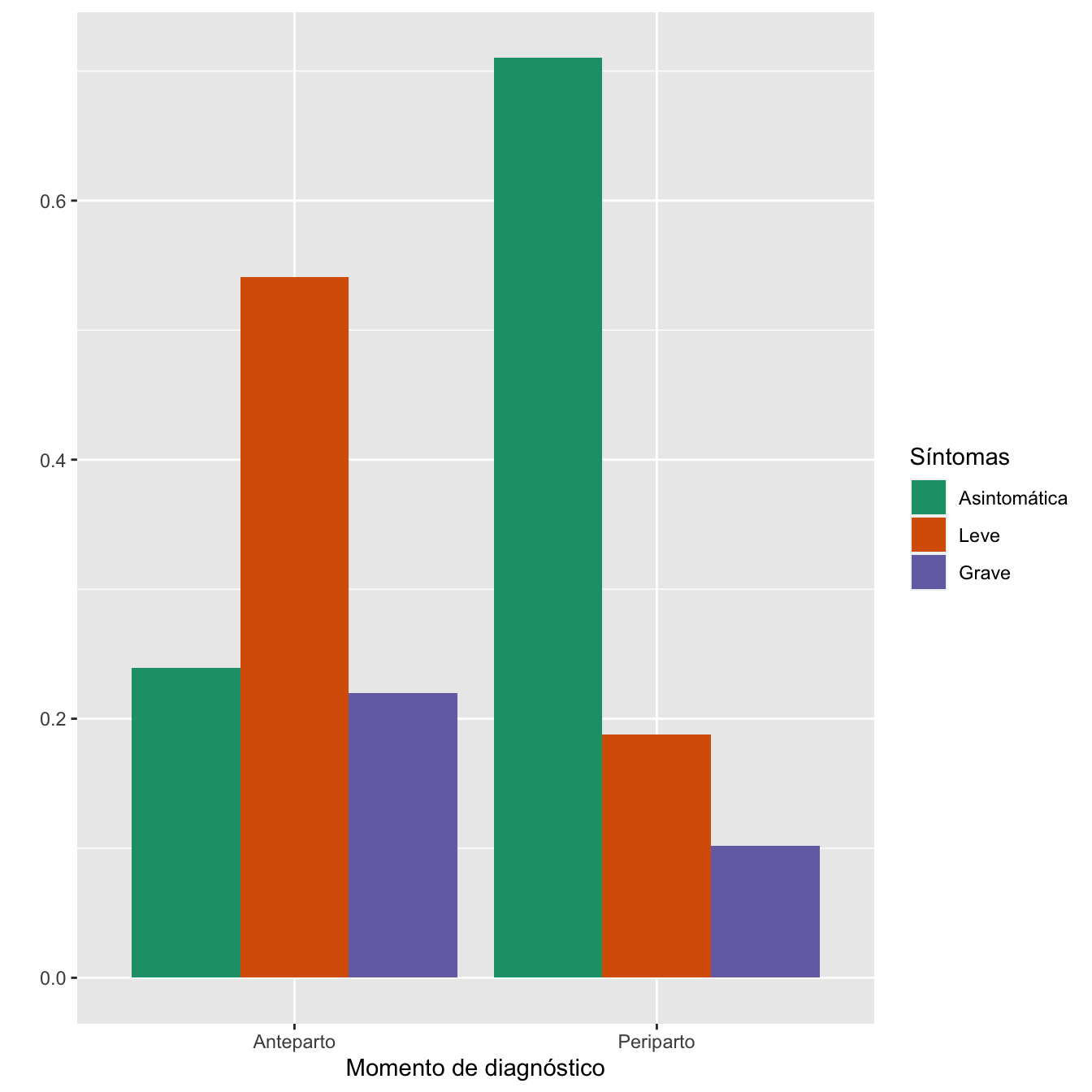
ggplot(data2, aes(fill=Grupo, y=valor2, x=Síntomas)) +
geom_bar(position="dodge", stat="identity")+
xlab("Síntomas")+
ylab("")+
labs(fill = "Momento diag.")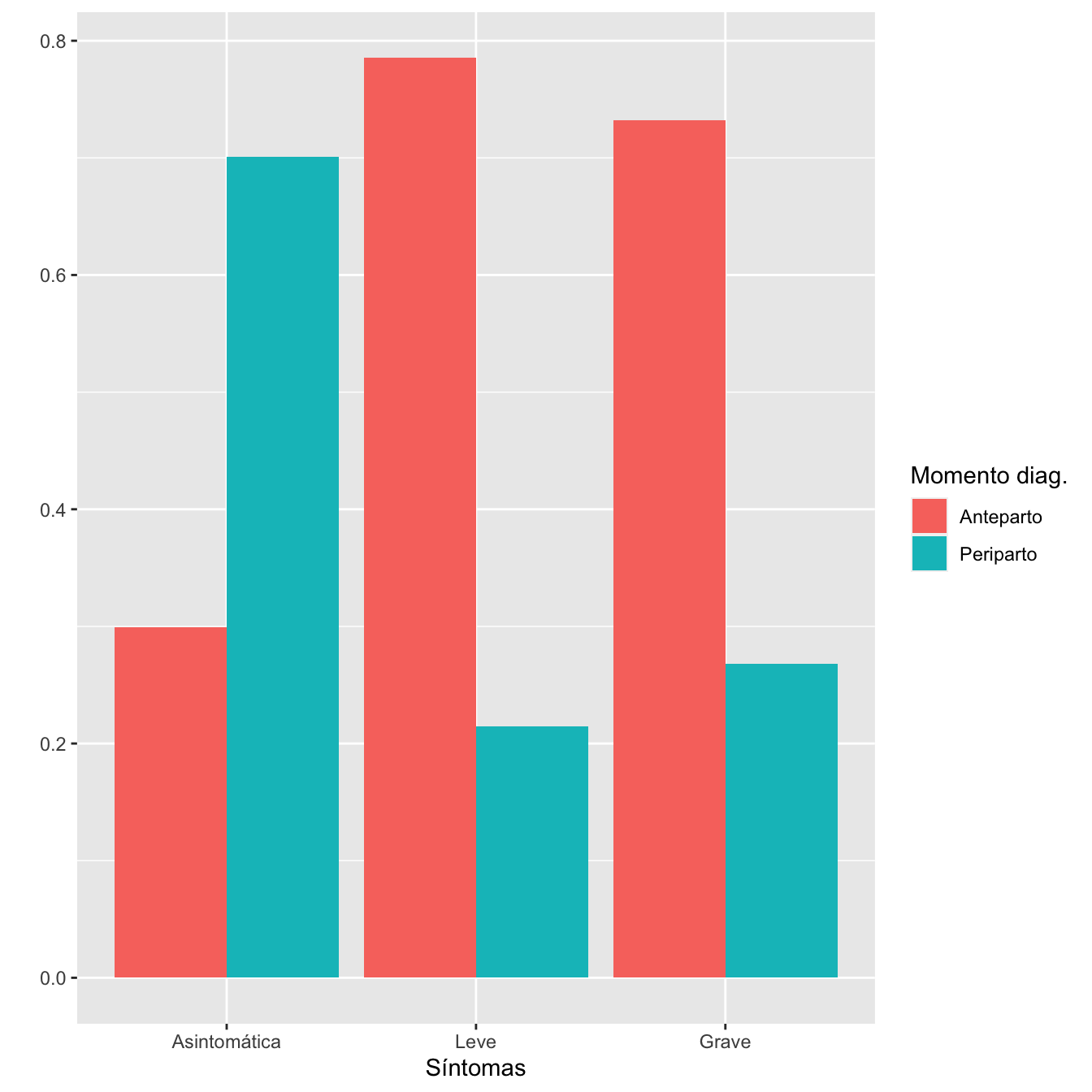
- Asociación entre los grupos de sintomatología y el momento del diagnóstico: test \(\chi^2\), p-valor \(2\times 10^{-81}\)
7.4.1 Preeclampsia
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| PE | 14 | 6.3 | 27 | 5.3 | 8 | 3.9 | 22 | 4.2 | 8 | 5.8 | 22 | 4.2 |
| No PE | 209 | 93.7 | 478 | 94.7 | 197 | 96.1 | 500 | 95.8 | 130 | 94.2 | 500 | 95.8 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.2 Preeclampsia con CG
Casos.PE=Casos[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1,]
I=Casos.PE$Preeclampsia.grave_HELLP_ECLAMPSIA
Sint.PE=Casos.PE$SINTOMAS_DIAGNOSTICO
TablaAntePeriCasos(Casos.PE,I, c("PE con CG", "PE sin CG"),Sint.PE)| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| PE con CG | 2 | 14.3 | 10 | 37 | 3 | 37.5 | 7 | 31.8 | 3 | 37.5 | 7 | 31.8 |
| PE sin CG | 12 | 85.7 | 17 | 63 | 5 | 62.5 | 15 | 68.2 | 5 | 62.5 | 15 | 68.2 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.3 Anomalías congénitas
I=Casos$Diagnóstico.de.malformación.ecográfica._.semana.20._
TablaAntePeriCasos(Casos,I, c("Anomalías congénitas", "No anomalías congénitas"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Anomalías congénitas | 3 | 1.4 | 10 | 2 | 3 | 1.5 | 8 | 1.6 | 4 | 2.9 | 8 | 1.6 |
| No anomalías congénitas | 209 | 98.6 | 485 | 98 | 193 | 98.5 | 500 | 98.4 | 132 | 97.1 | 500 | 98.4 |
| Datos perdidos | 11 | 10 | 9 | 14 | 2 | 14 |
7.4.4 Retraso del crecimiento intrauterino
I=Casos$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.
TablaAntePeriCasos(Casos,I, c("RCIU","No RCIU"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| RCIU | 18 | 8.1 | 15 | 3 | 6 | 2.9 | 14 | 2.7 | 5 | 3.6 | 14 | 2.7 |
| No RCIU | 205 | 91.9 | 490 | 97 | 199 | 97.1 | 508 | 97.3 | 133 | 96.4 | 508 | 97.3 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.5 Diabetes gestacional
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DG | 18 | 8.1 | 39 | 7.7 | 16 | 7.8 | 35 | 6.7 | 10 | 7.2 | 35 | 6.7 |
| No DG | 205 | 91.9 | 466 | 92.3 | 189 | 92.2 | 487 | 93.3 | 128 | 92.8 | 487 | 93.3 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.6 Hipertensión gestacional
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HG | 5 | 2.2 | 12 | 2.4 | 5 | 2.4 | 15 | 2.9 | 4 | 2.9 | 15 | 2.9 |
| No HG | 218 | 97.8 | 493 | 97.6 | 200 | 97.6 | 507 | 97.1 | 134 | 97.1 | 507 | 97.1 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.7 Rotura prematura de membranas
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| RPM | 23 | 10.3 | 71 | 14.1 | 26 | 12.7 | 80 | 15.3 | 26 | 18.8 | 80 | 15.3 |
| No RPM | 200 | 89.7 | 434 | 85.9 | 179 | 87.3 | 442 | 84.7 | 112 | 81.2 | 442 | 84.7 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.8 Ingreso materno en UCI
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| UCI | 1 | 0.4 | 3 | 0.6 | 15 | 7.3 | 2 | 0.4 | 4 | 2.9 | 2 | 0.4 |
| No UCI | 222 | 99.6 | 502 | 99.4 | 190 | 92.7 | 520 | 99.6 | 134 | 97.1 | 520 | 99.6 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.9 Ingreso materno en UCI según el momento del parto o cesárea
Casos.UCIM=Casos[Casos$UCI==1,]
I=Casos.UCIM$UCI_ANTES.DESPUES.DEL.PARTO
Sint.UCIM=Casos.UCIM$SINTOMAS_DIAGNOSTICO
TablaAntePeriCasos(Casos.UCIM,I, c("Antes del parto", "Después del parto"),Sint.UCIM)| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Antes del parto | 2 | 66.7 | 8 | 53.3 | 3 | 75 | ||||||
| Después del parto | 1 | 100 | 1 | 33.3 | 7 | 46.7 | 2 | 100 | 1 | 25 | 2 | 100 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.10 Feto muerto anteparto
Casos.Fetos=rbind(Casos,CasosGM)
I1=Casos$Feto.muerto.intraútero
I1[I1=="Sí"]=1
I1[I1=="No"]=0
I2=CasosGM$Feto.vivo
I2[I2=="Sí"]=0
I2[I2=="No"]=1
I=c(I1,I2)
Sint.Fetos=Casos.Fetos$SINTOMAS_DIAGNOSTICO
#TablaAntePeriCasos(Casos.Fetos,c("Feto muerto anteparto","Feto vivo"), Sint.Fetos)
CasosX=Casos.Fetos
SintX=Sint.Fetos
L=c("Feto muerto anteparto","Feto vivo")
I.AP.A=I[CasosX$PreP==1& Sint=="Asintomática"]
I.AP.L=I[CasosX$PreP==1& SintX=="Leve"]
I.AP.G=I[CasosX$PreP==1& SintX=="Grave"]
I.PP.A=I[CasosX$PreP==0& SintX=="Asintomática"]
I.PP.L=I[CasosX$PreP==0& SintX=="Leve"]
I.PP.G=I[CasosX$PreP==0& SintX=="Asintomática"]
NA_I.AP.A=length(I.AP.A[is.na(I.AP.A)])
NA_I.AP.L=length(I.AP.L[is.na(I.AP.L)])
NA_I.AP.G=length(I.AP.G[is.na(I.AP.G)])
NA_I.PP.A=length(I.PP.A[is.na(I.PP.A)])
NA_I.PP.L=length(I.PP.L[is.na(I.PP.L)])
NA_I.PP.G=length(I.PP.G[is.na(I.PP.G)])
EEExt=rbind(c(as.vector(table(I.AP.A))[2:1],NA_I.AP.A),
c(round(100*as.vector(prop.table(table(I.AP.A)))[2:1],1),NA),
c(as.vector(table(I.AP.L))[2:1],NA_I.AP.L),
c(round(100*as.vector(prop.table(table(I.AP.L)))[2:1],1),NA),
c(as.vector(table(I.AP.G))[2:1],NA_I.AP.G),
c(round(100*as.vector(prop.table(table(I.AP.G)))[2:1],1),NA),
c(as.vector(table(I.PP.A))[2:1],NA_I.PP.A),
c(round(100*as.vector(prop.table(table(I.PP.A)))[2:1],1),NA),
c(as.vector(table(I.PP.L))[2:1],NA_I.PP.L),
c(round(100*as.vector(prop.table(table(I.PP.L)))[2:1],1),NA),
c(as.vector(table(I.PP.G))[2:1],NA_I.PP.G),
c(round(100*as.vector(prop.table(table(I.PP.G)))[2:1],1),NA)
)
colnames(EEExt)=c(L,"Datos perdidos")
rownames(EEExt)=c("Inf. AP. Asintomáticas (N)", "Inf. AP. Asintomáticas (%)",
"Inf. AP. Leves (N)", "Inf. AP. Leves (%)",
"Inf. AP. Graves (N)", "Inf. AP. Graves (%)",
"Inf. PP. Asintomáticas (N)", "Inf. PP. Asintomáticas (%)",
"Inf. PP. Leves (N)", "Inf. PP. Leves (%)",
"Inf. PP. Graves (N)", "Inf. PP. Graves (%)")
t(EEExt) %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;") | Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Feto muerto anteparto | 1 | 0.4 | 4 | 0.8 | 1 | 0.5 | 7 | 1.3 | 5 | 3.6 | 7 | 1.3 |
| Feto vivo | 229 | 99.6 | 515 | 99.2 | 207 | 99.5 | 520 | 98.7 | 135 | 96.4 | 520 | 98.7 |
| Datos perdidos | 1 | 1 | 0 | 0 | 0 | 0 |
7.4.11 Prematurez
| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Prematuro | 23 | 10.3 | 34 | 6.7 | 32 | 15.6 | 30 | 5.7 | 29 | 21 | 30 | 5.7 |
| A término | 200 | 89.7 | 471 | 93.3 | 173 | 84.4 | 492 | 94.3 | 109 | 79 | 492 | 94.3 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.12 Eventos trombóticos
I=Casos$EVENTOS_TROMBO_TOTALES
TablaAntePeriCasos(Casos,I, c("Eventos trombóticos","No eventos trombóticos"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos trombóticos | 4 | 2 | 1 | 0.2 | 1 | 0.2 | ||||||
| No eventos trombóticos | 223 | 100 | 505 | 100 | 201 | 98 | 521 | 99.8 | 138 | 100 | 521 | 99.8 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.13 Eventos hemorrágicos
I=Casos$EVENTOS_HEMORRAGICOS_TOTAL
TablaAntePeriCasos(Casos,I, c("Eventos hemorrágicos","No eventos hemorrágicos"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos hemorrágicos | 6 | 2.7 | 29 | 5.7 | 10 | 4.9 | 23 | 4.4 | 10 | 7.2 | 23 | 4.4 |
| No eventos hemorrágicos | 217 | 97.3 | 476 | 94.3 | 195 | 95.1 | 499 | 95.6 | 128 | 92.8 | 499 | 95.6 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.14 Hemorragias potsparto
I=Casos$Hemorragia.postparto
I.sino=I
I.sino[I.sino!="No" & !is.na(I.sino)]="Sí"
TablaAntePeriCasos(Casos,I.sino, c("HPP","No HPP"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HPP | 5 | 2.3 | 21 | 4.2 | 9 | 4.5 | 17 | 3.3 | 9 | 6.6 | 17 | 3.3 |
| No HPP | 214 | 97.7 | 480 | 95.8 | 191 | 95.5 | 496 | 96.7 | 127 | 93.4 | 496 | 96.7 |
| Datos perdidos | 4 | 4 | 5 | 9 | 2 | 9 |
I=ordered(I,levels=names(table(I))[c(2,3,1,4)])
TablaAntePeriCasos.m(Casos,I,c("Tto. médico",
"Tto. Qx. conservador","Histerectomía",
"No HPP"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Tto. médico | 4 | 1.8 | 18 | 3.6 | 8 | 4.0 | 16 | 3.1 | 5 | 3.7 | 16 | 3.1 |
| Tto. Qx. conservador | 1 | 0.5 | 2 | 0.4 | 1 | 0.5 | 1 | 0.2 | 2 | 1.5 | 1 | 0.2 |
| Histerectomía | 0 | 0.0 | 1 | 0.2 | 0 | 0.0 | 0 | 0.0 | 2 | 1.5 | 0 | 0.0 |
| No HPP | 214 | 97.7 | 480 | 95.8 | 191 | 95.5 | 496 | 96.7 | 127 | 93.4 | 496 | 96.7 |
| Datos perdidos | 4 | 4 | 5 | 9 | 2 | 9 |
- Diferencia entre infectadas ante y periparto: test \(\chi^2\) de Montecarlo, p-valor NaN
7.4.15 Inicio del parto
I=Casos$Inicio.de.parto
I=factor(Casos$Inicio.de.parto,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
TablaAntePeriCasos.m(Casos,I,c("Espontáneo", "Inducido", "Cesárea"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Espontáneo | 111 | 49.8 | 263 | 52.1 | 93 | 45.4 | 312 | 59.8 | 69 | 50.4 | 312 | 59.8 |
| Inducido | 92 | 41.3 | 194 | 38.4 | 79 | 38.5 | 181 | 34.7 | 52 | 38.0 | 181 | 34.7 |
| Cesárea | 20 | 9.0 | 48 | 9.5 | 33 | 16.1 | 29 | 5.6 | 16 | 11.7 | 29 | 5.6 |
| Datos perdidos | 0 | 0 | 0 | 0 | 1 | 0 |
7.4.16 Tipo de parto
I.In=Casos$Inicio.de.parto
I.In[I.In=="Cesárea"]="Cesárea programada"
I=Casos$Tipo.de.parto
I[I.In=="Cesárea programada"]="Cesárea programada"
I[I=="Cesárea"]="Cesárea urgente"
I=factor(I,levels=c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
TablaAntePeriCasos.m(Casos,I,c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"))| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eutócico | 139 | 62.3 | 336 | 66.5 | 131 | 63.9 | 349 | 66.9 | 76 | 55.1 | 349 | 66.9 |
| Instrumental | 27 | 12.1 | 56 | 11.1 | 10 | 4.9 | 58 | 11.1 | 15 | 10.9 | 58 | 11.1 |
| Cesárea programada | 20 | 9.0 | 48 | 9.5 | 33 | 16.1 | 29 | 5.6 | 16 | 11.6 | 29 | 5.6 |
| Cesárea urgente | 37 | 16.6 | 65 | 12.9 | 31 | 15.1 | 86 | 16.5 | 31 | 22.5 | 86 | 16.5 |
| Datos perdidos | 0 | 0 | 0 | 0 | 0 | 0 |
7.4.17 Test de Apgar
I=c(Casos$APGAR.5...126,CasosGM$APGAR.5...150)
I.cut=cut(I,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I.cut=ordered(I.cut,levels=c("8-10","0-7"))
TablaAntePeriCasos(Casos.Fetos,I.cut,c("Apgar.5≤7", "Apgar.5≥8"),Sint.Fetos)| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Apgar.5≤7 | 4 | 1.8 | 12 | 2.3 | 4 | 2 | 15 | 2.9 | 8 | 5.8 | 15 | 2.9 |
| Apgar.5≥8 | 216 | 98.2 | 504 | 97.7 | 201 | 98 | 508 | 97.1 | 129 | 94.2 | 508 | 97.1 |
| Datos perdidos | 6 | 4 | 3 | 4 | 3 | 4 |
7.4.18 Ingreso del neonato en UCI
Casos.FetosVivos=rbind(Casos[Casos$Feto.muerto.intraútero=="No",],CasosGM[CasosGM$Feto.vivo=="Sí",])
Sint.FetosVivos=Casos.FetosVivos$SINTOMAS_DIAGNOSTICO
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,CasosGM[CasosGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
TablaAntePeriCasos(Casos.FetosVivos,I,c("UCIN", "No UCIN"),Sint.FetosVivos)| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| UCIN | 13 | 5.9 | 35 | 6.9 | 23 | 11.2 | 39 | 7.6 | 20 | 15 | 39 | 7.6 |
| No UCIN | 207 | 94.1 | 475 | 93.1 | 182 | 88.8 | 475 | 92.4 | 113 | 85 | 475 | 92.4 |
| Datos perdidos | 6 | 6 | 3 | 7 | 3 | 7 |
7.4.19 Peso de neonatos
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí",]$Peso._gramos_...247)
I.AP.A=I[Casos$PreP==1&Casos$SINTOMAS_DIAGNOSTICO=="Asintomática"]
I.AP.L=I[Casos$PreP==1&Casos$SINTOMAS_DIAGNOSTICO=="Leve"]
I.AP.G=I[Casos$PreP==1&Casos$SINTOMAS_DIAGNOSTICO=="Grave"]
I.PP.A=I[Casos$PreP==0&Casos$SINTOMAS_DIAGNOSTICO=="Asintomática"]
I.PP.L=I[Casos$PreP==0&Casos$SINTOMAS_DIAGNOSTICO=="Leve"]
I.PP.G=I[Casos$PreP==0&Casos$SINTOMAS_DIAGNOSTICO=="Grave"]Dades=rbind(c(round(min(I.AP.A,na.rm=TRUE),1),round(max(I.AP.A,na.rm=TRUE),1), round(mean(I.AP.A,na.rm=TRUE),1),round(median(I.AP.A,na.rm=TRUE),1),round(quantile(I.AP.A,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.AP.A,na.rm=TRUE),1)),
c(round(min(I.AP.L,na.rm=TRUE),1),round(max(I.AP.L,na.rm=TRUE),1), round(mean(I.AP.L,na.rm=TRUE),1),round(median(I.AP.L,na.rm=TRUE),1),round(quantile(I.AP.L,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.AP.L,na.rm=TRUE),1)),
c(round(min(I.AP.G,na.rm=TRUE),1),round(max(I.AP.G,na.rm=TRUE),1), round(mean(I.AP.G,na.rm=TRUE),1),round(median(I.AP.G,na.rm=TRUE),1),round(quantile(I.AP.G,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.AP.G,na.rm=TRUE),1)),
c(round(min(I.PP.A,na.rm=TRUE),1),round(max(I.PP.A,na.rm=TRUE),1), round(mean(I.PP.A,na.rm=TRUE),1),round(median(I.PP.A,na.rm=TRUE),1),round(quantile(I.PP.A,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.PP.A,na.rm=TRUE),1)),
c(round(min(I.PP.L,na.rm=TRUE),1),round(max(I.PP.L,na.rm=TRUE),1), round(mean(I.PP.L,na.rm=TRUE),1),round(median(I.PP.L,na.rm=TRUE),1),round(quantile(I.PP.L,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.PP.L,na.rm=TRUE),1)),
c(round(min(I.PP.G,na.rm=TRUE),1),round(max(I.PP.G,na.rm=TRUE),1), round(mean(I.PP.G,na.rm=TRUE),1),round(median(I.PP.G,na.rm=TRUE),1),round(quantile(I.PP.G,c(0.25,0.75),na.rm=TRUE),1), round(sd(I.PP.G,na.rm=TRUE),1)))
colnames(Dades)=c("Peso mínimo","Peso máximo","Peso medio", "Peso mediano", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Inf. AP. asintómaticas", "Inf. AP. leves", "Inf. AP. graves","Inf. PP. asintómaticas", "Inf. PP. leves", "Inf. PP. graves")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Peso mínimo | Peso máximo | Peso medio | Peso mediano | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Inf. AP. asintómaticas | 315 | 4740 | 3197.2 | 3300.0 | 2940.0 | 3532.5 | 635.9 |
| Inf. AP. leves | 585 | 4750 | 3232.3 | 3292.5 | 2928.8 | 3600.0 | 564.3 |
| Inf. AP. graves | 980 | 4640 | 3169.9 | 3205.0 | 2860.0 | 3550.0 | 597.0 |
| Inf. PP. asintómaticas | 1100 | 4720 | 3221.1 | 3265.5 | 2912.5 | 3597.5 | 573.0 |
| Inf. PP. leves | 940 | 4540 | 3202.1 | 3230.0 | 2940.0 | 3526.2 | 585.1 |
| Inf. PP. graves | 680 | 5190 | 3114.0 | 3205.0 | 2817.5 | 3462.5 | 711.4 |
Bajo peso
I.cut=cut(I,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
I.cut=ordered(I.cut,levels=c(0,1))
TablaAntePeriCasos(Casos.FetosVivos,I.cut,c("Bajo peso", "No bajo peso"),Sint.FetosVivos)| Inf. AP. Asintomáticas (N) | Inf. AP. Asintomáticas (%) | Inf. AP. Leves (N) | Inf. AP. Leves (%) | Inf. AP. Graves (N) | Inf. AP. Graves (%) | Inf. PP. Asintomáticas (N) | Inf. PP. Asintomáticas (%) | Inf. PP. Leves (N) | Inf. PP. Leves (%) | Inf. PP. Graves (N) | Inf. PP. Graves (%) | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Bajo peso | 28 | 13.1 | 30 | 6.1 | 23 | 11.6 | 35 | 6.9 | 21 | 16.5 | 35 | 6.9 |
| No bajo peso | 186 | 86.9 | 463 | 93.9 | 176 | 88.4 | 473 | 93.1 | 106 | 83.5 | 473 | 93.1 |
| Datos perdidos | 12 | 23 | 9 | 13 | 9 | 13 |