Lección 8 Comparación de las dos olas
8.1 Gravedad en la infeccion
Sint1w=Casos1w$SINTOMAS_DIAGNOSTICO
Sint2w=Casos2w$SINTOMAS_DIAGNOSTICO
Sint=c(Sint1w,Sint2w)
EEExt=rbind(c(table(Sint1w)[1],round(100*prop.table(table(Sint1w)),2)[1],
table(Sint1w)[2],round(100*prop.table(table(Sint1w)),2)[2],
table(Sint1w)[3],round(100*prop.table(table(Sint1w)),2)[3],
length(Sint1w)),
c(table(Sint2w)[1],round(100*prop.table(table(Sint2w)),2)[1],
table(Sint2w)[2],round(100*prop.table(table(Sint2w)),2)[2],
table(Sint2w)[3],round(100*prop.table(table(Sint2w)),2)[3],
length(Sint2w))
)
EEE=EEExt[,c(1,3,5)]
rownames(EEExt)=c("Infectadas 1a ola", "Infectadas 2a ola")
colnames(EEExt)=c("Asintomáticas (N)", "Asintomáticas (%)","Síntomas leves (N)", "Síntomas leves (%)", "Neumonía (N)", "Neumonía (%)","Total")
EEExt %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Asintomáticas (N) | Asintomáticas (%) | Síntomas leves (N) | Síntomas leves (%) | Neumonía (N) | Neumonía (%) | Total | |
|---|---|---|---|---|---|---|---|
| Infectadas 1a ola | 357 | 40.71 | 314 | 35.80 | 206 | 23.49 | 877 |
| Infectadas 2a ola | 388 | 49.05 | 329 | 41.59 | 74 | 9.36 | 791 |
df =data.frame(
Ola=c(rep("1a ola",length(Sint1w)), rep("2a ola",length(Sint2w))),
Sintomas=c(Sint1w,Sint2w)
)
Gravedad=ordered(rep(c("Asintomáticas", "Síntomas leves", "Neumonía"), each=2),levels=c("Asintomáticas", "Síntomas leves", "Neumonía"))
Grupo=rep(c("1a ola","2a ola") , 3)
valor=as.vector(prop.table(table(df), margin=1))
data <- data.frame(Gravedad,Grupo,valor)
ggplot(data, aes(fill=Gravedad, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("Olas")+
ylab("")+
scale_fill_brewer(palette="Dark2")+labs(fill="")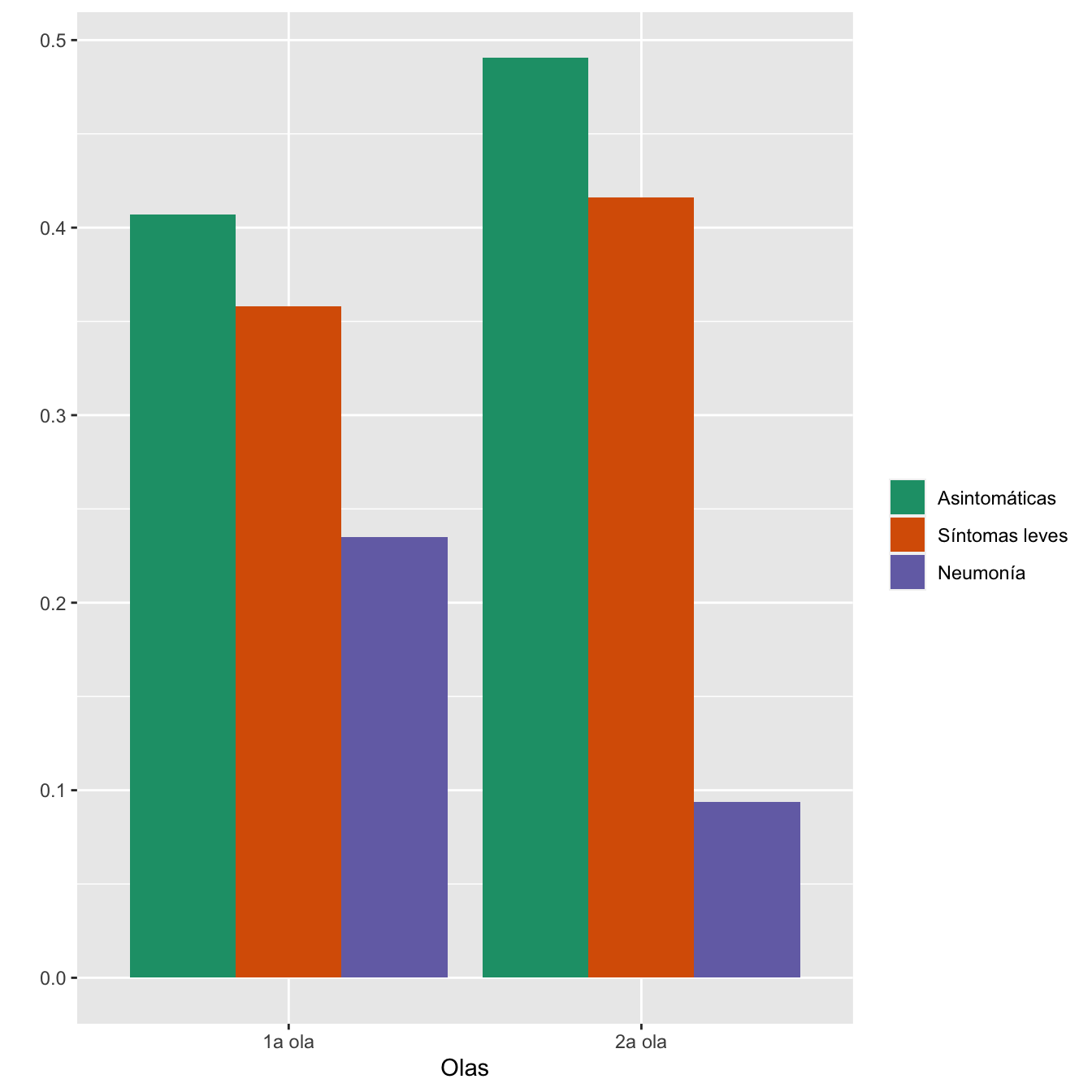
Figura 8.1:
- Asociación entre los grupos de sintomatología y las olas: test \(\chi^2\), p-valor \(10^{-13}\)
8.2 Comparación de antecedentes
8.2.1 Franja de edad
ECa1=Casos1w$Edad._años_
ECa2=Casos2w$Edad._años_
ECo1=Controls1w$Edad._años_
ECo2=Controls2w$Edad._años_
fligner.test(list(ECa1,ECa2))$p.value## [1] 0.166121## [1] 0.707814Igualdad de edades medias entre olas
Para los casos: test t, p-valor \(3\times 10^{-9}\); IC-95% para la diferencia de medias [1.209, 2.387]
Para los controles: test t, p-valor 0.244; IC-95% para la difound(erencia de medias [-0.258, 1.014]
En tablas como la que sigue, para cada fila considerada como antecedente:
- Cociente de OR: cociente de la OR de infección relativa al antecedente en la 1a ola entre la correspondiente OR en la 2a ola
- p-valor igualdad OR: p-valor del contraste de igualdad entre las OR de infección relativas al antecedente en las dos olas, ajustados por Bonferroni
- IC: IC-95% para el cociente de las OR
- p-valor global olas: p-valor del test de Fisher de la igualdad de proporciones del antecendente en las dos olas, ajustados por Bonferroni
- p-valor casos: p-valor del test de Fisher de la igualdad de proporciones del antecendente entre las infectadas de las dos olas, ajustados por Bonferroni
- p-valor controles: p-valor del test de Fisher de la igualdad de proporciones del antecendente entre las no infectadas de las dos olas, ajustados por Bonferroni
I1=cut(Casos1w$Edad._años_,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
NI1=cut(Controls1w$Edad._años_,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
I2=cut(Casos2w$Edad._años_,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
NI2=cut(Controls2w$Edad._años_,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
#
ComparOlasCCm(I1,I2,NI1,NI2,c("18-30","31-40",">40"))| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 18-30 | 286 | 32.8 | 416 | 35.3 | 361 | 45.7 | 151 | 37.2 | 0.63 | 0.46 | 0.86 | 0.009300 | 0.000004 | 0.000000 | 1 |
| 31-40 | 516 | 59.1 | 695 | 58.9 | 385 | 48.7 | 236 | 58.1 | 1.47 | 1.09 | 1.98 | 0.035766 | 0.000261 | 0.000068 | 1 |
| >40 | 71 | 8.1 | 68 | 5.8 | 44 | 5.6 | 19 | 4.7 | 1.20 | 0.63 | 2.31 | 1.000000 | 0.291949 | 0.126685 | 1 |
| Datos perdidos | 4 | 12 | 1 | 10 |
Tipo= c(I1,I2,NI1,NI2)
dtbp=data.frame(Grupo=c(rep("Inf.",length(I1)+length(I2)),rep("No inf.",length(NI1)+length(NI2))),
Ola= c(rep("1a ola",length(I1)),rep("2a ola",length(I2)),rep("1a ola",length(NI1)),rep("2a ola",length(NI2))),
Tipo
)
SRH=scheirerRayHare(Tipo ~ Grupo + Ola,data=dtbp,verbose=FALSE)$p.value[3]- Test de interacción de Scheirer-Ray-Hare: p-valor, 0.003. Un p-valor pequeño da evidencia estadística de que las diferencia de la “franja mediana de edad” en infectadas y no infectadas en las dos olas han sido diferentes.
8.2.2 Etnia
I1=Casos1w$Etnia
NI1=Controls1w$Etnia
I2=Casos2w$Etnia
NI2=Controls2w$Etnia
I1[I1=="Asia"]="Asiática"
I1=factor(I1)
I2[I2=="Asia"]="Asiática"
I2=factor(I2)
#
ComparOlasCCm(I1,I2,NI1,NI2,c("Árabe", "Asiática", "Blanca", "Latinoamericana","Negra"))| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Árabe | 54 | 6.2 | 108 | 9.1 | 87 | 11.0 | 31 | 7.5 | 0.43 | 0.25 | 0.75 | 0.013101 | 0.000000 | 0.002044 | 1 |
| Asiática | 24 | 2.7 | 27 | 2.3 | 25 | 3.2 | 14 | 3.4 | 1.30 | 0.55 | 3.10 | 1.000000 | 0.255819 | 1.000000 | 1 |
| Blanca | 537 | 61.4 | 922 | 77.7 | 387 | 49.0 | 321 | 77.9 | 1.68 | 1.20 | 2.34 | 0.011709 | 0.000894 | 0.000002 | 1 |
| Latinoamericana | 240 | 27.5 | 114 | 9.6 | 265 | 33.6 | 41 | 10.0 | 0.78 | 0.51 | 1.20 | 1.000000 | 0.000000 | 0.037803 | 1 |
| Negra | 19 | 2.2 | 16 | 1.3 | 25 | 3.2 | 5 | 1.2 | 0.61 | 0.19 | 1.98 | 1.000000 | 0.035735 | 1.000000 | 1 |
| Datos perdidos | 3 | 4 | 2 | 4 |
Como las etnias no son ordinales, no hay ningún test sensato para comparar si la diferencia entre casos y controles en la primera ola és “diferente” de la diferencia entre casos y controles en segunda ola.
8.2.3 Hábito tabáquico
En tablas como la que sigue:
- Cociente de OR: cociente de la OR de infección relativa al antecedente en la 1a ola entre la correspondiente OR en la 2a ola
- p-valor igualdad OR: p-valor del contraste de igualdad entre las OR de infección relativas al antecedente en las dos olas;
- IC: IC-95% para el cociente de las OR
- p-valor global olas: p-valor del test de Fisher de la igualdad de proporciones del antecendente en las dos olas
- p-valor casos: p-valor del test de Fisher de la igualdad de proporciones del antecendente entre las infectadas de las dos olas
- p-valor controles: p-valor del test de Fisher de la igualdad de proporciones del antecendente entre las no infectadas de las dos olas
I1=Casos1w$FUMADORA_CAT
I2=Casos2w$FUMADORA_CAT
NI1=Controls1w$Fumadora_cat
NI2=Controls2w$Fumadora_cat
#
ComparOlasCC(I1,I2,NI1,NI2,"Fumadoras","No fumadoras")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Fumadoras | 96 | 11.4 | 144 | 12.9 | 57 | 7.4 | 49 | 12.7 | 1.5923 | 0.9772 | 2.5947 | 0.061867 | 0.009313 | 0.006423 | 1 |
| No fumadoras | 743 | 88.6 | 976 | 87.1 | 711 | 92.6 | 336 | 87.3 | |||||||
| Datos perdidos | 38 | 71 | 23 | 31 |
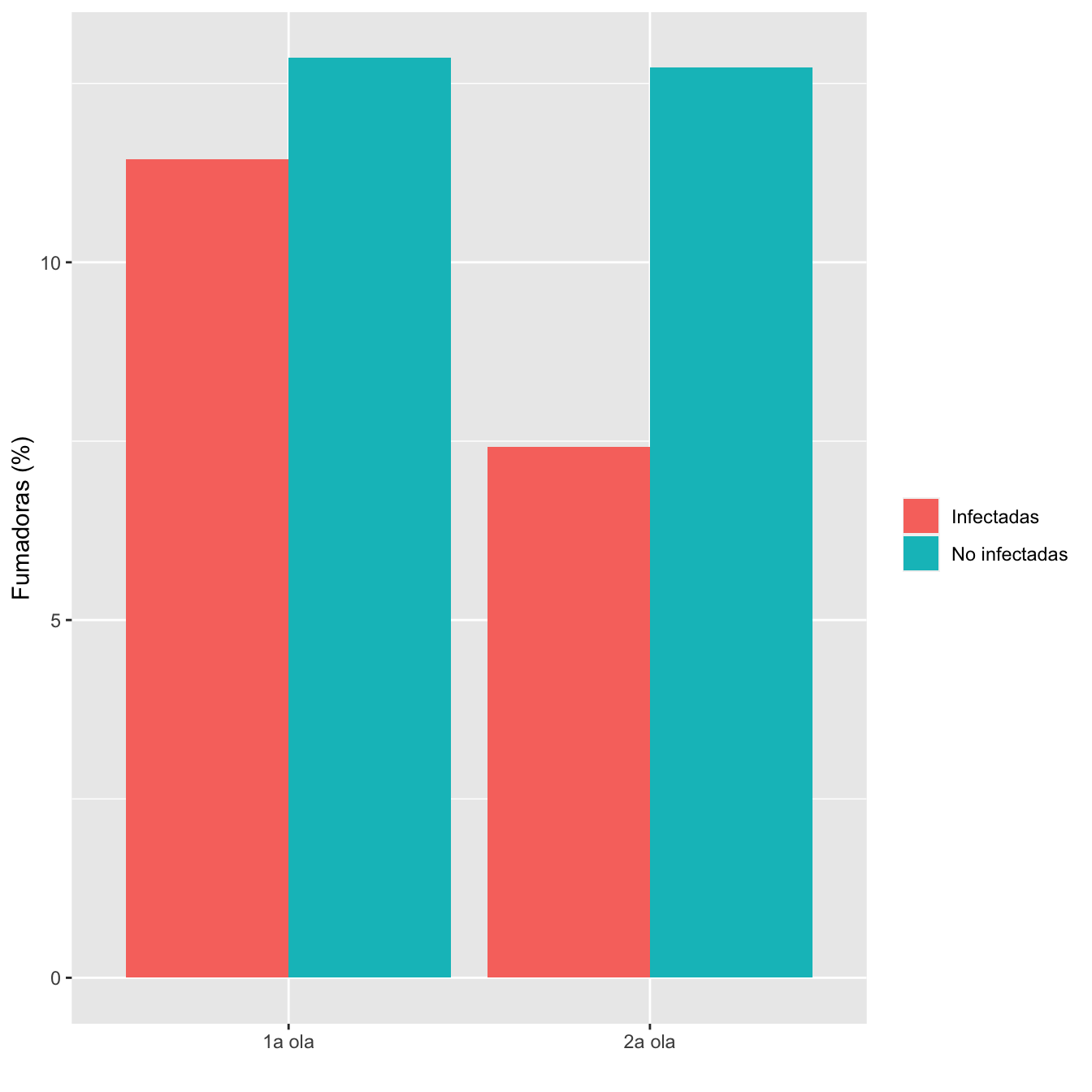
Figura 8.2:
8.2.4 Obesidad
I1=Casos1w$Obesidad
I2=Casos2w$Obesidad
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Obesidad
NI2=Controls2w$Obesidad
#
ComparOlasCC(I1,I2,NI1,NI2,"Obesas","No obesas")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Obesas | 161 | 18.4 | 198 | 17.5 | 144 | 18.2 | 51 | 13.4 | 0.7353 | 0.4855 | 1.1136 | 0.14649 | 0.383481 | 0.949494 | 0.066203 |
| No obesas | 716 | 81.6 | 935 | 82.5 | 647 | 81.8 | 331 | 86.6 | |||||||
| Datos perdidos | 0 | 58 | 0 | 34 |
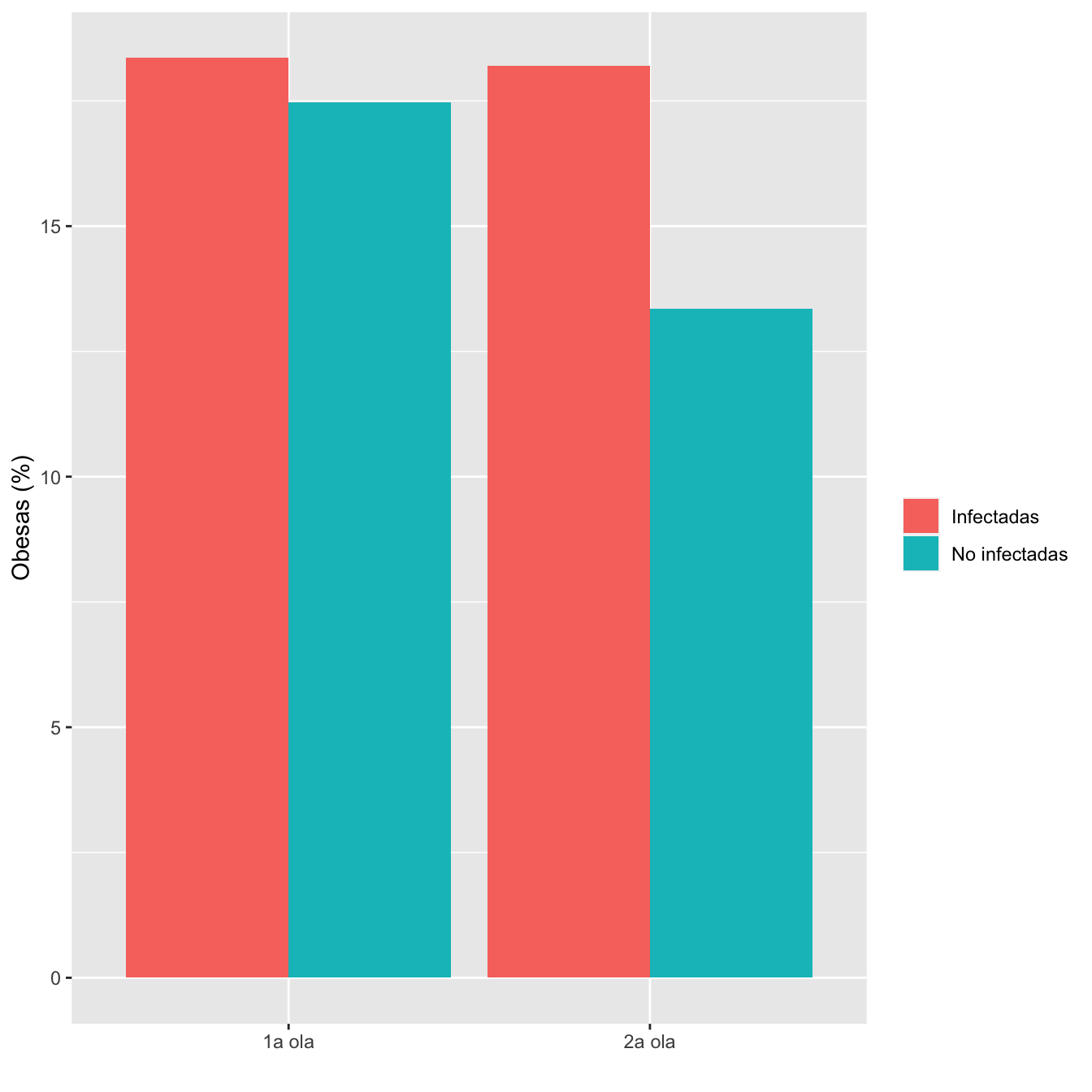
Figura 8.3:
8.2.5 Hipertensión pregestacional
I1=Casos1w$Hipertensión.pregestacional
I2=Casos2w$Hipertensión.pregestacional
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Hipertensión.pregestacional
NI2=Controls2w$Hipertensión.pregestacional
#
ComparOlasCC(I1,I2,NI1,NI2,"HTA","No HTA")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HTA | 14 | 1.6 | 13 | 1.1 | 11 | 1.4 | 4 | 1.1 | 1.0628 | 0.2676 | 4.2216 | 0.931042 | 1 | 0.840958 | 1 |
| No HTA | 863 | 98.4 | 1123 | 98.9 | 780 | 98.6 | 374 | 98.9 | |||||||
| Datos perdidos | 0 | 55 | 0 | 38 |
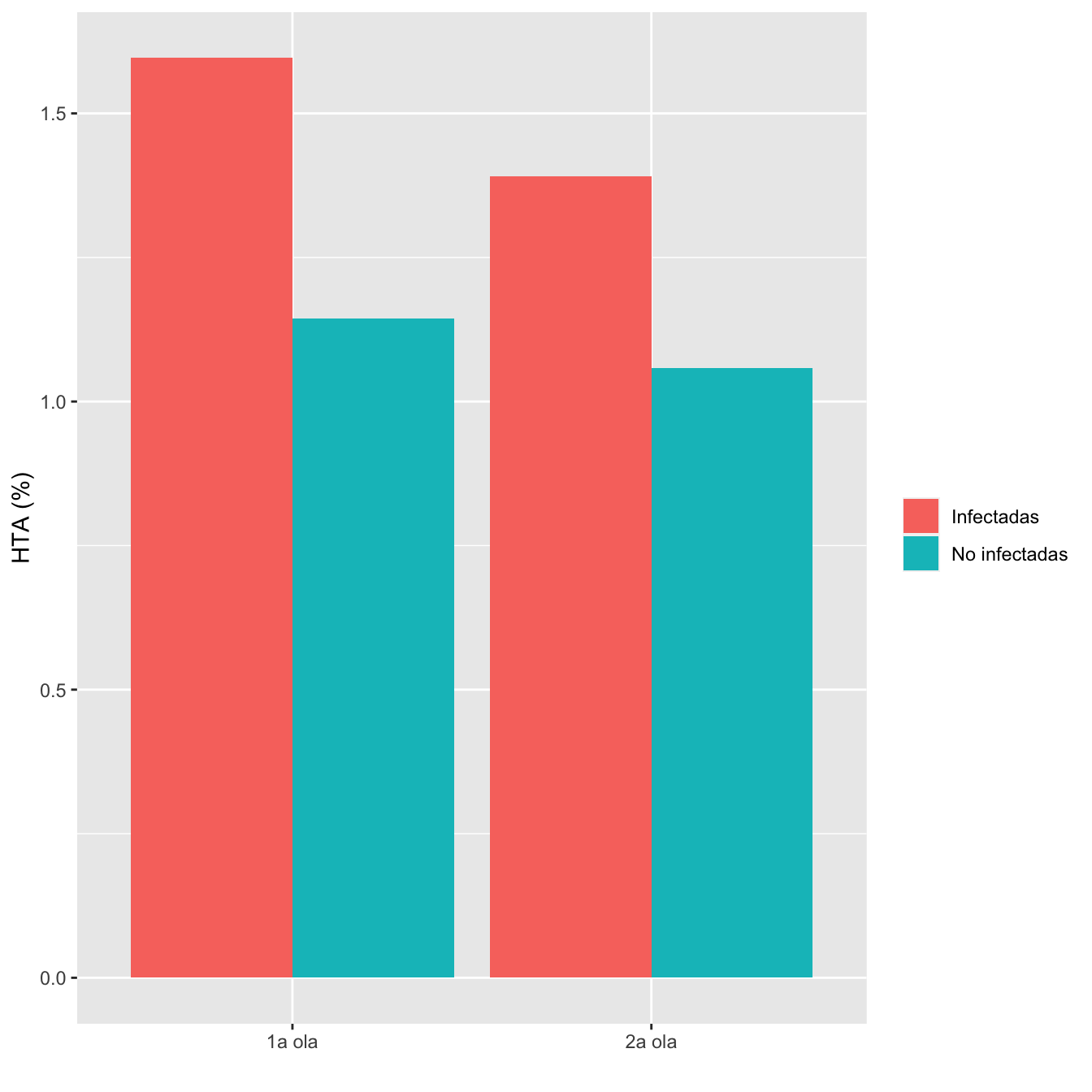
Figura 8.4:
8.2.6 Diabetes Mellitus
I1=Casos1w$DIABETES
I2=Casos2w$DIABETES
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Diabetes
NI2=Controls2w$Diabetes
#
ComparOlasCC(I1,I2,NI1,NI2,"DM","No DM")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DM | 14 | 1.6 | 17 | 1.4 | 21 | 2.7 | 11 | 2.6 | 1.1156 | 0.3995 | 3.1156 | 0.83458 | 0.024607 | 0.170512 | 0.125623 |
| No DM | 863 | 98.4 | 1174 | 98.6 | 770 | 97.3 | 405 | 97.4 | |||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
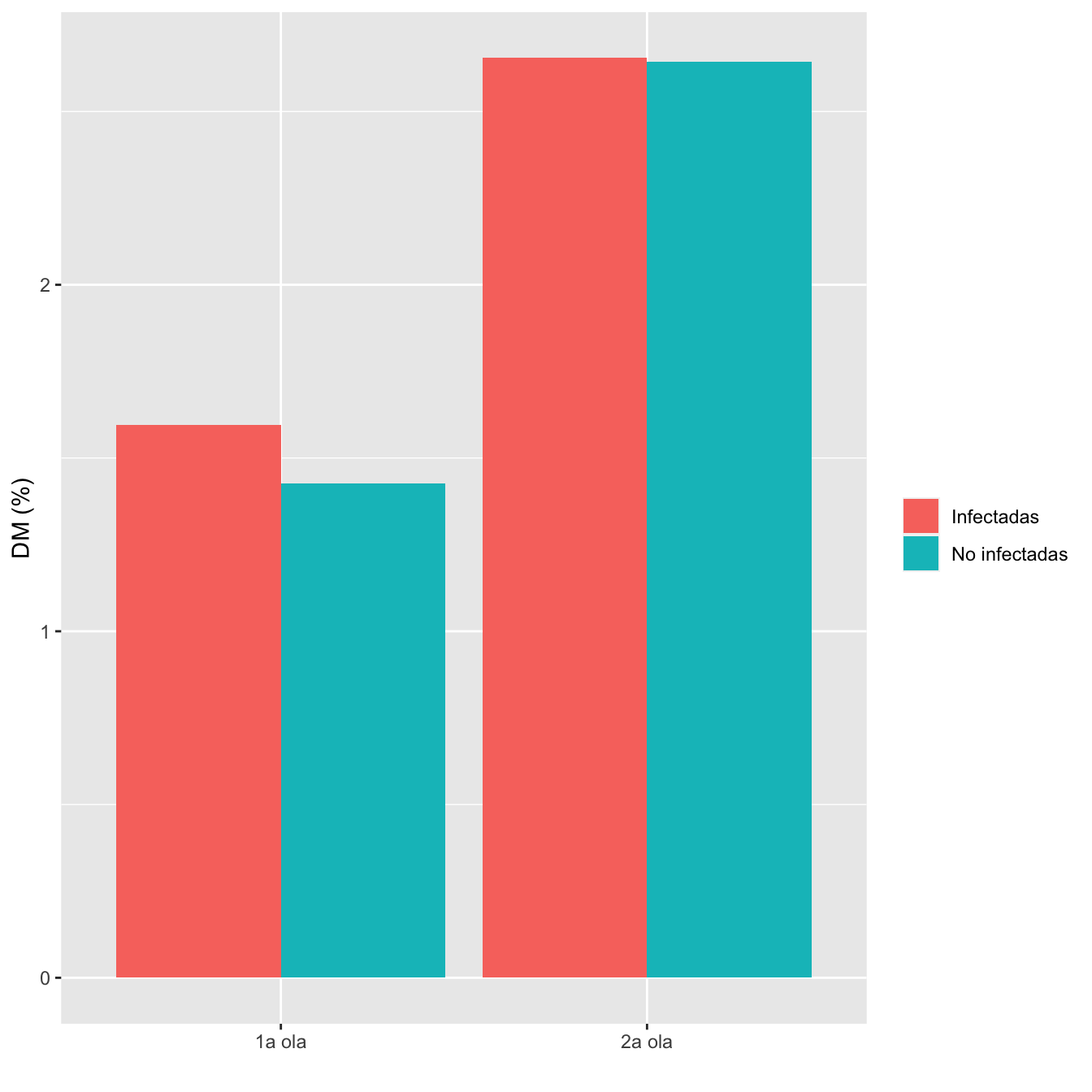
Figura 8.5:
8.2.7 Enfermedades cardiacas crónicas
I1=Casos1w$Enfermedad.cardiaca.crónica
I2=Casos2w$Enfermedad.cardiaca.crónica
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$ENF.CARDIACA.TODO
NI2=Controls2w$ENF.CARDIACA.TODO
#
ComparOlasCC(I1,I2,NI1,NI2,"ECC","No ECC")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ECC | 11 | 1.3 | 21 | 1.8 | 8 | 1 | 6 | 1.6 | 1.057 | 0.2897 | 3.8568 | 0.933077 | 0.442291 | 0.818066 | 0.826361 |
| No ECC | 866 | 98.7 | 1134 | 98.2 | 783 | 99 | 381 | 98.4 | |||||||
| Datos perdidos | 0 | 36 | 0 | 29 |
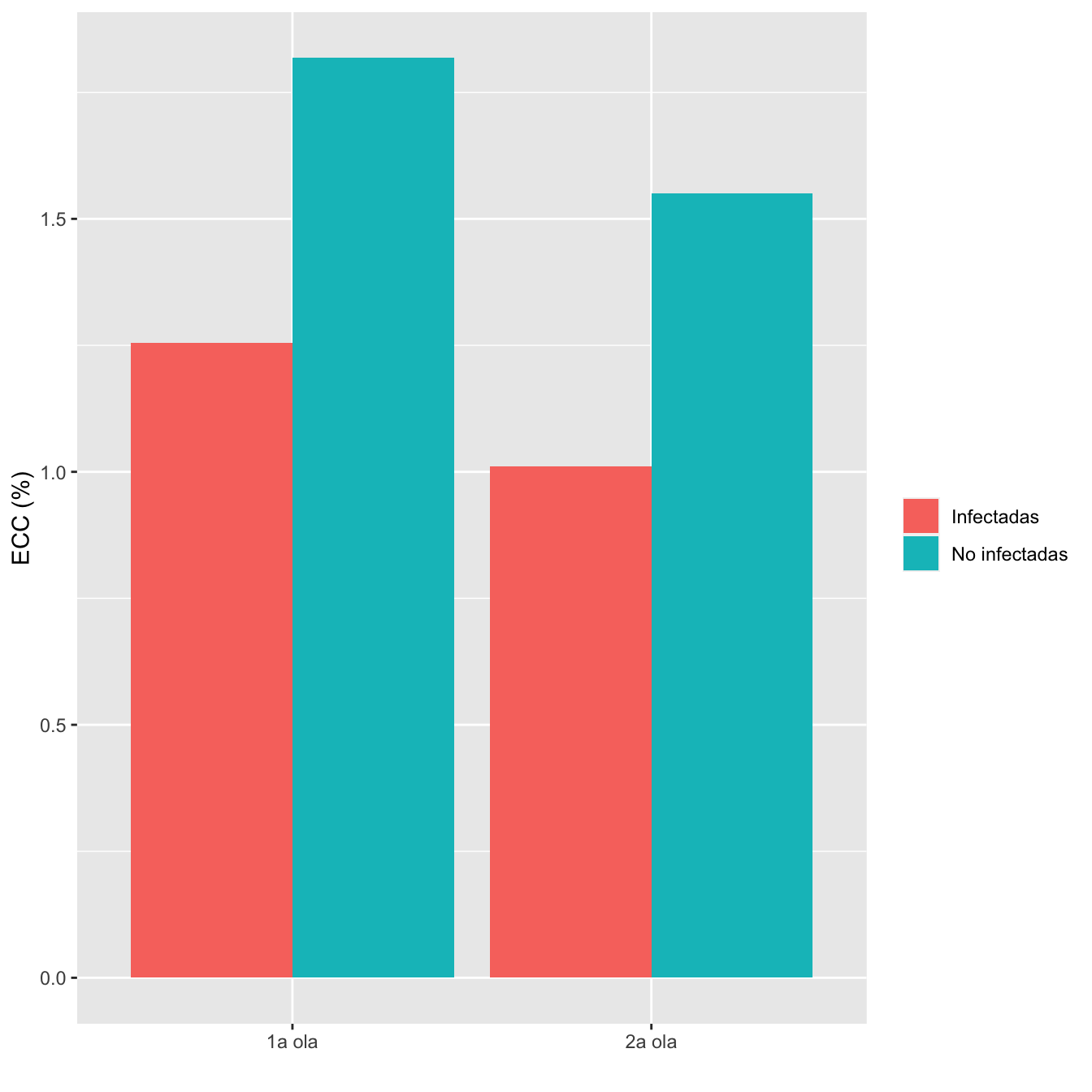
Figura 8.6:
8.2.8 Enfermedades pulmonares crónicas (incluyendo asma)
INA1w=Casos1w$Enfermedad.pulmonar.crónica.no.asma
NINA1w=Controls1w$Enfermedad.pulmonar.crónica.no.asma
NINA1w[NINA1w==0]="No"
NINA1w[NINA1w==1]="Sí"
IA1w=Casos1w$Diagnóstico.clínico.de.Asma
NIA1w=Controls1w$Diagnóstico.clínico.de.Asma
NIA1w[NIA1w==0]="No"
NIA1w[NIA1w==1]="Sí"
I1=rep(NA,n_I1)
for (i in 1:n_I1){I1[i]=max(INA1w[i],IA1w[i],na.rm=TRUE)}
NI1=rep(NA,n_NI1)
for (i in 1:n_NI1){NI1[i]=max(NINA1w[i],NIA1w[i],na.rm=TRUE)}
NI1[NI1==-Inf]=NA
INA2w=Casos2w$Enfermedad.pulmonar.crónica.no.asma
NINA2w=Controls2w$Enfermedad.pulmonar.crónica.no.asma
NINA2w[NINA2w==0]="No"
NINA2w[NINA2w==1]="Sí"
IA2w=Casos2w$Diagnóstico.clínico.de.Asma
NIA2w=Controls2w$Diagnóstico.clínico.de.Asma
NIA2w[NIA2w==0]="No"
NIA2w[NIA2w==1]="Sí"
I2=rep(NA,n_I2)
for (i in 1:n_I2){I2[i]=max(INA2w[i],IA2w[i],na.rm=TRUE)}
NI2=rep(NA,n_NI2)
for (i in 1:n_NI2){NI2[i]=max(NINA2w[i],NIA2w[i],na.rm=TRUE)}
NI2[NI2==-Inf]=NA
#
ComparOlasCC(I1,I2,NI1,NI2,"EPC","No EPC")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EPC | 38 | 4.3 | 42 | 3.6 | 36 | 4.6 | 12 | 3.1 | 0.8021 | 0.3598 | 1.7881 | 0.589774 | 0.852058 | 0.905326 | 0.749704 |
| No EPC | 839 | 95.7 | 1111 | 96.4 | 755 | 95.4 | 376 | 96.9 | |||||||
| Datos perdidos | 0 | 38 | 0 | 28 |
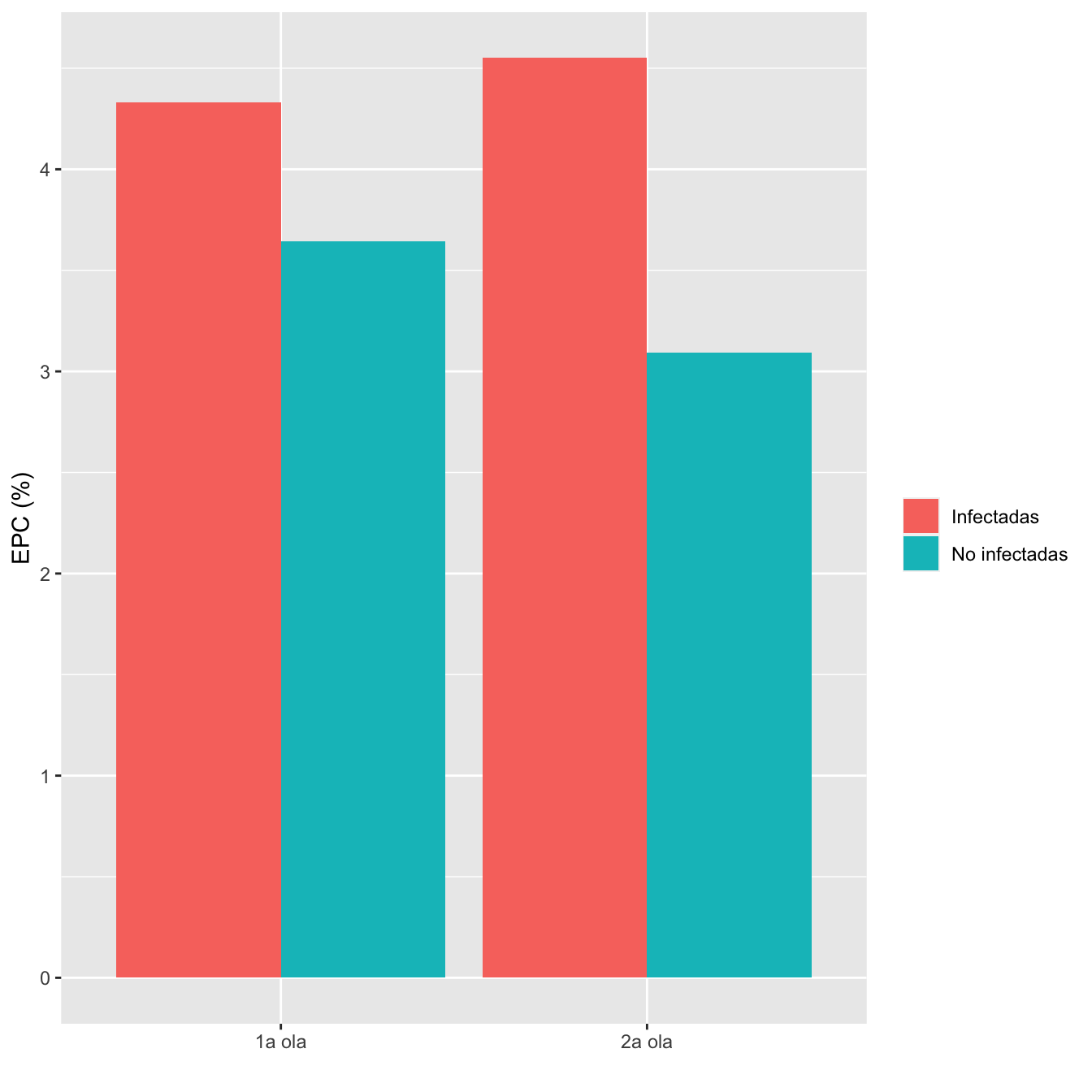
Figura 8.7:
8.2.9 Paridad
I1=Casos1w$NULIPARA
I2=Casos2w$NULIPARA
NI1=Controls1w$Nuliparous
NI2=Controls2w$Nuliparous
EE=cbind(as.vector(table(I1))[2:1],
as.vector(table(NI1))[2:1],
as.vector(table(I2))[2:1],
as.vector(table(NI2))[2:1])
#
ComparOlasCC(I1,I2,NI1,NI2,"Nulípara","Multípara")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Nulípara | 319 | 37 | 451 | 38.1 | 287 | 36.4 | 193 | 46.7 | 1.4599 | 1.0792 | 1.9748 | 0.014109 | 0.204173 | 0.83805 | 0.002438 |
| Multípara | 544 | 63 | 732 | 61.9 | 502 | 63.6 | 220 | 53.3 | |||||||
| Datos perdidos | 14 | 8 | 2 | 3 |
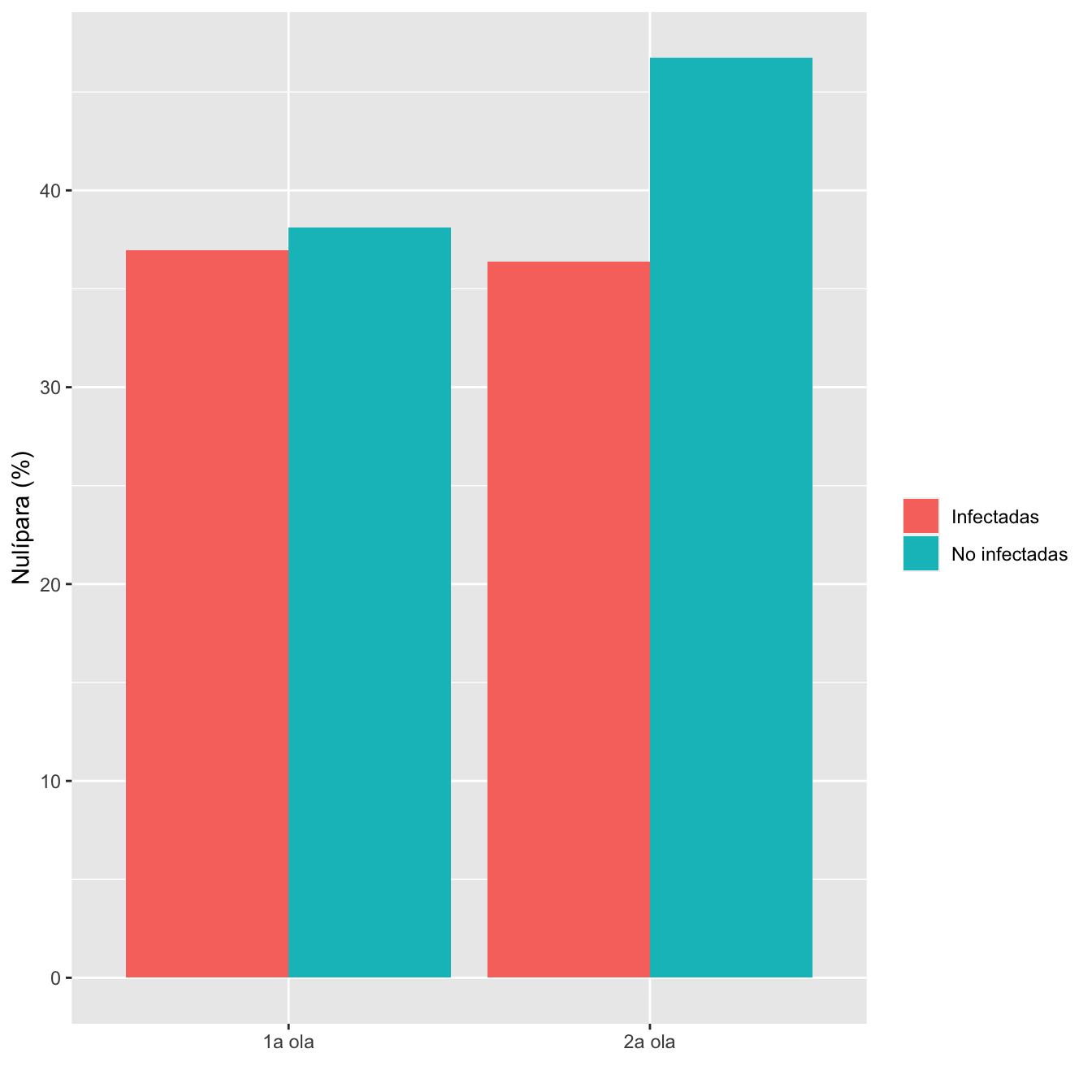
Figura 8.8:
8.2.10 Gestación múltip
I1=Casos1w$Gestación.Múltiple
I2=Casos2w$Gestación.Múltiple
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Gestación.Múltiple
NI2=Controls2w$Gestación.Múltiple
#
ComparOlasCC(I1,I2,NI1,NI2,"Gestación múltiple", "Gestación única")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Cociente de OR | Extr. inf. IC | Extr. sup. IC | p-valor igualdad OR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Gestación múltiple | 18 | 2.1 | 28 | 2.4 | 13 | 1.6 | 6 | 1.4 | 0.7624 | 0.2429 | 2.3931 | 0.642082 | 0.242333 | 0.589242 | 0.325861 |
| Gestación única | 859 | 97.9 | 1163 | 97.6 | 778 | 98.4 | 410 | 98.6 | |||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
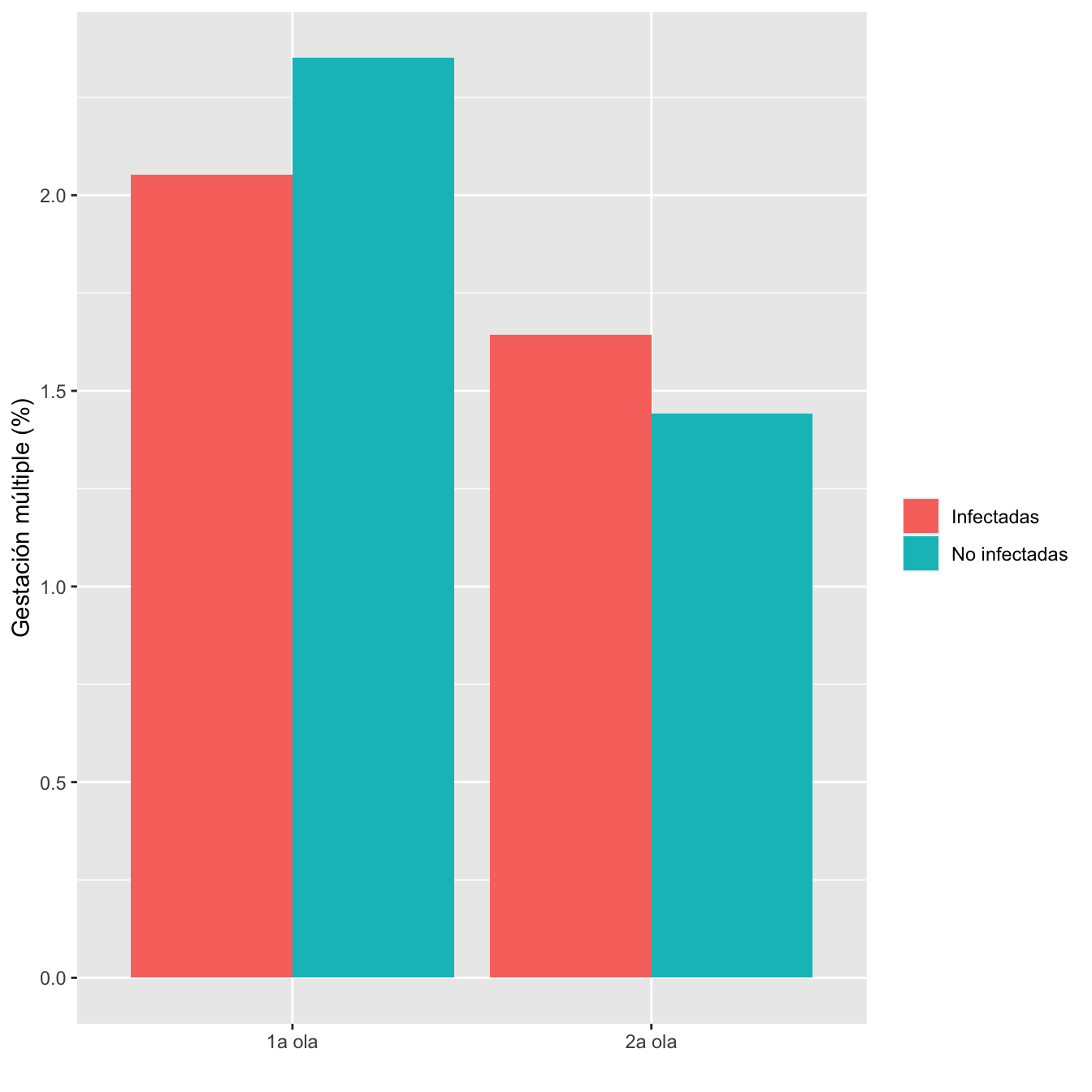
Figura 8.9:
8.3 Comparación de riesgos de desenlaces
8.3.1 Hipertensión gestacional
En tablas como la que sigue:
- Diferencia RA: Diferencia entre los RA del desenlace en la 1a menos la 2a ola
- IC RA: IC 95% para esta diferencia
- p-valor igualdad RA: p-valor del contraste de igualdad entre los RA del desenlace en las dos olas
- Cociente RR: Cociente de los RR del desenlace en la 1a dividido por la 2a ola
- IC RR: IC 95% para este cociente
- p-valor igualdad RR: p-valor del contraste de igualdad entre los RR del desenlace en las dos olas
- p-valor global olas, p-valor casos, p-valor controles: como antes
I1=Casos1w$Hipertensión.gestacional
I2=Casos2w$Hipertensión.gestacional
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Hipertensión.gestacional
NI2=Controls2w$Hipertensión.gestacional
#
ComparOlasRR(I1,I2,NI1,NI2,"HG", "No HG")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HG | 20 | 2.3 | 29 | 2.5 | 22 | 2.8 | 7 | 1.7 | -0.0126 | -0.0338 | 0.0109 | 0.270177 | 0.5672 | 0.206 | 1.5619 | 0.272565 | 1 | 0.53493 | 0.445232 |
| No HG | 857 | 97.7 | 1146 | 97.5 | 769 | 97.2 | 403 | 98.3 | |||||||||||
| Datos perdidos | 0 | 16 | 0 | 6 |
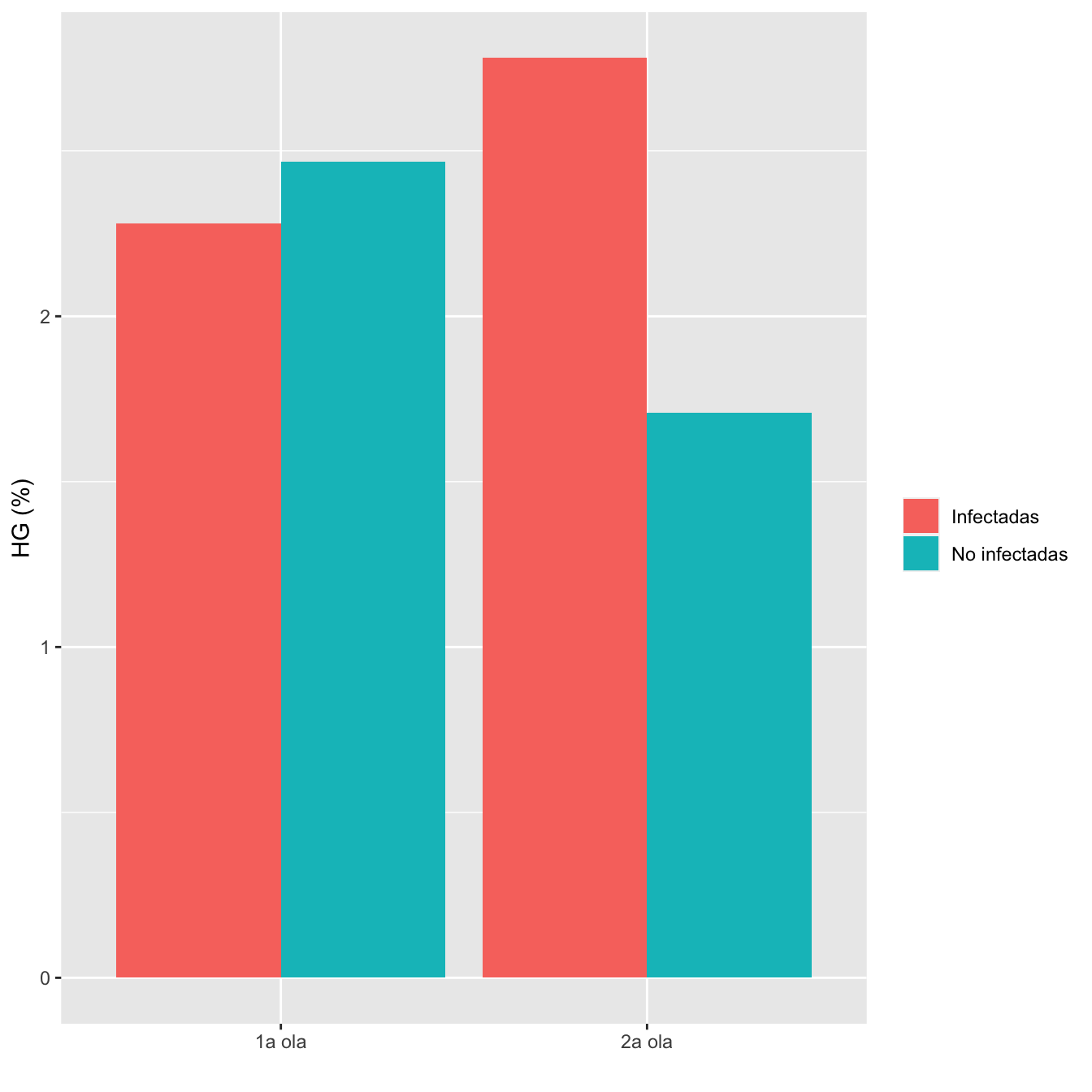
Figura 8.10:
8.3.2 Diabetes gestacional
I1=Casos1w$Diabetes.gestacional
I2=Casos2w$Diabetes.gestacional
I2[I2=="No"]=0
I2[I2=="Sí"]=1
I1[I1=="No"]=0
I1[I1=="Sí"]=1
#
NI1=Controls1w$Diabetes.gestacional
NI2=Controls2w$Diabetes.gestacional
#
ComparOlasRR(I1,I2,NI1,NI2,"DG", "No DG")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DG | 66 | 7.5 | 95 | 8.1 | 59 | 7.5 | 41 | 10 | 0.0197 | -0.0205 | 0.0631 | 0.343527 | 1.2468 | 0.7674 | 2.0258 | 0.372978 | 0.640031 | 1 | 0.25976 |
| No DG | 811 | 92.5 | 1079 | 91.9 | 732 | 92.5 | 369 | 90 | |||||||||||
| Datos perdidos | 0 | 17 | 0 | 6 |
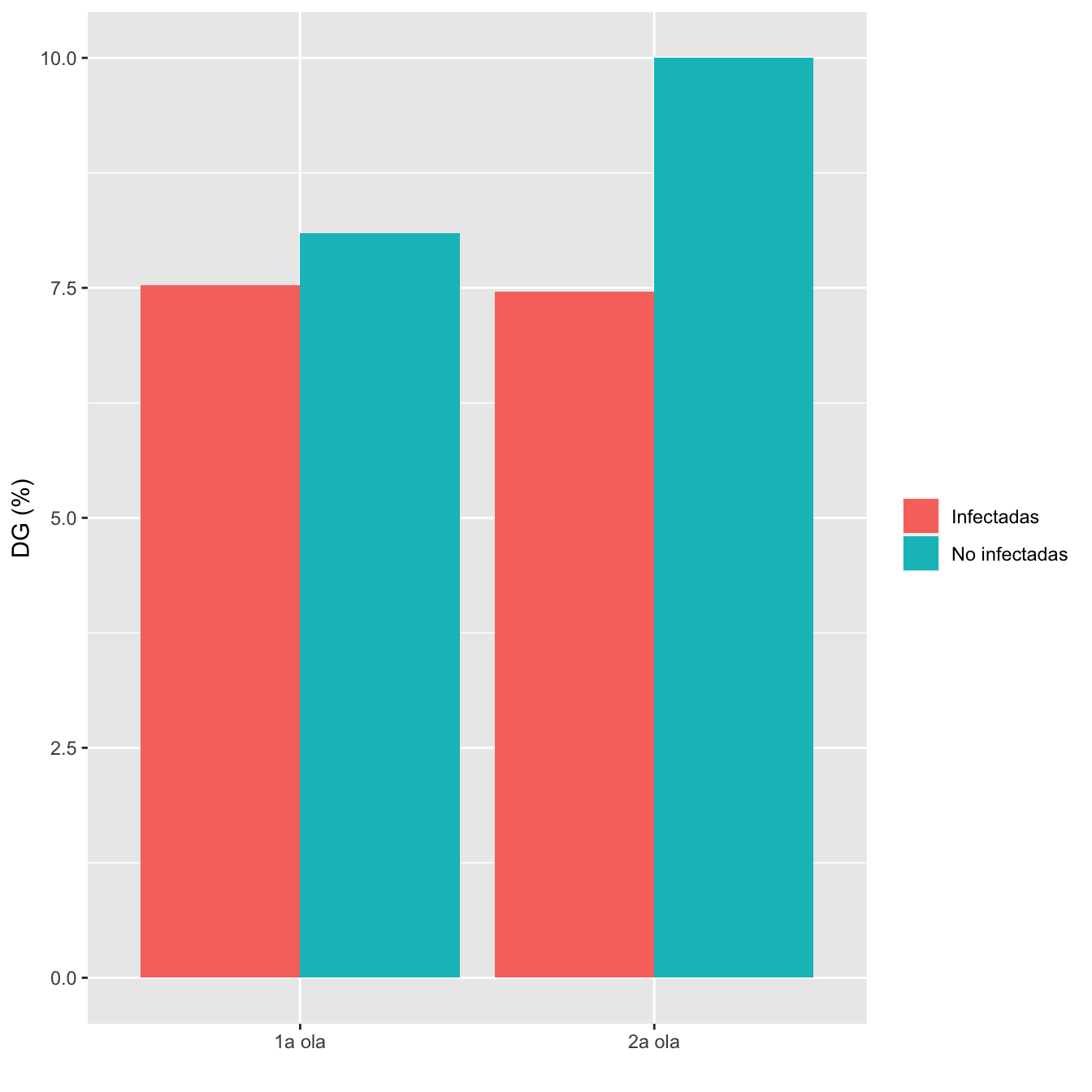
Figura 8.11:
8.3.3 Preeclampsia
I1=Casos1w$PREECLAMPSIA_ECLAMPSIA_TOTAL
I2=Casos2w$PREECLAMPSIA_ECLAMPSIA_TOTAL
NI1=Controls1w$PREECLAMPSIA
NI2=Controls2w$PREECLAMPSIA
#
ComparOlasRR(I1,I2,NI1,NI2,"PE", "No PE")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| PE | 46 | 5.2 | 56 | 4.7 | 40 | 5.1 | 8 | 1.9 | -0.0259 | -0.0535 | 0.003 | 0.077255 | 0.4242 | 0.183 | 0.9833 | 0.045573 | 0.225527 | 0.911918 | 0.012567 |
| No PE | 831 | 94.8 | 1135 | 95.3 | 751 | 94.9 | 408 | 98.1 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
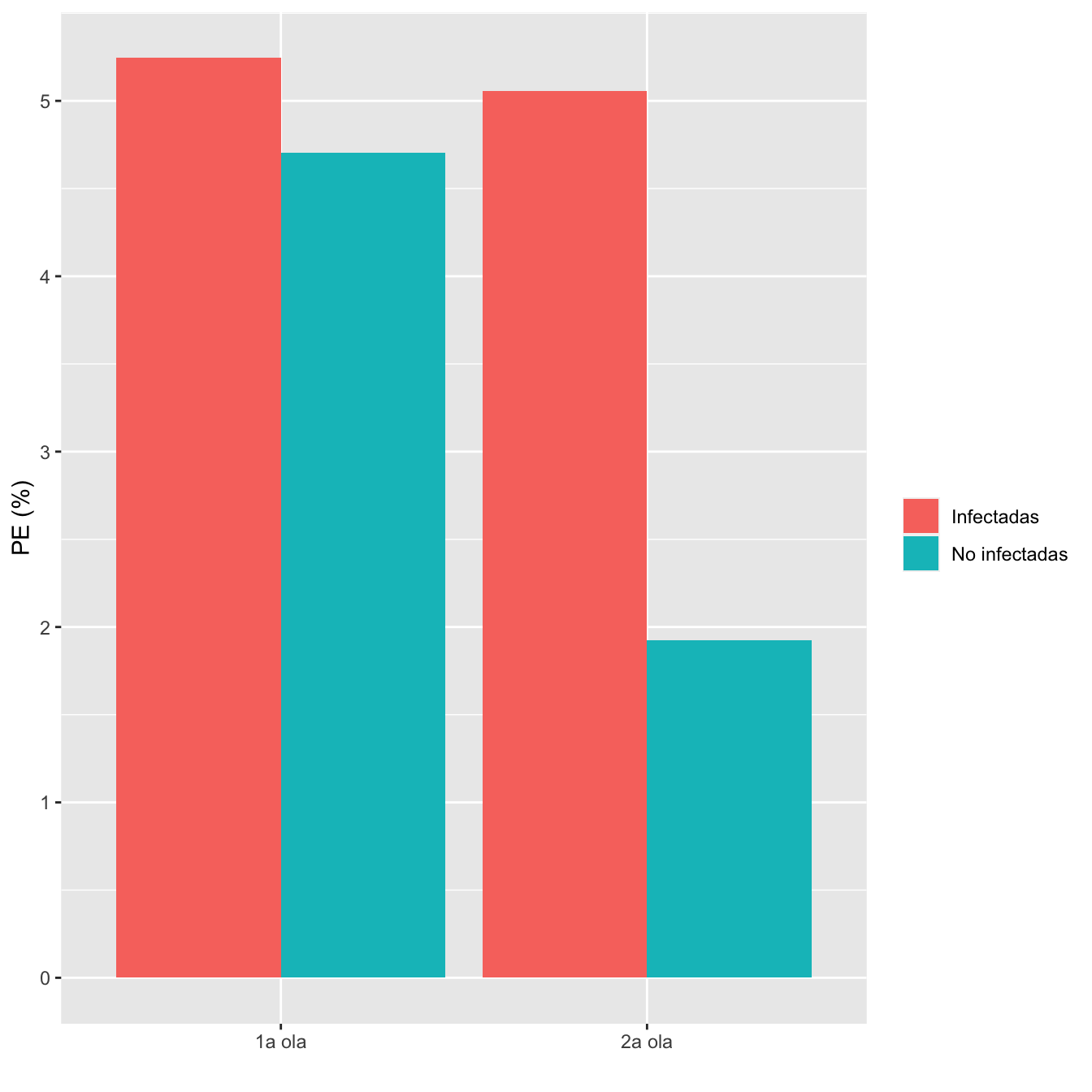
Figura 8.12:
8.3.4 Preeclampsia con CG entre las preeclámpsicas
I1=Casos1w[Casos1w$PREECLAMPSIA_ECLAMPSIA_TOTAL==1,]$Preeclampsia.grave_HELLP_ECLAMPSIA
I1[I1=="Sí"]=1
I1[I1=="No"]=0
I2=Casos2w[Casos2w$PREECLAMPSIA_ECLAMPSIA_TOTAL==1,]$Preeclampsia.grave_HELLP_ECLAMPSIA
I2[I2=="Sí"]=1
I2[I2=="No"]=0
NI1.1=Controls1w[Controls1w$PREECLAMPSIA==1,]$preeclampsia_severa
NI1.2=Controls1w[Controls1w$PREECLAMPSIA==1,]$Preeclampsia.grave.No.HELLP
NI1=rep(NA,length(NI1.1))
for (i in 1:length(NI1.1)){NI1[i]=max(NI1.1[i],NI1.2[i],na.rm=TRUE)}
NI2.1=Controls2w[Controls2w$PREECLAMPSIA==1,]$preeclampsia_severa
NI2.2=Controls2w[Controls2w$PREECLAMPSIA==1,]$Preeclampsia.grave.No.HELLP
NI2=rep(NA,length(NI2.1))
for (i in 1:length(NI2.1)){NI2[i]=max(NI2.1[i],NI2.2[i],na.rm=TRUE)}
#
ComparOlasRR(I1,I2,NI1,NI2,"PE con CG", "PE sin CG")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| PE con CG | 19 | 41.3 | 8 | 14.3 | 11 | 27.5 | 3 | 37.5 | 0.3702 | 0.0243 | 0.7629 | 0.033753 | 3.9427 | 1.1201 | 13.8784 | 0.032634 | 0.844514 | 0.256782 | 0.131402 |
| PE sin CG | 27 | 58.7 | 48 | 85.7 | 29 | 72.5 | 5 | 62.5 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
base=c("1a ola","2a ola")
df =data.frame(
Factor=c(rep("Infectadas 1a ola",length(I1)), rep("No infectadas 1a ola",length(NI1)),rep("Infectadas 2a ola",length(I2)), rep("No infectadas 2a ola",length(NI2))),
Grupo=c(I1,NI1,I2,NI2)
)
Grupo=ordered(rep(c("Infectadas","No infectadas"), 2),levels=c("Infectadas","No infectadas"))
Ola=ordered(rep(base , each=2),levels=base)
valor=as.vector(100*prop.table(table(df)[c(1,3,2,4),2:1], margin=1))[1:4]
data=data.frame(Grupo,Ola,valor)
data %>%
ggplot(aes(fill=Grupo, y=valor, x=Ola)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("Preeclampsias con CG (% de preeclampsias)")+
scale_fill_hue(c=100)+labs(fill="")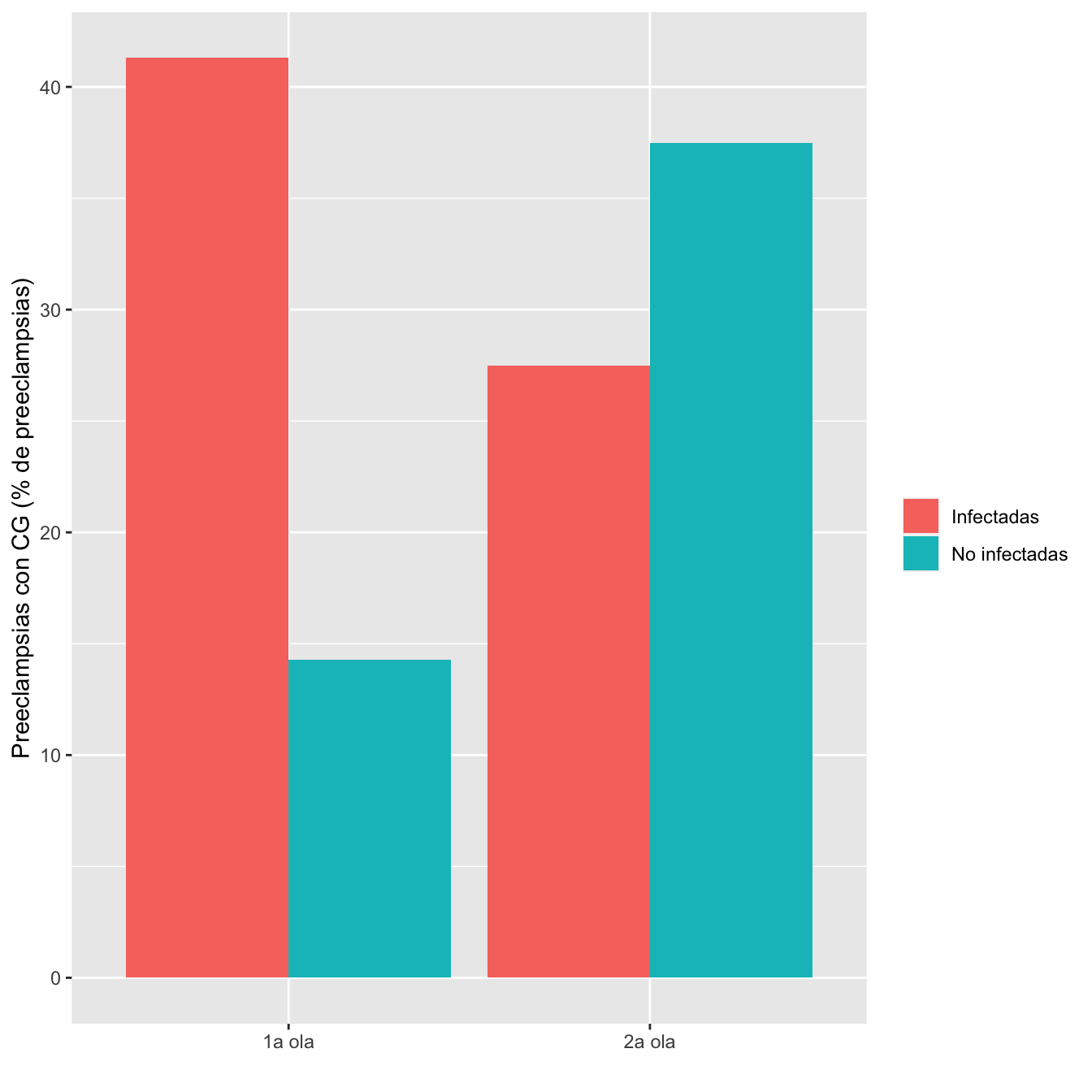
Figura 8.13:
8.3.5 Prematurez
I1=Casos1w$PREMATURO
I2=Casos2w$PREMATURO
NI1=Controls1w$Preterm.deliveries
NI2=Controls2w$Preterm.deliveries
#
ComparOlasRR(I1,I2,NI1,NI2,"Prematuro", "A término")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Prematuro | 104 | 11.9 | 75 | 6.3 | 65 | 8.2 | 19 | 4.6 | 0.0191 | -0.0178 | 0.0581 | 0.31395 | 1.0467 | 0.5904 | 1.8554 | 0.875918 | 0.095521 | 0.014738 | 0.225321 |
| A término | 773 | 88.1 | 1116 | 93.7 | 726 | 91.8 | 397 | 95.4 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
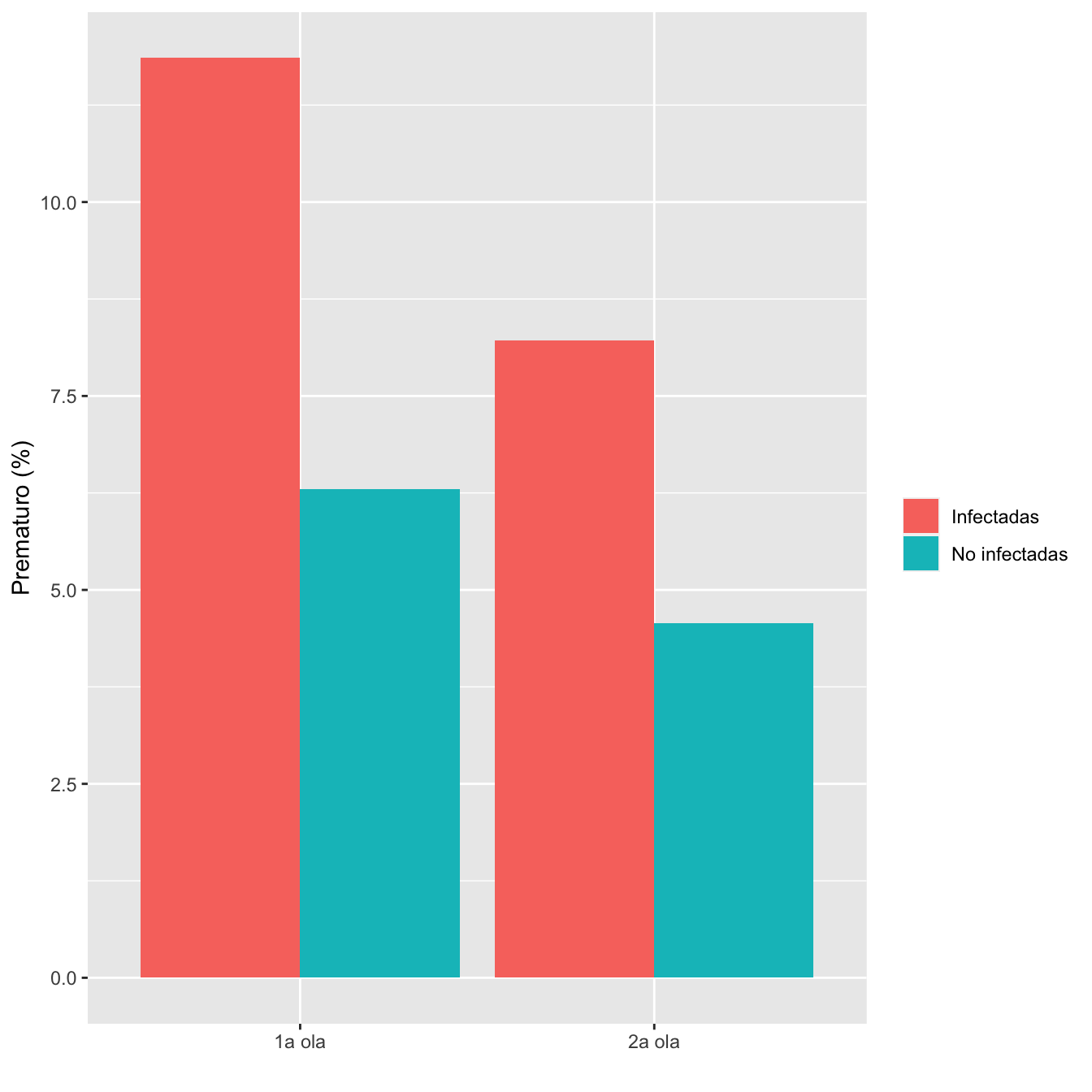
Figura 8.14:
8.3.6 Eventos hemorrágicos
I1=Casos1w$EVENTOS_HEMORRAGICOS_TOTAL
I2=Casos2w$EVENTOS_HEMORRAGICOS_TOTAL
NI1=Controls1w$eventos_hemorragicos
NI2=Controls2w$eventos_hemorragicos
#
ComparOlasRR(I1,I2,NI1,NI2,"Eventos hemorrágicos", "No eventos hemorrágicos")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos hemorrágicos | 51 | 5.8 | 64 | 5.4 | 40 | 5.1 | 25 | 6 | 0.0139 | -0.0189 | 0.05 | 0.413901 | 1.2861 | 0.7039 | 2.3499 | 0.41332 | 0.873878 | 0.518663 | 0.619647 |
| No eventos hemorrágicos | 826 | 94.2 | 1127 | 94.6 | 751 | 94.9 | 391 | 94 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
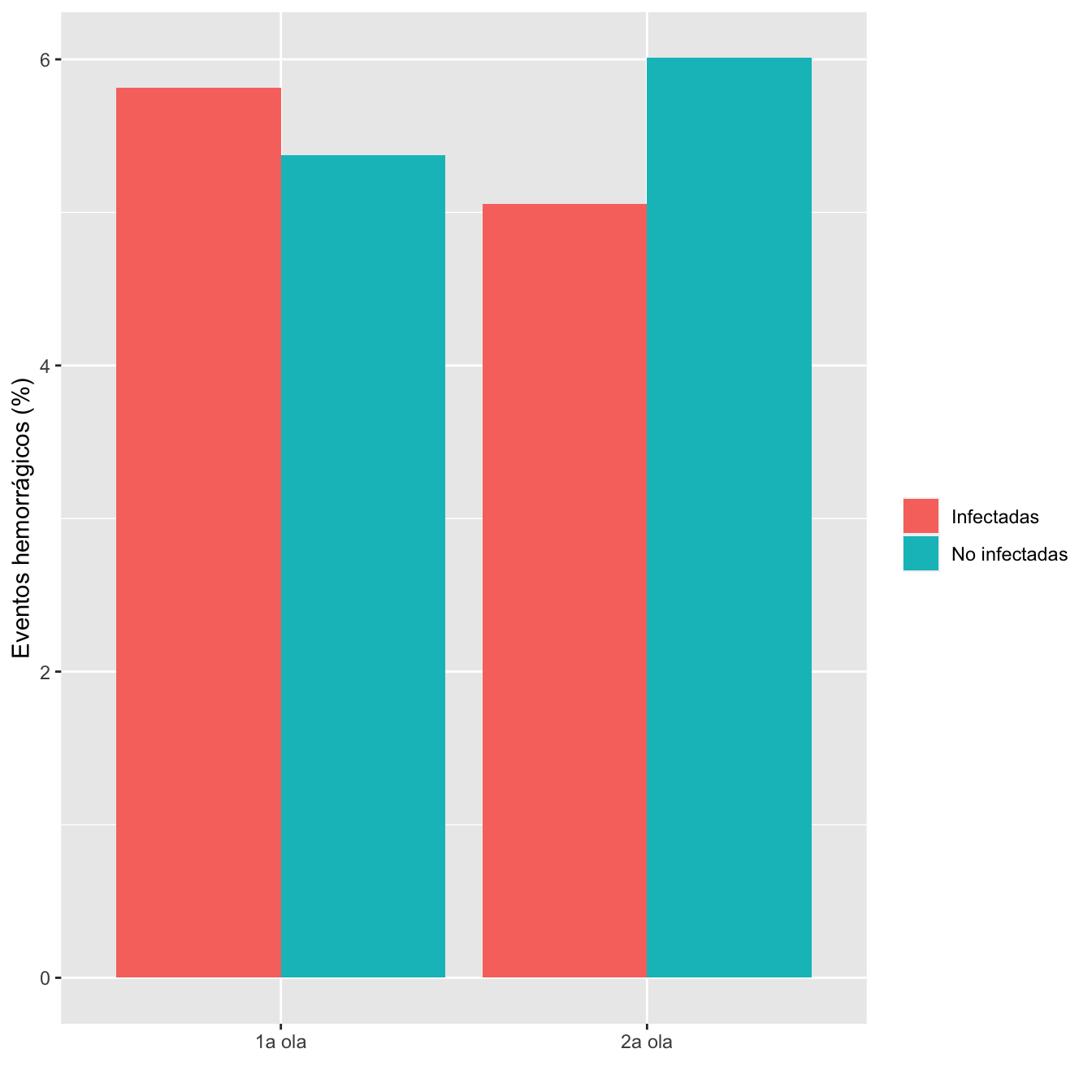
Figura 8.15:
8.3.7 Eventos trombóticos
I1=factor(Casos1w$EVENTOS_TROMBO_TOTALES,levels=0:1)
I2=factor(Casos2w$EVENTOS_TROMBO_TOTALES,levels=0:1)
NI1a=Controls1w$DVT
NI1b=Controls1w$PE
NI1=rep(NA,length(NI1a))
for (i in 1:length(NI1a)){NI1[i]=max(NI1a[i],NI1b[i],na.rm=TRUE)}
NI1=factor(NI1,levels=0:1)
NI2a=Controls2w$DVT
NI2b=Controls2w$PE
NI2=rep(NA,length(NI2a))
for (i in 1:length(NI2a)){NI2[i]=max(NI2a[i],NI2b[i],na.rm=TRUE)}
NI2=factor(NI2,levels=0:1)
#
ComparOlasRR(I1,I2,NI1,NI2,"Eventos trombóticos", "No eventos trombóticos")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eventos trombóticos | 7 | 0.8 | 0 | 0 | 5 | 0.6 | 2 | 0.5 | 0.0065 | -0.0038 | 0.0205 | 0.202718 | Inf | Inf | 0.405389 | 0.777207 | 0.066893 | ||
| No eventos trombóticos | 870 | 99.2 | 1191 | 100 | 786 | 99.4 | 414 | 99.5 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
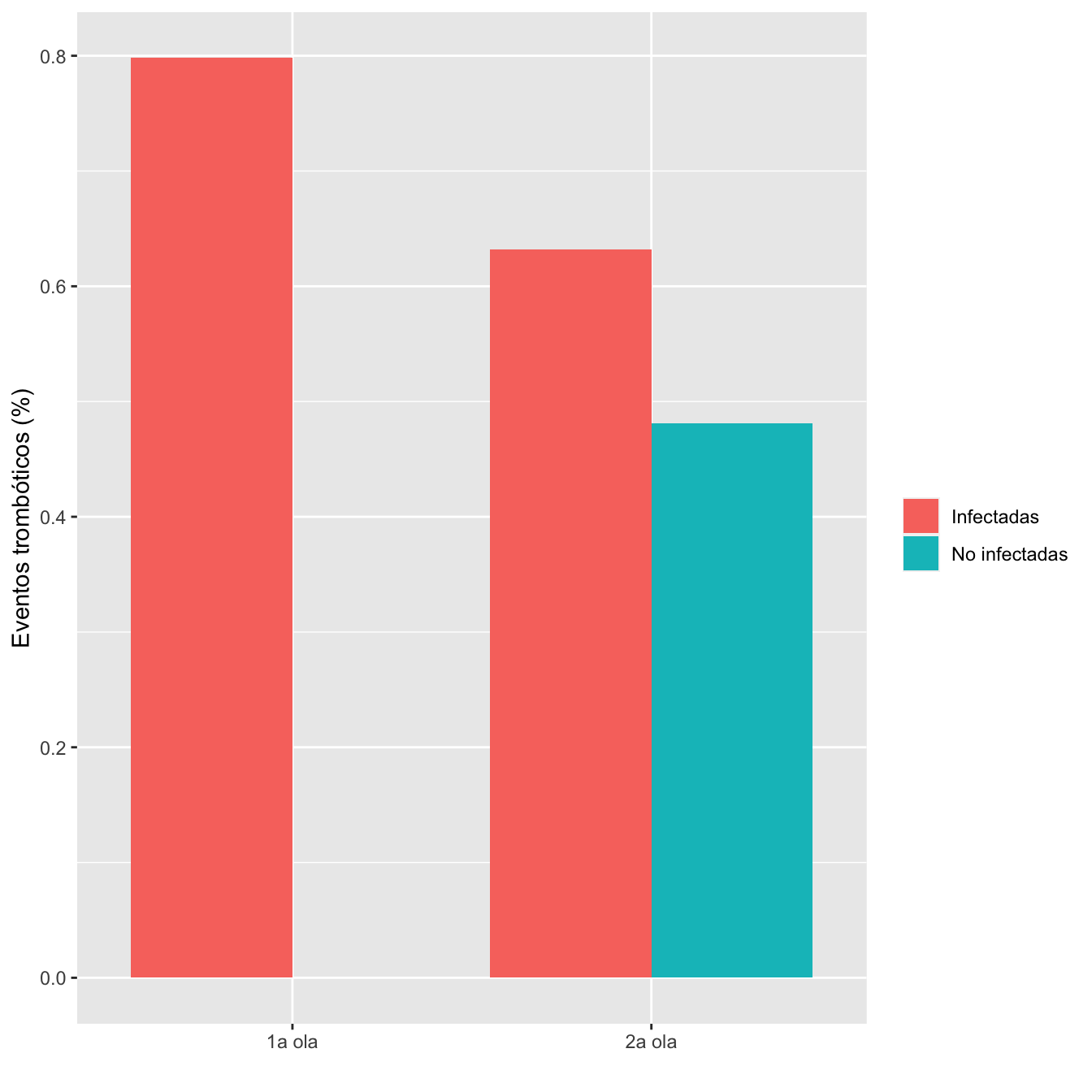
Figura 8.16:
8.3.8 Anomalías congénitas
I1=Casos1w$Diagnóstico.de.malformación.ecográfica._.semana.20._
I1[I1=="No"]=0
I1[I1=="Sí"]=1
I2=Casos2w$Diagnóstico.de.malformación.ecográfica._.semana.20._
I2[I2=="No"]=0
I2[I2=="Sí"]=1
NI1=Controls1w$Diagnóstico.de.malformación.ecográfica._.semana.20._
NI2=Controls2w$Diagnóstico.de.malformación.ecográfica._.semana.20._
#
ComparOlasRR(I1,I2,NI1,NI2,"Anomalías congénitas", "No anomalías congénitas")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Anomalías congénitas | 14 | 1.6 | 12 | 1 | 14 | 1.8 | 4 | 1 | -0.0022 | -0.0191 | 0.017 | 0.798298 | 0.8645 | 0.2254 | 3.3156 | 0.831884 | 0.636883 | 0.849622 | 1 |
| No anomalías congénitas | 835 | 98.4 | 1161 | 99 | 750 | 98.2 | 403 | 99 | |||||||||||
| Datos perdidos | 28 | 18 | 27 | 9 |
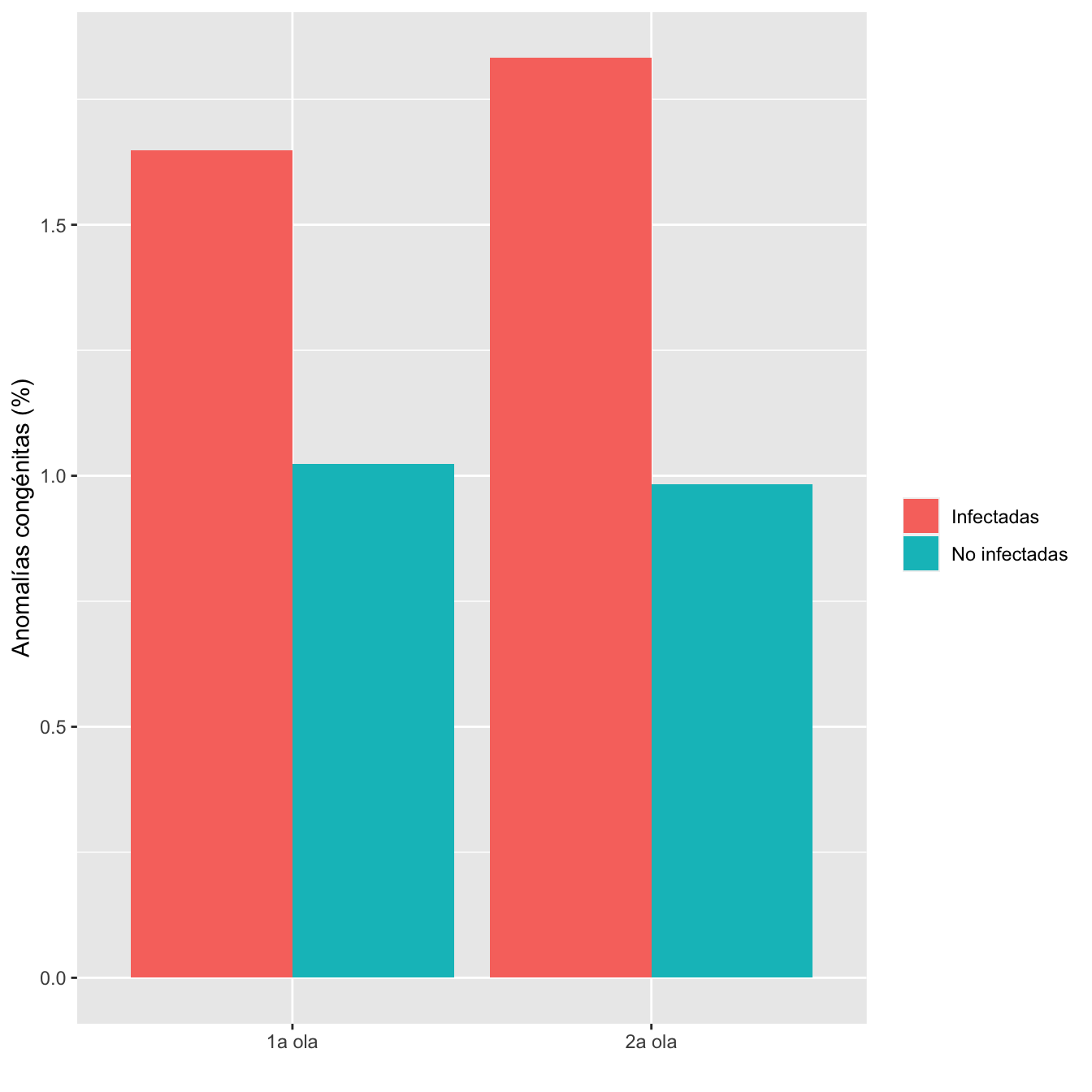
Figura 8.17:
8.3.9 Embarazos con feto muerto anteparto
I1=Casos1w$Feto.muerto.intraútero
I1[I1=="Sí"]=1
I1[I1=="No"]=0
I2=Casos2w$Feto.muerto.intraútero
I2[I2=="Sí"]=1
I2[I2=="No"]=0
NI1a=Controls1w$Feto.vivo...194
NI1a[NI1a=="Sí"]=0
NI1a[NI1a=="No"]=1
NI1b=Controls1w$Feto.vivo...245
NI1b[NI1b=="Sí"]=0
NI1b[NI1b=="No"]=1
NI1=NI1a
for (i in 1:length(NI1a)){NI1[i]=max(NI1a[i],NI1b[i],na.rm=TRUE)}
NI2a=Controls2w$Feto.vivo...194
NI2a[NI2a=="Sí"]=0
NI2a[NI2a=="No"]=1
NI2b=Controls2w$Feto.vivo...245
NI2b[NI2b=="Sí"]=0
NI2b[NI2b=="No"]=1
NI2=NI2a
for (i in 1:length(NI2a)){NI2[i]=max(NI2a[i],NI2b[i],na.rm=TRUE)}
NI2=factor(NI2,levels=0:1)
#
ComparOlasRR(I1,I2,NI1,NI2,"Algún feto muerto anteparto", "Ningún feto muerto anteparto")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Algún feto muerto anteparto | 9 | 1 | 3 | 0.3 | 10 | 1.3 | 0 | 0 | -0.0049 | -0.0169 | 0.0061 | 0.364361 | 0 | 0 | 0 | 0.653241 | 0.572991 | ||
| Ningún feto muerto anteparto | 868 | 99 | 1188 | 99.7 | 781 | 98.7 | 416 | 100 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
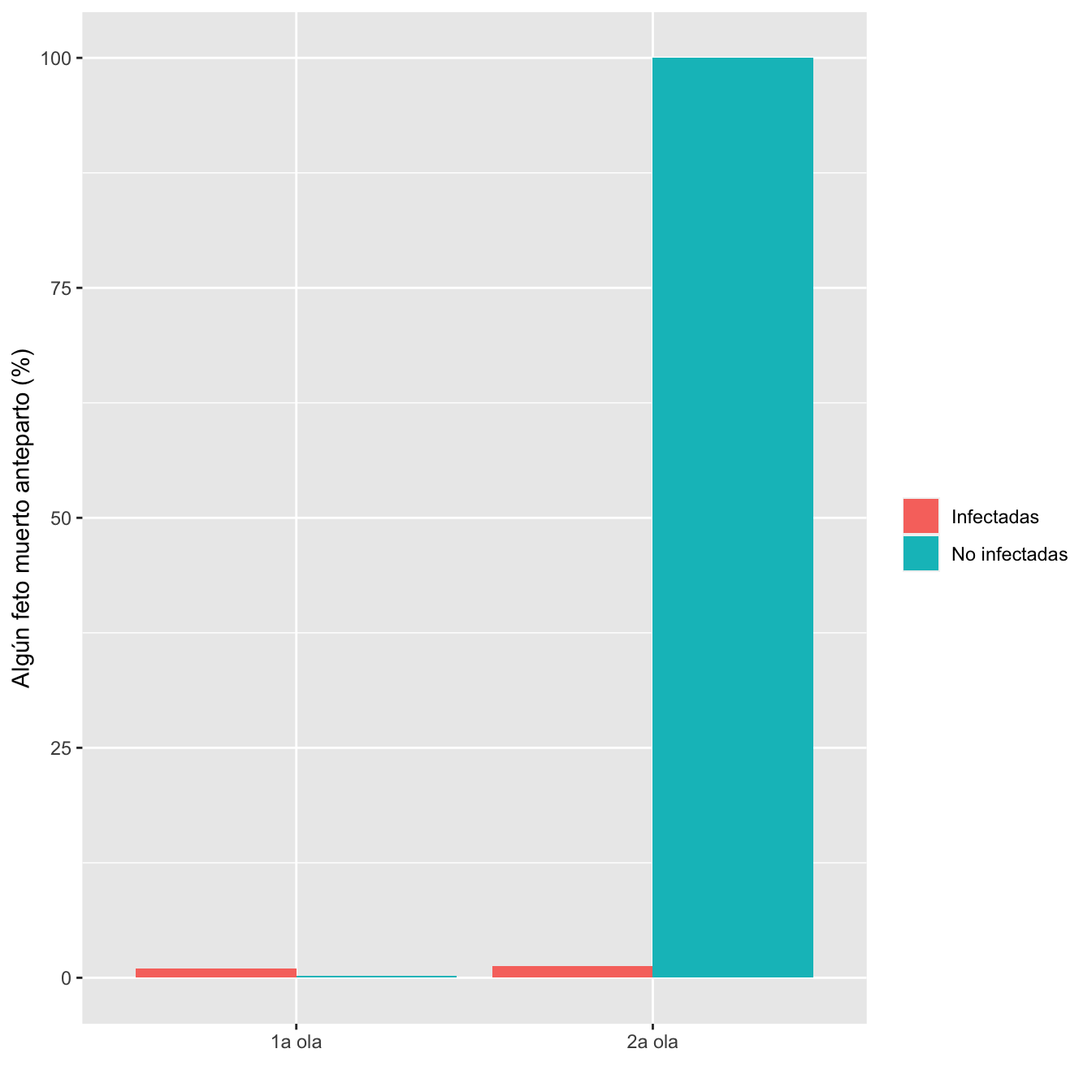
Figura 8.18:
8.3.10 Inicios de parto
En tablas como la que sigue:
- Lo mismo que en las anteriores, pero por filas: cada fila considerada com un desenlace contra el resto
- Los p-valores han sido ajustados por Bonferroni
I1=Casos1w$Inicio.de.parto
I1=factor(I1,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
I2=Casos2w$Inicio.de.parto
I2=factor(I2,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
NI1=Controls1w$Inicio.de.parto
NI1[NI1=="Cesárea programada"]="Cesárea"
NI1=factor(NI1,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
NI2=Controls2w$Inicio.de.parto
NI2[NI2=="Cesárea programada"]="Cesárea"
NI2=factor(NI2,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
ComparOlasRR.m(I1,I2,NI1,NI2,c("Espontáneo", "Inducido", "Cesárea programada"))| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Espontáneo | 442 | 50.5 | 734 | 61.6 | 429 | 54.2 | 266 | 63.9 | -0.01 | -0.09 | 0.06 | 1.0000 | 0.97 | 0.85 | 1.09 | 1.00000 | 1.000000 | 0.421874 | 1 |
| Inducido | 325 | 37.1 | 393 | 33.0 | 303 | 38.3 | 129 | 31.0 | -0.03 | -0.10 | 0.04 | 1.0000 | 0.91 | 0.74 | 1.12 | 1.00000 | 1.000000 | 1.000000 | 1 |
| Cesárea programada | 108 | 12.3 | 64 | 5.4 | 59 | 7.5 | 21 | 5.0 | 0.05 | 0.01 | 0.08 | 0.0441 | 1.55 | 0.88 | 2.74 | 0.38254 | 0.267502 | 0.003122 | 1 |
| Datos perdidos | 2 | 0 | 0 | 0 |
Tipo= c(I1,I2,NI1,NI2)
Tipo[Tipo=="Cesárea"]="Cesárea programada"
Tipo=ordered(Tipo, levels=c("Espontáneo", "Inducido", "Cesárea programada"))
dtbp=data.frame(Grupo=c(rep("Inf.",length(I1)+length(I2)),rep("No inf.",length(NI1)+length(NI2))),
Ola= c(rep("1a ola",length(I1)),rep("2a ola",length(I2)),rep("1a ola",length(NI1)),rep("2a ola",length(NI2))),
Tipo
)
ggplot(data=na.omit(dtbp))+
geom_mosaic(aes(x=product(Tipo,Ola), fill=Grupo)) +
theme_mosaic()+
ylab("Inicio de parto")+
xlab("")+
scale_fill_discrete(name ="", labels=c("Infectadas", "No infectadas"))+
scale_x_productlist(labels=c("Infectadas\n 1a ola", "No infectadas\n 1a ola", "Infectadas\n 2a ola", "No infectadas\n 2a ola"))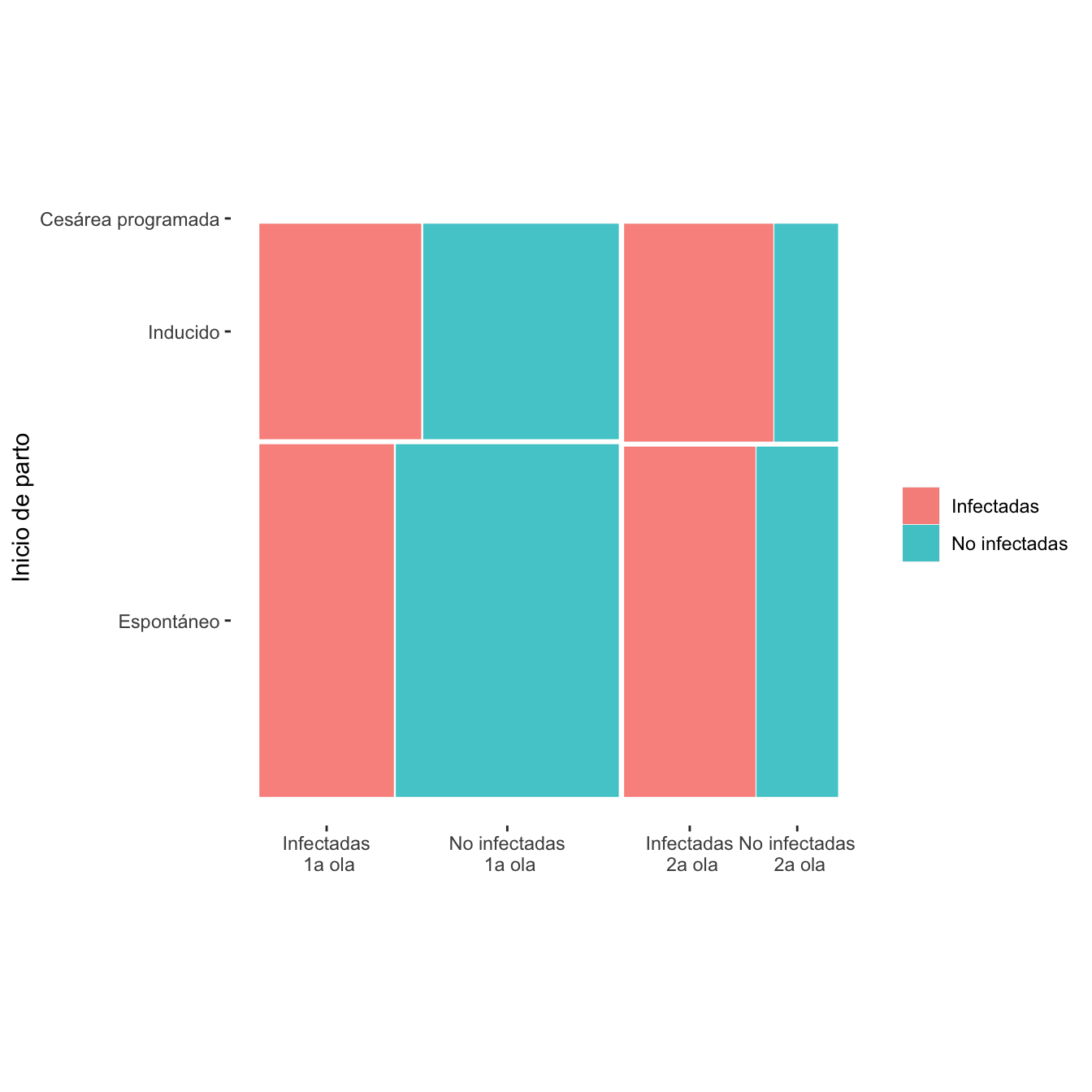
Figura 8.19:
- Test de interacción de Scheirer-Ray-Hare: p-valor, 0.745. Un p-valor pequeño hubiera sido evidencia estadística de que la diferencia de la “gravedad mediana” de los inicios de parto en infectadas y no infectadas en las dos olas han sido diferentes. No ha sido el caso.
8.3.11 Tipos de parto
I1.In=Casos1w$Inicio.de.parto
I1=Casos1w$Tipo.de.parto
I1[I1.In=="Cesárea"]="Cesárea programada"
I1[I1=="Cesárea"]="Cesárea urgente"
I1[I1=="Eutocico"]="Eutócico"
I1=factor(I1,levels=c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
I2.In=Casos2w$Inicio.de.parto
I2=Casos2w$Tipo.de.parto
I2[I2.In=="Cesárea"]="Cesárea programada"
I2[I2=="Cesárea"]="Cesárea urgente"
I2[I2=="Eutocico"]="Eutócico"
I2=factor(I2,levels=c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
NI1.In=Controls1w$Inicio.de.parto
NI1=Controls1w$Tipo.de.parto
NI1[NI1.In=="Cesárea programada"]="Cesárea programada"
NI1[NI1=="Cesárea"]="Cesárea urgente"
NI1[NI1=="Eutocico"]="Eutócico"
NI1=factor(NI1,levels=c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
NI2.In=Controls2w$Inicio.de.parto
NI2=Controls2w$Tipo.de.parto
NI2[NI2.In=="Cesárea programada"]="Cesárea programada"
NI2[NI2=="Cesárea"]="Cesárea urgente"
NI2[NI2=="Eutocico"]="Eutócico"
NI2=factor(NI2,levels=c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
ComparOlasRR.m(I1,I2,NI1,NI2,c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"))| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Eutócico | 528 | 60.2 | 767 | 64.4 | 527 | 66.6 | 277 | 66.6 | -0.04 | -0.11 | 0.03 | 0.940115 | 0.93 | 0.84 | 1.04 | 0.877288 | 0.093918 | 0.028144 | 1 |
| Instrumental | 100 | 11.4 | 177 | 14.9 | 77 | 9.7 | 58 | 13.9 | 0.01 | -0.04 | 0.06 | 1.000000 | 1.10 | 0.74 | 1.63 | 1.000000 | 0.285699 | 1.000000 | 1 |
| Cesárea programada | 108 | 12.3 | 64 | 5.4 | 59 | 7.5 | 21 | 5.0 | 0.05 | 0.01 | 0.08 | 0.061065 | 1.55 | 0.88 | 2.74 | 0.518006 | 0.356675 | 0.004188 | 1 |
| Cesárea urgente | 141 | 16.1 | 183 | 15.4 | 128 | 16.2 | 60 | 14.4 | -0.01 | -0.06 | 0.04 | 1.000000 | 0.93 | 0.66 | 1.32 | 1.000000 | 1.000000 | 1.000000 | 1 |
| Datos perdidos | 0 | 0 | 0 | 0 |
8.3.12 Retraso del crecimiento
I1=factor(Casos1w$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.,labels=0:1)
I2=factor(Casos2w$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.,labels=0:1)
NI1=factor(Controls1w$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.)
NI2=factor(Controls2w$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.)
ComparOlasRR(I1,I2,NI1,NI2,"RCIU", "No RCIU")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| RCIU | 32 | 3.6 | 33 | 2.9 | 27 | 3.4 | 11 | 2.7 | 6e-04 | -0.0241 | 0.0282 | 0.960944 | 1.0047 | 0.4336 | 2.3277 | 0.991361 | 1 | 0.894551 | 1 |
| No RCIU | 845 | 96.4 | 1123 | 97.1 | 764 | 96.6 | 399 | 97.3 | |||||||||||
| Datos perdidos | 0 | 35 | 0 | 6 |
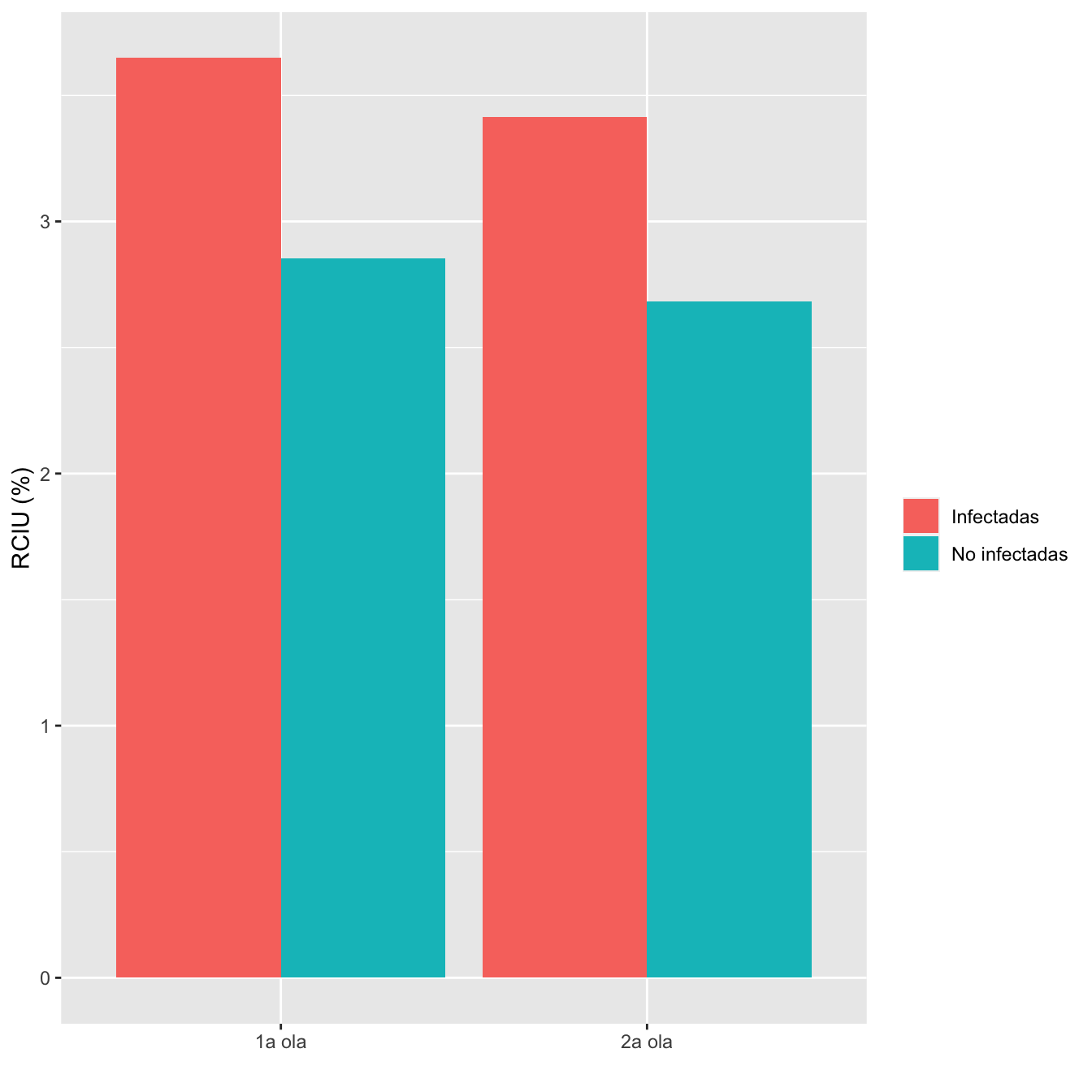
Figura 8.20:
8.3.13 Hemorragia postparto
I1=Casos1w$Hemorragia.postparto
I1.sino=I1
I1.sino[!is.na(I1.sino)& I1.sino!="No"]="Sí"
I1=ordered(I1,levels=names(table(I1))[c(2,3,1,4)])
I2=Casos2w$Hemorragia.postparto
I2.sino=I2
I2.sino[!is.na(I2.sino)& I2.sino!="No"]="Sí"
I2=ordered(I2,levels=names(table(I2))[c(2,3,1,4)])
NI1=Controls1w$Hemorragia.postparto
NI1.sino=NI1
NI1.sino[!is.na(NI1.sino)& NI1.sino!="No"]="Sí"
NI1=ordered(NI1,levels=names(table(NI1))[c(2,3,1,4)])
NI2=Controls2w$Hemorragia.postparto
NI2.sino=NI2
NI2.sino[!is.na(NI2.sino)& NI2.sino!="No"]="Sí"
NI2=ordered(NI2,levels=names(table(NI2))[c(2,3,1,4)])
ComparOlasRR(I1.sino,I2.sino,NI1.sino,NI2.sino,"HPP", "No HPP")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HPP | 41 | 4.8 | 50 | 4.3 | 32 | 4.1 | 20 | 4.9 | 0.0127 | -0.0172 | 0.0459 | 0.412874 | 1.3272 | 0.6732 | 2.6162 | 0.413699 | 0.929475 | 0.549725 | 0.675903 |
| No HPP | 814 | 95.2 | 1121 | 95.7 | 747 | 95.9 | 392 | 95.1 | |||||||||||
| Datos perdidos | 22 | 20 | 12 | 4 |
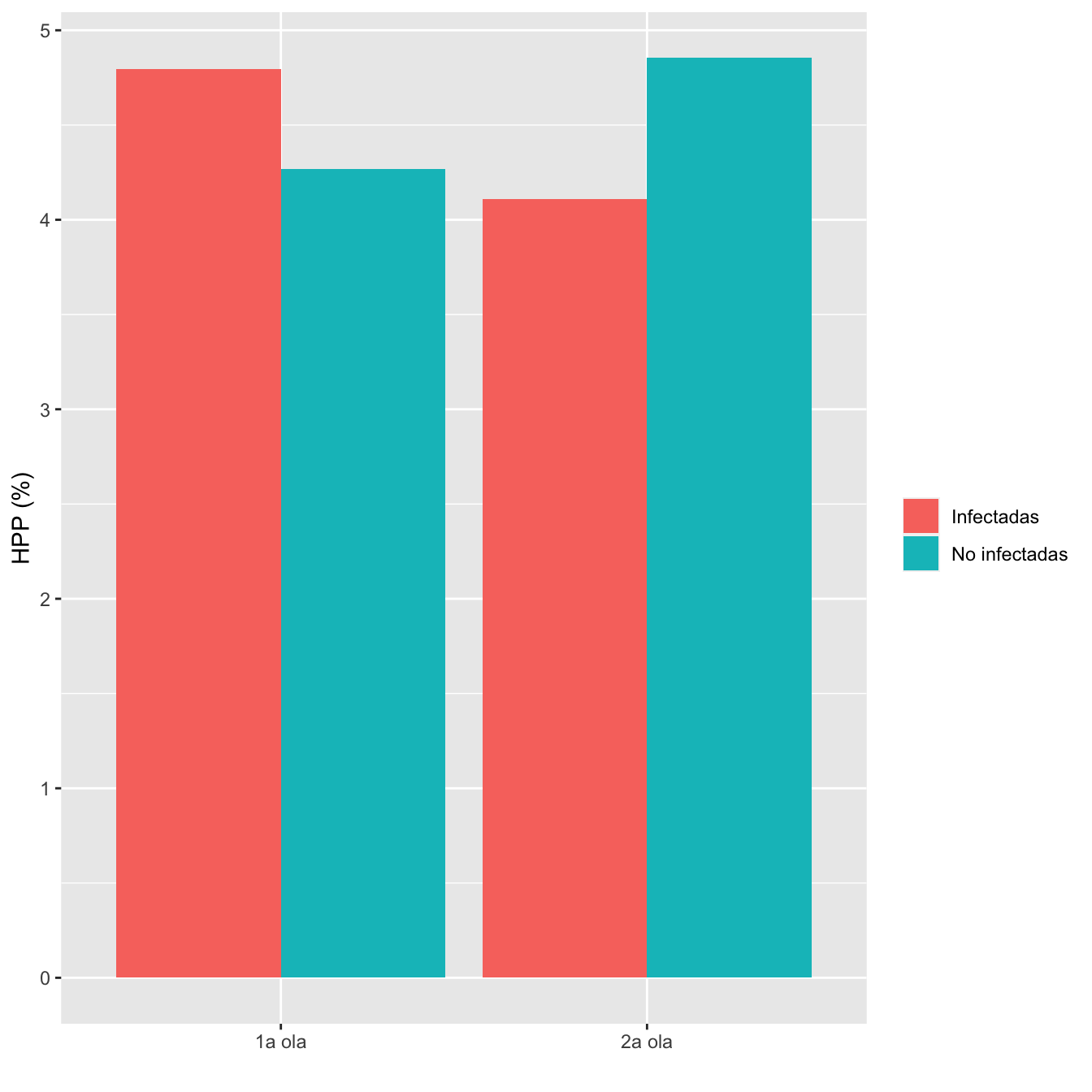
Figura 8.21:
ComparOlasRR.m(I1,I2,NI1,NI2,c( "HPP Tto médico", "HPP Tto Qx conservador", "Histerectomía", "No HPP"),r=6)| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| HPP Tto médico | 32 | 3.7 | 42 | 3.6 | 5 | 0.6 | 3 | 0.7 | 0.00 | -0.02 | 0.02 | 1 | 1.18 | 0.26 | 5.28 | 1.000000 | 0 | 6.0e-05 | 0.006249 |
| HPP Tto Qx conservador | 4 | 0.5 | 7 | 0.6 | 747 | 95.9 | 392 | 95.1 | -0.01 | -0.04 | 0.02 | 1 | 0.78 | 0.23 | 2.64 | 1.000000 | 0 | 0.0e+00 | 0.000000 |
| Histerectomía | 5 | 0.6 | 1 | 0.1 | 27 | 3.5 | 17 | 4.1 | 0.01 | -0.01 | 0.04 | 1 | 8.15 | 0.88 | 75.47 | 0.259221 | 0 | 8.3e-05 | 0.000000 |
| No HPP | 814 | 95.2 | 1121 | 95.7 | 12 | 4 | 0.00 | -0.03 | 0.02 | 1 | 1.13 | 0.27 | 4.69 | 1.000000 | 0 | 0.0e+00 | 0.000000 | ||
| Datos perdidos | 22 | 20 | 5 | 0.6 | 3 | 0.7 |
Tipo= c(I1,I2,NI1,NI2)
dtbp=data.frame(Grupo=c(rep("Inf.",length(I1)+length(I2)),rep("No inf.",length(NI1)+length(NI2))),
Ola= c(rep("1a ola",length(I1)),rep("2a ola",length(I2)),rep("1a ola",length(NI1)),rep("2a ola",length(NI2))),
Tipo
)
ggplot(data=na.omit(dtbp))+
geom_mosaic(aes(x=product(Tipo,Ola), fill=Grupo)) +
theme_mosaic()+
ylab("Tipo de HPP")+
xlab("")+
scale_fill_discrete(name ="", labels=c("Infectadas", "No infectadas"))+
scale_x_productlist(labels=c("Infectadas\n 1a ola", "No infectadas\n 1a ola", "Infectadas\n 2a ola", "No infectadas\n 2a ola"))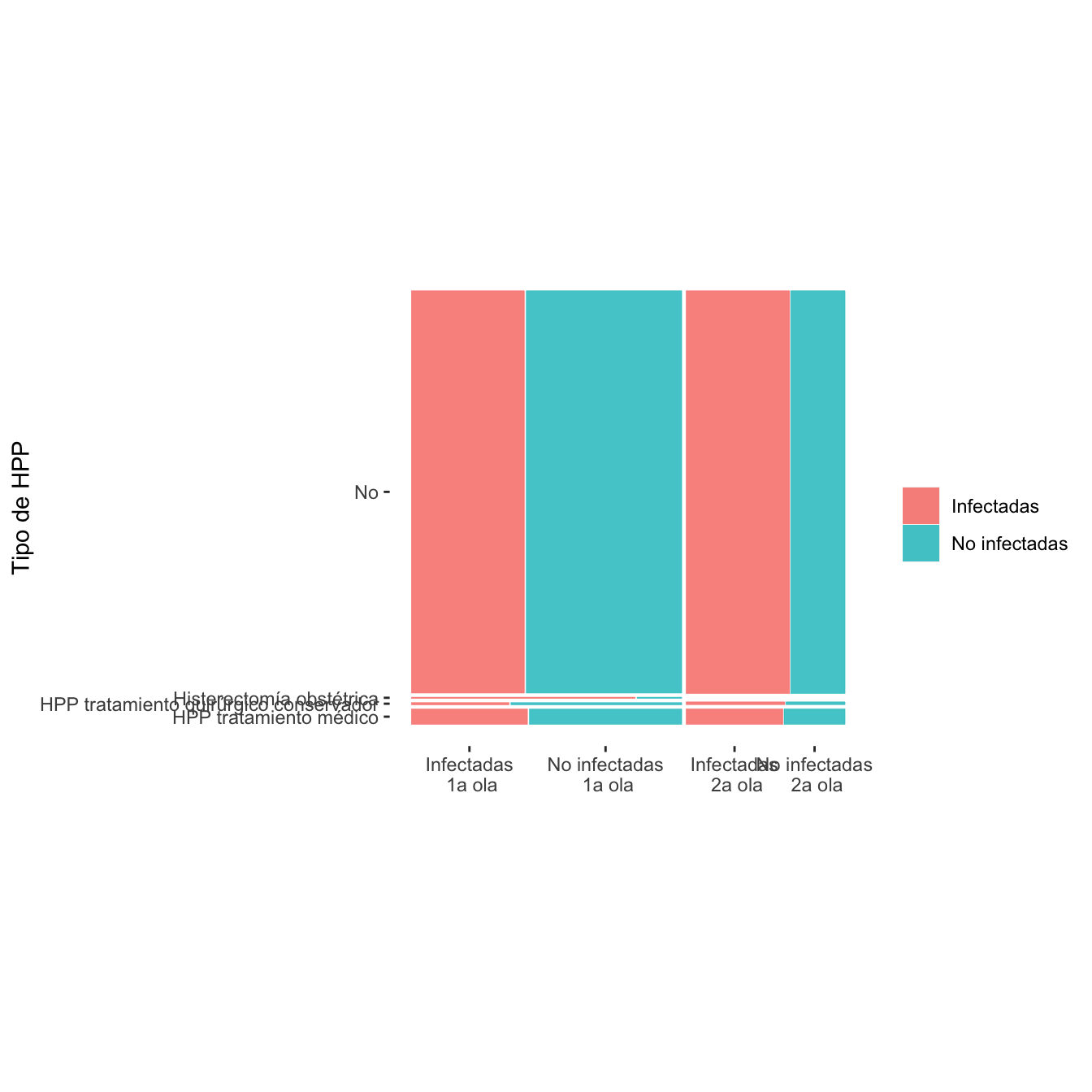
Figura 8.22:
##
## DV: Tipo
## Observations: 3217
## D: 0.1274646
## MS total: 862692.2- Test de interacción de Scheirer-Ray-Hare: p-valor, 0.42.
8.3.14 Ingreso materno en UCI
I1=Casos1w$UCI
I2=Casos2w$UCI
NI1=factor(Controls1w$UCI...9)
NI2=factor(Controls2w$UCI...9,levels=0:1)
ComparOlasRR(I1,I2,NI1,NI2,"UCI", "No UCI")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| UCI | 24 | 2.7 | 2 | 0.2 | 13 | 1.6 | 0 | 0 | 0.0093 | -0.0053 | 0.0242 | 0.204274 | 0 | 0 | 0 | 0.13762 | 1 | ||
| No UCI | 853 | 97.3 | 1189 | 99.8 | 778 | 98.4 | 416 | 100 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
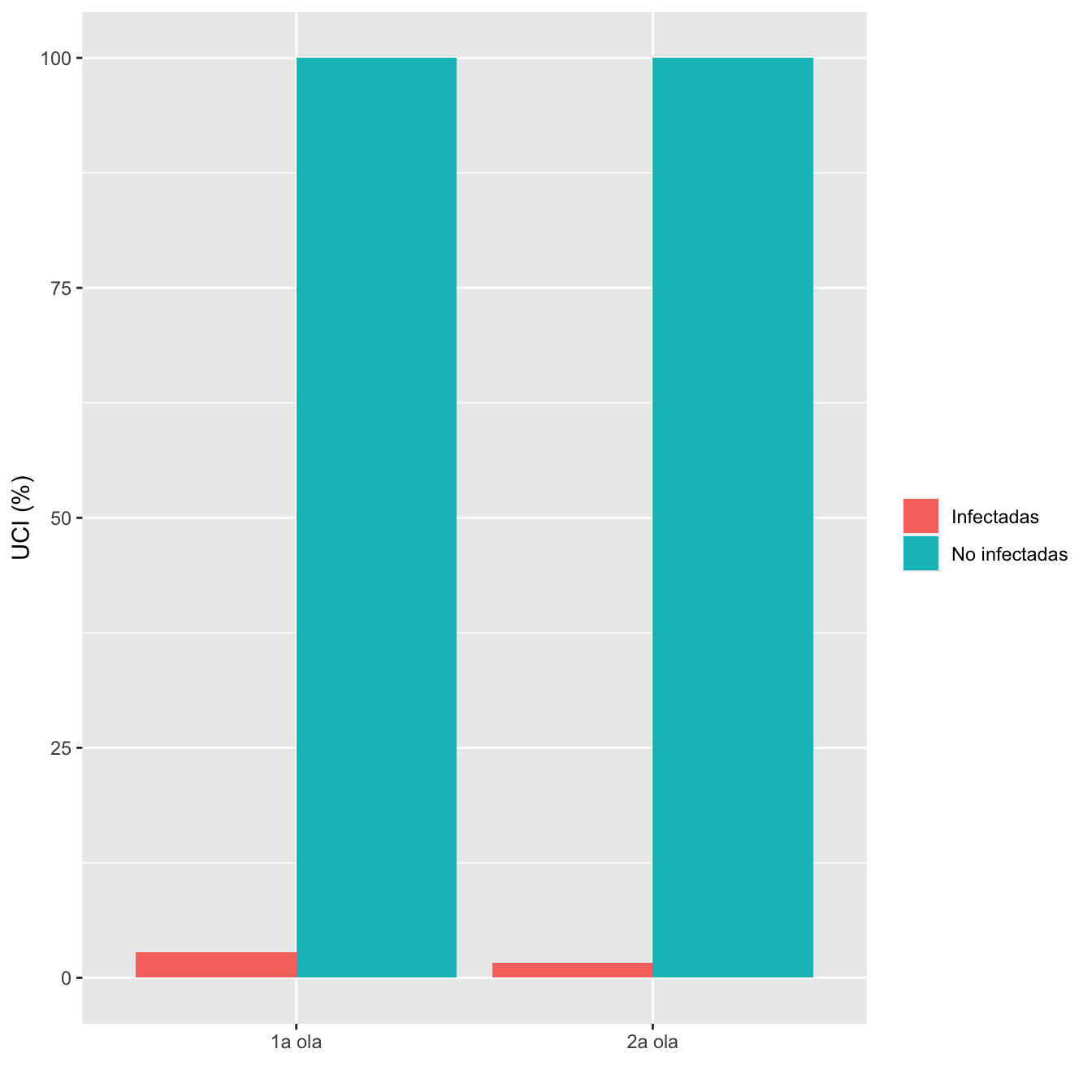
8.3.15 Rotura prematura de membranas
I1=Casos1w$Bolsa.rota.anteparto
I2=Casos2w$Bolsa.rota.anteparto
NI1=Controls1w$Bolsa.rota.anteparto
NI2=Controls2w$Bolsa.rota.anteparto
ComparOlasRR(I1,I2,NI1,NI2,"RPM", "No RPM")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| RPM | 124 | 14.1 | 130 | 10.9 | 111 | 14 | 49 | 11.8 | 0.0097 | -0.0382 | 0.0599 | 0.696105 | 1.0873 | 0.7364 | 1.6054 | 0.673785 | 0.445473 | 1 | 0.650994 |
| No RPM | 753 | 85.9 | 1061 | 89.1 | 680 | 86 | 367 | 88.2 | |||||||||||
| Datos perdidos | 0 | 0 | 0 | 0 |
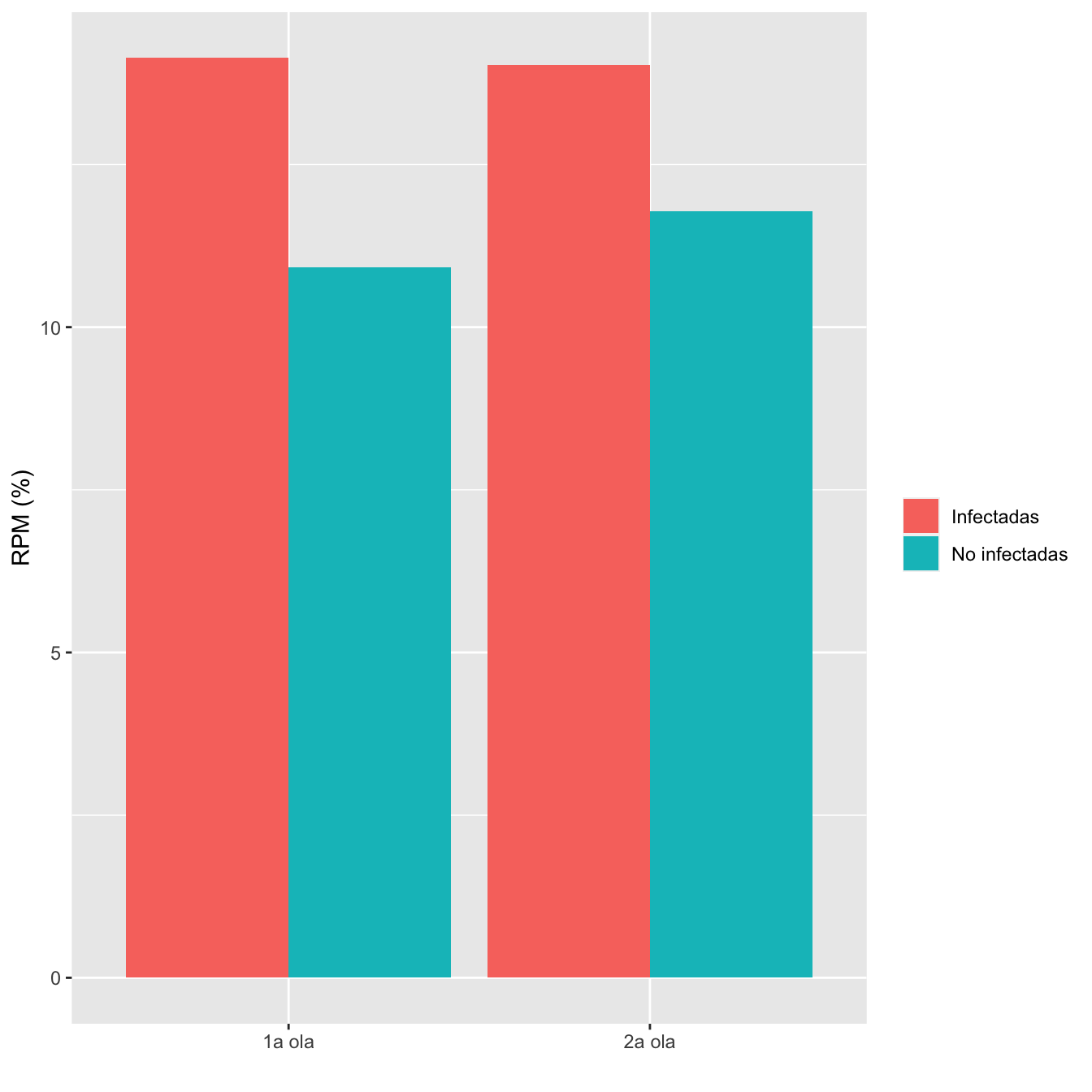
Figura 8.23:
8.3.16 Ingreso neonatal en UCIN
I1=c(Casos1w[Casos1w$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,Casos1wGM[Casos1wGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
I2=c(Casos2w[Casos2w$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,Casos2wGM[Casos2wGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
NI1=c(Controls1w[Controls1w$Feto.vivo...194=="Sí",]$Ingreso.en.UCI...213,Controls1wGM[Controls1wGM$Feto.vivo...245=="Sí",]$Ingreso.en.UCI...260)
NI2=c(Controls2w[Controls2w$Feto.vivo...194=="Sí",]$Ingreso.en.UCI...213,Controls2wGM[Controls2wGM$Feto.vivo...245=="Sí",]$Ingreso.en.UCI...260)
ComparOlasRR(I1,I2,NI1,NI2,"UCIN", "No UCIN")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| UCIN | 93 | 10.6 | 34 | 2.8 | 64 | 8.2 | 8 | 1.9 | 0.015 | -0.0171 | 0.0482 | 0.360706 | 0.875 | 0.3853 | 1.9873 | 0.749748 | 0.939581 | 0.110174 | 0.374128 |
| No UCIN | 784 | 89.4 | 1173 | 97.2 | 717 | 91.8 | 412 | 98.1 | |||||||||||
| Datos perdidos | 9 | 9 | 13 | 2 |
df =data.frame(
Factor=c(rep("Infectadas 1a ola",length(I1)), rep("No infectadas 1a ola",length(NI1)),rep("Infectadas 2a ola",length(I2)), rep("No infectadas 2a ola",length(NI2))),
Grupo=c(I1,NI1,I2,NI2 )
)
Grupo=ordered(rep(c("Infectadas","No infectadas"), 2),levels=c("Infectadas","No infectadas"))
Ola=ordered(rep(base , each=2),levels=base)
valor=as.vector(100*prop.table(table(df)[c(1,3,2,4),2:1], margin=1))[1:4]
data=data.frame(Grupo,Ola,valor)
data %>%
ggplot(aes(fill=Grupo, y=valor, x=Ola)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("UCI neonatal (%)")+
scale_fill_hue(c=100)+labs(fill="")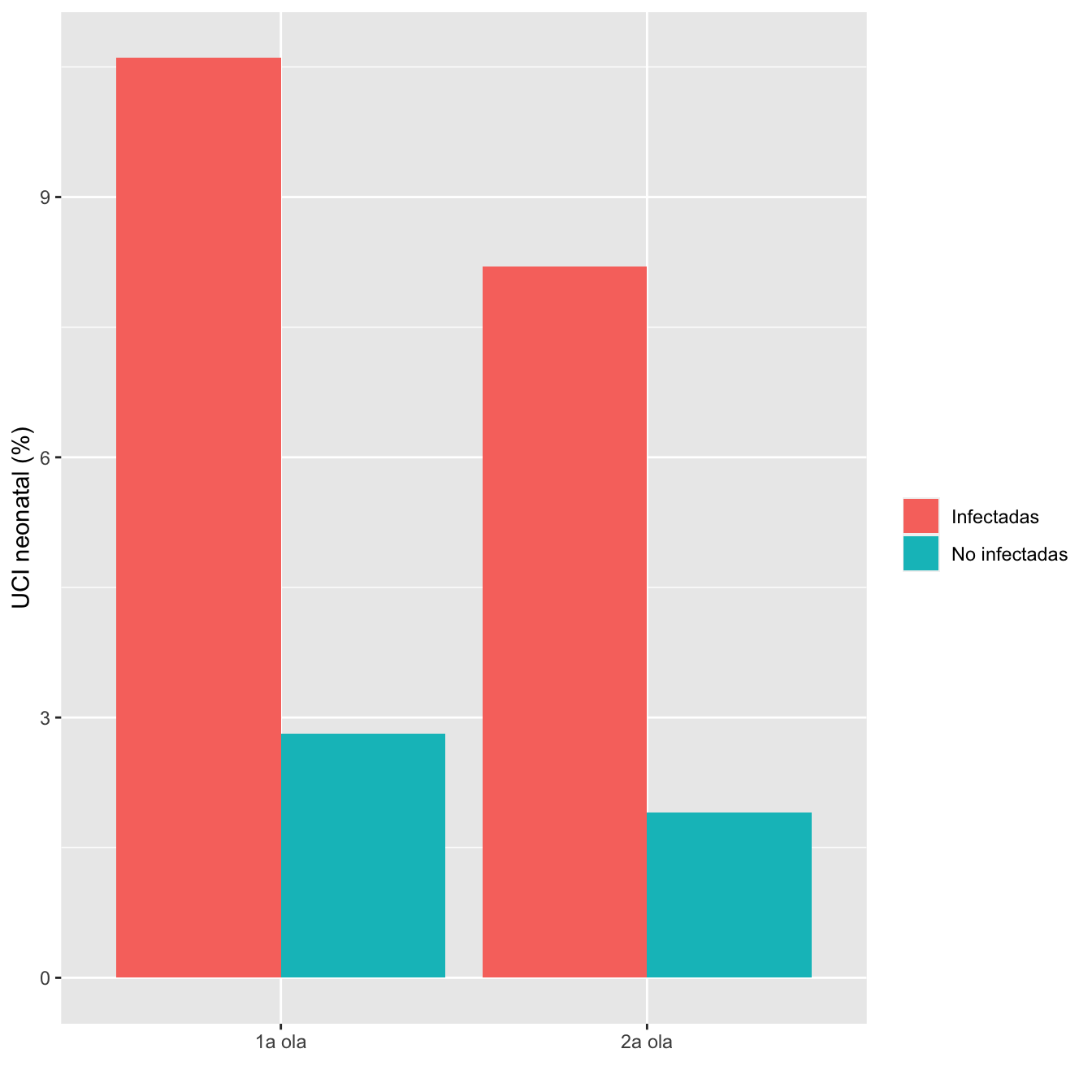
Figura 8.24:
8.3.17 Apgar
I1=c(Casos1w$APGAR.5...126,Casos1wGM$APGAR.5...150)
I2=c(Casos2w$APGAR.5...126,Casos2wGM$APGAR.5...150)
I2[I2==19]=NA
NI1=c(Controls1w$APGAR.5...200,Controls1wGM$APGAR.5...249)
NI2=c(Controls2w$APGAR.5...200,Controls2wGM$APGAR.5...249)
I1=cut(I1,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I2=cut(I2,breaks=c(-1,7,20),labels=c("0-7","8-10"))
NI1=cut(NI1,breaks=c(-1,7,20),labels=c("0-7","8-10"))
NI2=cut(NI2,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I1=ordered(I1,levels=c("8-10","0-7"))
I2=ordered(I2,levels=c("8-10","0-7"))
NI1=ordered(NI1,levels=c("8-10","0-7"))
NI2=ordered(NI2,levels=c("8-10","0-7"))
ComparOlasRR(I1,I2,NI1,NI2,"Apgar.5≤7", "Apgar.5≥8")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Apgar.5≤7 | 26 | 2.9 | 28 | 2.3 | 24 | 3 | 5 | 1.2 | -0.0122 | -0.0332 | 0.0105 | 0.273419 | 0.5004 | 0.1679 | 1.4907 | 0.213808 | 0.817923 | 1 | 0.225749 |
| Apgar.5≥8 | 860 | 97.1 | 1183 | 97.7 | 767 | 97 | 413 | 98.8 | |||||||||||
| Datos perdidos | 9 | 8 | 13 | 4 |
df =data.frame(
Factor=c(rep("Infectadas 1a ola",length(I1)), rep("No infectadas 1a ola",length(NI1)),rep("Infectadas 2a ola",length(I2)), rep("No infectadas 2a ola",length(NI2))),
Grupo=c(I1,NI1,I2,NI2 )
)
Grupo=ordered(rep(c("Infectadas","No infectadas"), 2),levels=c("Infectadas","No infectadas"))
Ola=ordered(rep(base , each=2),levels=base)
valor=as.vector(100*prop.table(table(df)[c(1,3,2,4),], margin=1))[1:4]
data=data.frame(Grupo,Ola,valor)
data %>%
ggplot(aes(fill=Grupo, y=valor, x=Ola)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("UCI materna (%)")+
scale_fill_hue(c=100)+labs(fill="")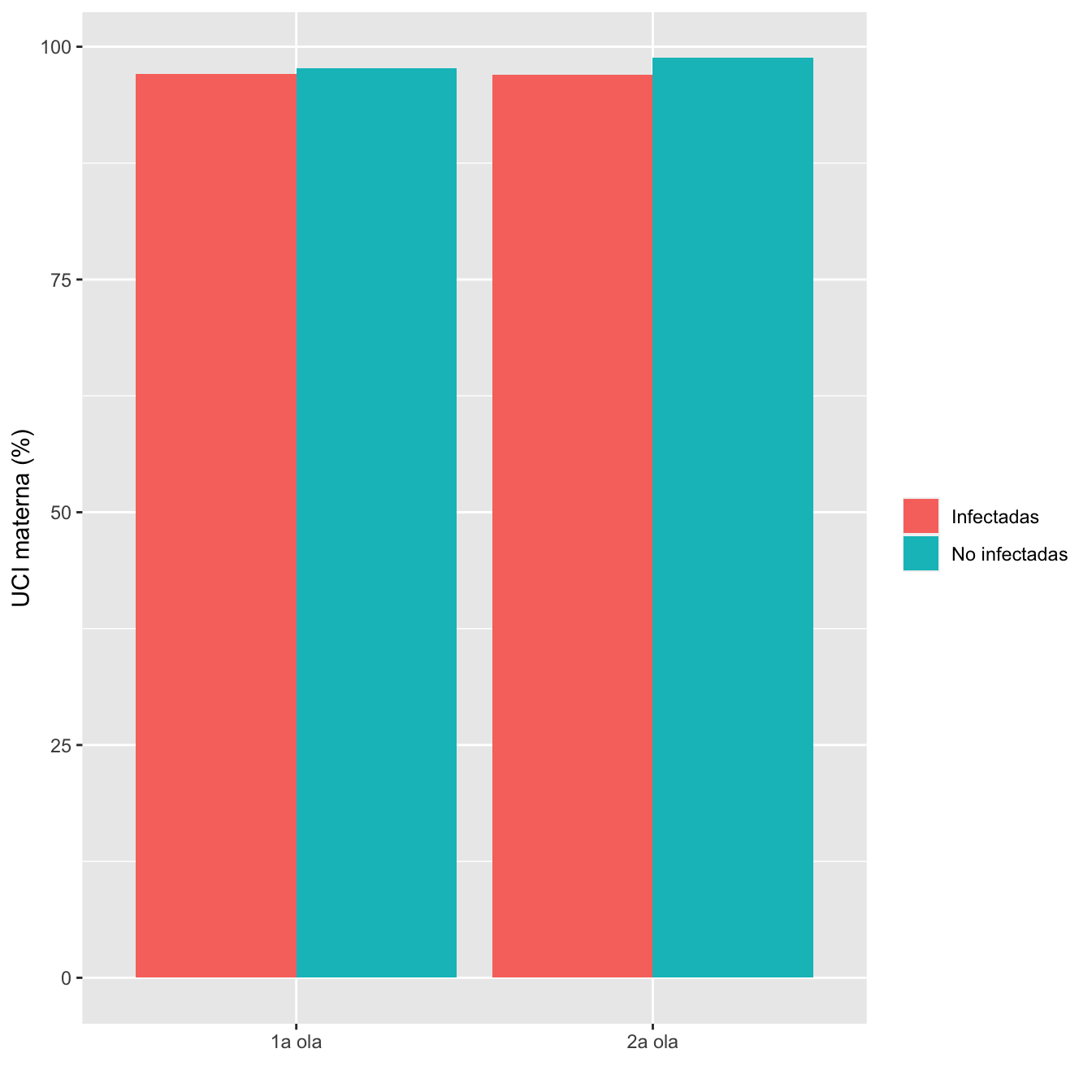
Figura 8.25:
8.3.18 Edad gestacional al parto
I1=Casos1w$EG_TOTAL_PARTO
I2=Casos2w$EG_TOTAL_PARTO
NI1=Controls1w$EDAD_GEST
NI2=Controls2w$EDAD_GEST ## [1] 0.1098393## [1] 0.7167789Igualdad de edades gestacionales medias entre olas
Para los casos: test t, p-valor 0.135; IC-95% para la diferencia de medias [-0.395, 0.053]
Para los controles: test t, p-valor 0.685; IC-95% para la difound(erencia de medias [-0.163, 0.248]
Dades=rbind(c(round(min(I1,na.rm=TRUE),1),round(max(I1,na.rm=TRUE),1), round(mean(I1,na.rm=TRUE),1),round(median(I1,na.rm=TRUE),1),round(quantile(I1,c(0.25,0.75),na.rm=TRUE),1), round(sd(I1,na.rm=TRUE),1)),
c(round(min(NI1,na.rm=TRUE),1),round(max(NI1,na.rm=TRUE),1), round(mean(NI1,na.rm=TRUE),1),round(median(NI1,na.rm=TRUE),1),round(quantile(NI1,c(0.25,0.75),na.rm=TRUE),1),round(sd(NI1,na.rm=TRUE),1)),
c(round(min(I2,na.rm=TRUE),1),round(max(I2,na.rm=TRUE),1), round(mean(I2,na.rm=TRUE),1),round(median(I2,na.rm=TRUE),1),round(quantile(I2,c(0.25,0.75),na.rm=TRUE),1), round(sd(I2,na.rm=TRUE),1)),
c(round(min(NI2,na.rm=TRUE),1),round(max(NI2,na.rm=TRUE),1), round(mean(NI2,na.rm=TRUE),1),round(median(NI2,na.rm=TRUE),1),round(quantile(NI2,c(0.25,0.75),na.rm=TRUE),1),round(sd(NI2,na.rm=TRUE),1)))
colnames(Dades)=c("Edad gest. mínima","Edad gest. máxima","Edad gest. media", "Edad gest. mediana", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Infectadas 1a ola", "No infectadas 1a ola","Infectadas 2a ola", "No infectadas 2a ola")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Edad gest. mínima | Edad gest. máxima | Edad gest. media | Edad gest. mediana | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Infectadas 1a ola | 16.4 | 42.4 | 38.9 | 39.4 | 38.3 | 40.4 | 2.5 |
| No infectadas 1a ola | 23.7 | 42.0 | 39.5 | 39.7 | 38.9 | 40.6 | 1.9 |
| Infectadas 2a ola | 18.0 | 42.1 | 39.1 | 39.4 | 38.4 | 40.4 | 2.2 |
| No infectadas 2a ola | 23.0 | 42.0 | 39.4 | 39.6 | 38.9 | 40.4 | 1.7 |
data =data.frame(
name=ordered(c( rep("Infectadas 1a ola",length(I1)), rep("No infectadas 1a ola",length(NI1)),rep("Infectadas 2a ola",length(I2)), rep("No infectadas 2a ola",length(NI2))),levels=c("Infectadas 1a ola","No infectadas 1a ola","Infectadas 2a ola","No infectadas 2a ola")),
Edades=c( I1,NI1,I2,NI2 ),
Color=c( rep("Infectadas",length(I1)), rep("No infectadas",length(NI1)),rep("Infectadas",length(I2)), rep("No infectadas",length(NI2)))
)
data %>%
ggplot( aes(x=name, y=Edades, fill=name)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title=element_text(size=11)
) +
xlab("")+
ylab("Edades gestacionales (semanas)")+
scale_fill_manual(values=c("#F8766D" ,"#00BFC4" ,"#F8766D", "#00BFC4"))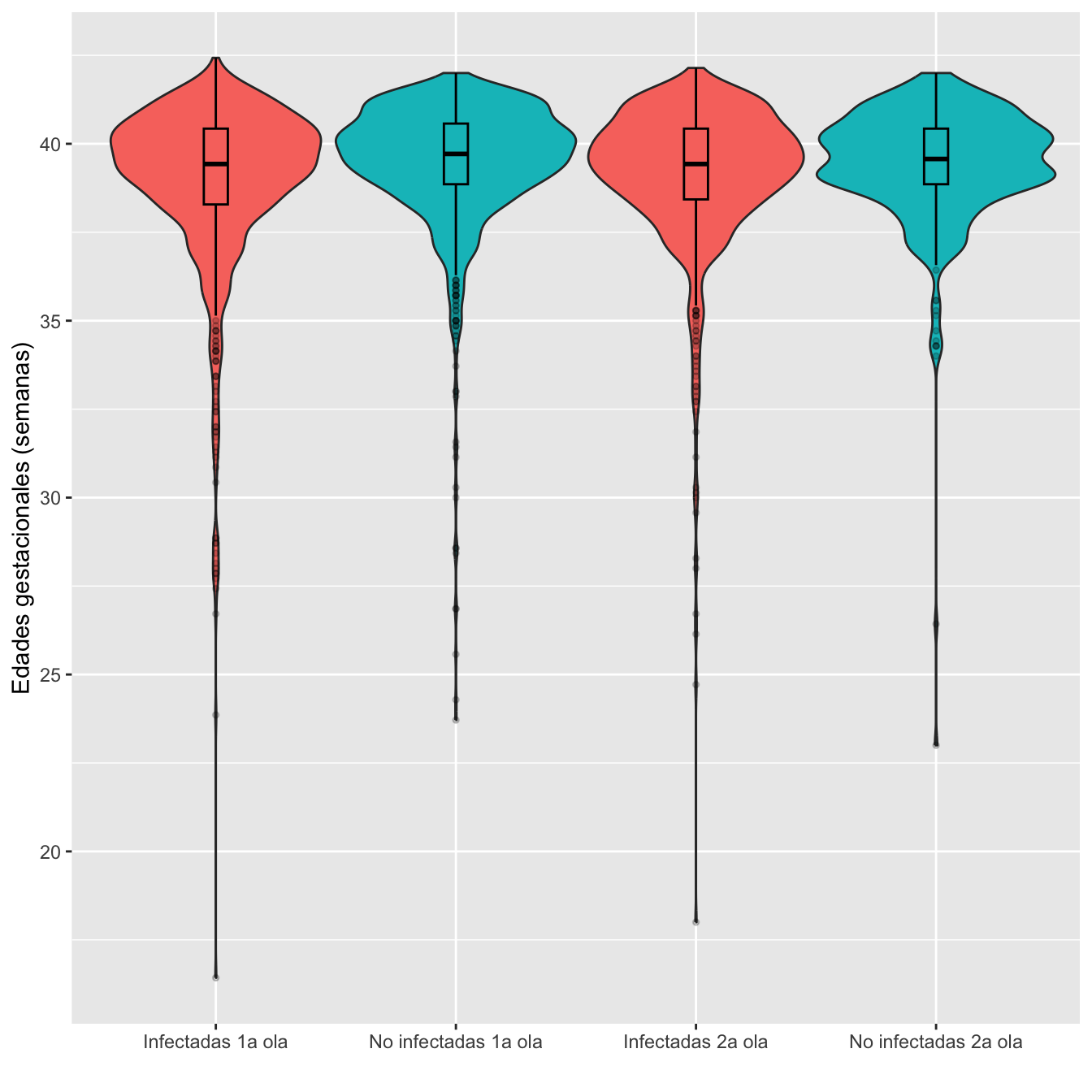
Figura 8.26:
- Ninguno de los cuatro grupos de edades gestacionales se ajusta a distribución normal: test de Shapiro-Wilkis, p-valor máximo \(2\times 10^{-23}\)
Tipo= c(I1,I2,NI1,NI2)
dtbp=data.frame(Grupo=c(rep("Inf.",length(I1)+length(I2)),rep("No inf.",length(NI1)+length(NI2))),
Ola= c(rep("1a ola",length(I1)),rep("2a ola",length(I2)),rep("1a ola",length(NI1)),rep("2a ola",length(NI2))),
Tipo
)
SRH=scheirerRayHare(Tipo ~ Grupo + Ola,
data=dtbp,verbose=FALSE)$p.value[3]- Test de interacción de Scheirer-Ray-Hare: p-valor 0.126: no hay evidencia de que la diferencia de edades medias gestacionales de infectadas y no infectadas sea diferente según la ola.
8.3.19 Peso de neonatos al nacimiento
I1=c(Casos1w[Casos1w$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,Casos1wGM[Casos1wGM$Feto.vivo=="Sí",]$Peso._gramos_...148)
NI1=c(Controls1w[Controls1w$Feto.vivo...194=="Sí",]$Peso._gramos_...198,Controls1wGM[Controls1wGM$Feto.vivo...245=="Sí",]$Peso._gramos_...247)
I2=c(Casos2w[Casos2w$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,Casos2wGM[Casos2wGM$Feto.vivo=="Sí",]$Peso._gramos_...148)
NI2=c(Controls2w[Controls2w$Feto.vivo...194=="Sí",]$Peso._gramos_...198,Controls2wGM[Controls2wGM$Feto.vivo...245=="Sí",]$Peso._gramos_...247)Dades=rbind(c(round(min(I1,na.rm=TRUE),1),round(max(I1,na.rm=TRUE),1), round(mean(I1,na.rm=TRUE),1),round(median(I1,na.rm=TRUE),1),round(quantile(I1,c(0.25,0.75),na.rm=TRUE),1), round(sd(I1,na.rm=TRUE),1)),
c(round(min(NI1,na.rm=TRUE),1),round(max(NI1,na.rm=TRUE),1), round(mean(NI1,na.rm=TRUE),1),round(median(NI1,na.rm=TRUE),1),round(quantile(NI1,c(0.25,0.75),na.rm=TRUE),1),round(sd(NI1,na.rm=TRUE),1)),
c(round(min(I2,na.rm=TRUE),1),round(max(I2,na.rm=TRUE),1), round(mean(I2,na.rm=TRUE),1),round(median(I2,na.rm=TRUE),1),round(quantile(I2,c(0.25,0.75),na.rm=TRUE),1), round(sd(I2,na.rm=TRUE),1)),
c(round(min(NI2,na.rm=TRUE),1),round(max(NI2,na.rm=TRUE),1), round(mean(NI2,na.rm=TRUE),1),round(median(NI2,na.rm=TRUE),1),round(quantile(NI2,c(0.25,0.75),na.rm=TRUE),1),round(sd(NI2,na.rm=TRUE),1)))
colnames(Dades)=c("Peso mínimo","Peso máximo","Peso medio", "Peso mediano", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Infectadas 1a ola", "No infectadas 1a ola","Infectadas 2a ola", "No infectadas 2a ola")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Peso mínimo | Peso máximo | Peso medio | Peso mediano | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Infectadas 1a ola | 315 | 5190 | 3157.3 | 3230.0 | 2872.5 | 3528.8 | 615.8 |
| No infectadas 1a ola | 490 | 4700 | 3242.9 | 3280.0 | 2947.5 | 3600.0 | 549.6 |
| Infectadas 2a ola | 585 | 4740 | 3229.0 | 3287.5 | 2913.8 | 3602.5 | 587.1 |
| No infectadas 2a ola | 538 | 4760 | 3265.7 | 3270.0 | 2970.0 | 3580.0 | 503.6 |
data =data.frame(
name=ordered(c( rep("Infectadas 1a ola",length(I1)), rep("No infectadas 1a ola",length(NI1)),rep("Infectadas 2a ola",length(I2)), rep("No infectadas 2a ola",length(NI2))),levels=c("Infectadas 1a ola","No infectadas 1a ola","Infectadas 2a ola","No infectadas 2a ola")),
Edades=c( I1,NI1,I2,NI2 ),
Color=c( rep("Infectadas",length(I1)), rep("No infectadas",length(NI1)),rep("Infectadas",length(I2)), rep("No infectadas",length(NI2)))
)
data %>%
ggplot( aes(x=name, y=Edades, fill=name)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title=element_text(size=11)
) +
xlab("")+
ylab("Pesos (gramos)")+
scale_fill_manual(values=c("#F8766D" ,"#00BFC4" ,"#F8766D", "#00BFC4"))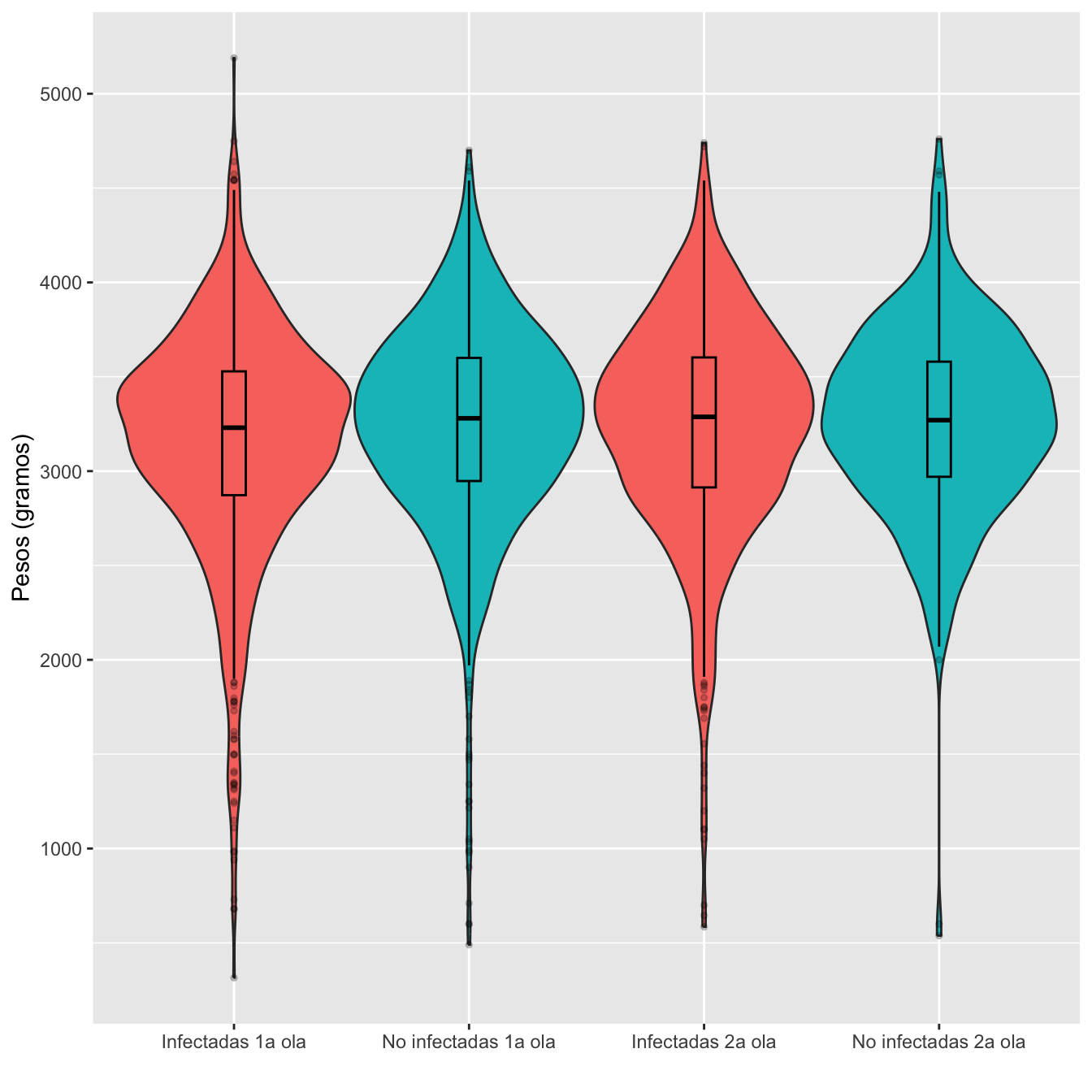
Figura 8.27:
- Ninguno de los cuatro grupos de edades gestacionales se ajusta a distribución normal: test de Shapiro-Wilkis, p-valor máximo \(3\times 10^{-8}\)
Tipo= c(I1,I2,NI1,NI2)
dtbp=data.frame(Grupo=c(rep("Inf.",length(I1)+length(I2)),rep("No inf.",length(NI1)+length(NI2))),
Ola= c(rep("1a ola",length(I1)),rep("2a ola",length(I2)),rep("1a ola",length(NI1)),rep("2a ola",length(NI2))),
Tipo
)
SRH=scheirerRayHare(Tipo ~ Grupo + Ola,
data=dtbp,verbose=FALSE)$p.value[3]## [1] 0.9520945## [1] 0.2056903Igualdad de edades gestacionales medias entre olas
Para los casos: test t, p-valor 0.016; IC-95% para la diferencia de medias [-130.116, -13.26]
Para los controles: test t, p-valor 0.456; IC-95% para la difound(erencia de medias [-82.923, 37.253]
Test de interacción de Scheirer-Ray-Hare: p-valor 0.194: no hay evidencia de que la diferencia de pesos medios de hijos de infectadas y no infectadas sea diferente según la ola.
I1w=cut(I1,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
NI1w=cut(NI1,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
I2w=cut(I2,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
NI2w=cut(NI2,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
I1w=ordered(I1w,levels=c(0,1))
I2w=ordered(I2w,levels=c(0,1))
NI1w=ordered(NI1w,levels=c(0,1))
NI2w=ordered(NI2w,levels=c(0,1))
ComparOlasRR(I1w,I2w,NI1w,NI2w,"Bajo peso", "No bajo peso")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Bajo peso | 100 | 11.5 | 91 | 7.6 | 72 | 9.3 | 22 | 5.3 | -9e-04 | -0.0397 | 0.0399 | 0.964724 | 0.8644 | 0.506 | 1.4767 | 0.593854 | 0.221133 | 0.169895 | 0.145856 |
| No bajo peso | 770 | 88.5 | 1113 | 92.4 | 700 | 90.7 | 393 | 94.7 | |||||||||||
| Datos perdidos | 16 | 12 | 22 | 7 |
df =data.frame(
Factor=c(rep("Infectadas 1a ola",length(I1w)), rep("No infectadas 1a ola",length(NI1w)),rep("Infectadas 2a ola",length(I2w)), rep("No infectadas 2a ola",length(NI2w))),
Grupo=c(I1w,NI1w,I2w,NI2w )
)
Grupo=ordered(rep(c("Infectadas","No infectadas"), 2),levels=c("Infectadas","No infectadas"))
Ola=ordered(rep(base , each=2),levels=base)
valor=as.vector(100*prop.table(table(df)[c(1,3,2,4),2:1], margin=1))[1:4]
data=data.frame(Grupo,Ola,valor)
data %>%
ggplot(aes(fill=Grupo, y=valor, x=Ola)) +
geom_bar(position="dodge", stat="identity")+
xlab("")+
ylab("RPM (%)")+
scale_fill_hue(c=100)+labs(fill="")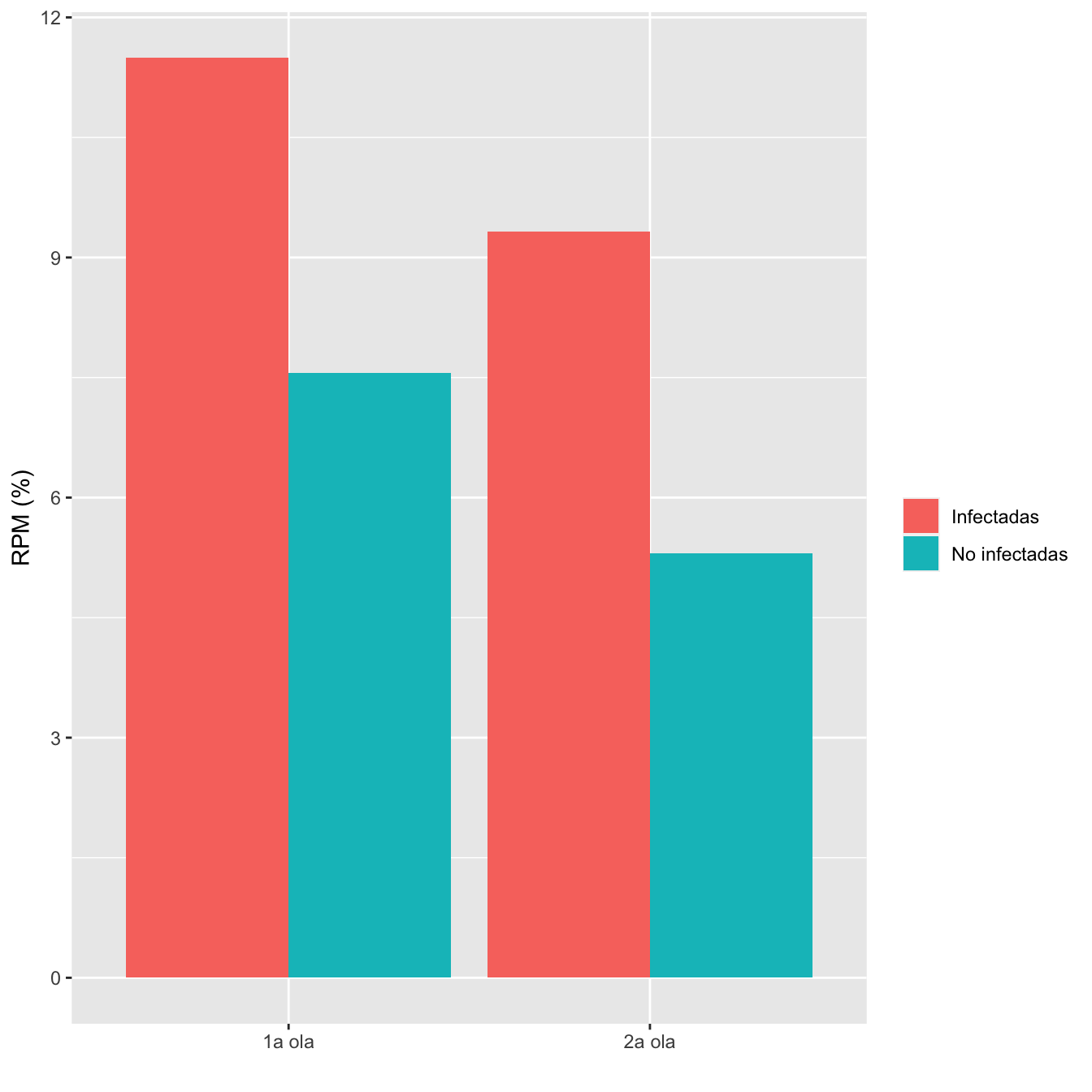
8.3.20 Fetos muertos anteparto
I1w1=Casos1w$Feto.muerto.intraútero
I1w1[I1w1=="Sí"]="Muerto"
I1w1[I1w1=="No"]="Vivo"
I1w2=Casos1wGM$Feto.vivo
I1w2[I1w2=="Sí"]="Vivo"
I1w2[I1w2=="No"]="Muerto"
I1w=c(I1w1,I1w2)
n_I1wFT=length(I1w)
n_VI1w=as.vector(table(I1w))[2]
I1w=ordered(I1w,levels=c("Vivo","Muerto"))
NI1w1=Controls1w$Feto.vivo...194
NI1w2=Controls1wGM$Feto.vivo...245
NI1w=c(NI1w1,NI1w2)
NI1w[NI1w=="Sí"]="Vivo"
NI1w[NI1w=="No"]="Muerto"
NI1w=factor(NI1w,levels=c("Muerto","Vivo"))
n_NI1wFT=length(NI1w)
n_VNI1w=as.vector(table(NI1w))[2]
I2w=ordered(I2w,levels=c("Vivo","Muerto"))
#
#
I2w1=Casos2w$Feto.muerto.intraútero
I2w1[I2w1=="Sí"]="Muerto"
I2w1[I2w1=="No"]="Vivo"
I2w2=Casos2wGM$Feto.vivo
I2w2[I2w2=="Sí"]="Vivo"
I2w2[I2w2=="No"]="Muerto"
I2w=c(I2w1,I2w2)
n_I2wFT=length(I2w)
n_VI2w=as.vector(table(I2w))[2]
NI1w=ordered(NI1w,levels=c("Vivo","Muerto"))
NI2w1=Controls2w$Feto.vivo...194
NI2w2=Controls2wGM$Feto.vivo...245
NI2w=c(NI2w1,NI2w2)
NI2w[NI2w=="Sí"]="Vivo"
NI2w[NI2w=="No"]="Muerto"
NI2w=factor(NI2w,levels=c("Muerto","Vivo"))
n_NI2wFT=length(NI2w)
n_VNI2w=as.vector(table(NI2w))[2]
NI1w=ordered(NI1w,levels=c("Vivo","Muerto"))
ComparOlasRRwe(I1w,I2w,NI1w,NI2w,"Feto muerto anteparto", "Feto vivo")| Infectadas 1a ola (N) | Infectadas 1a ola (%) | No infectadas 1a ola (N) | No infectadas 1 ola (%) | Infectadas 2 ola (N) | Infectadas 2 ola (%) | No infectadas 2 ola (N) | No infectadas 2 ola (%) | Diferencia RA | Extr. inf. IC RA | Extr. sup. IC RA | p-valor igualdad RA | Cociente RR | Extr. inf. IC RR | Extr. sup. IC RR | p-valor igualdad RR | p-valor global olas | p-valor casos | p-valor controles | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Feto muerto anteparto | 9 | 1 | 3 | 0.2 | 794 | 98.8 | 422 | 100 | 0.02 | 0.0109 | 0.0328 | 0.00345 | 4.1353 | 1.1227 | 15.2317 | 0.032845 | 0 | 0 | 0 |
| Feto vivo | 885 | 99 | 1214 | 99.8 | 10 | 1.2 | 0 | 0 | |||||||||||
| Datos perdidos | 1 | 2 | 0 | 0 |