Lección 4 Descripción de la muestra de casos según sintomatología
Hubo:
357 infecciones asintomáticas (40.71%)
314 infecciones leves (35.8%)
206 infecciones graves (23.49%)
Síntomas=ordered(c("Asintomática", "Leve", "Grave"),levels=c("Asintomática", "Leve", "Grave"))
valor=as.vector(prop.table(table(Sint)))
data <- data.frame(Síntomas,valor)
ggplot(data, aes(x=Síntomas, y=valor,fill=Síntomas)) +
geom_bar(stat="identity")+
xlab("")+
ylab("")+
scale_fill_brewer(palette = "Dark2") +
theme(legend.position="none")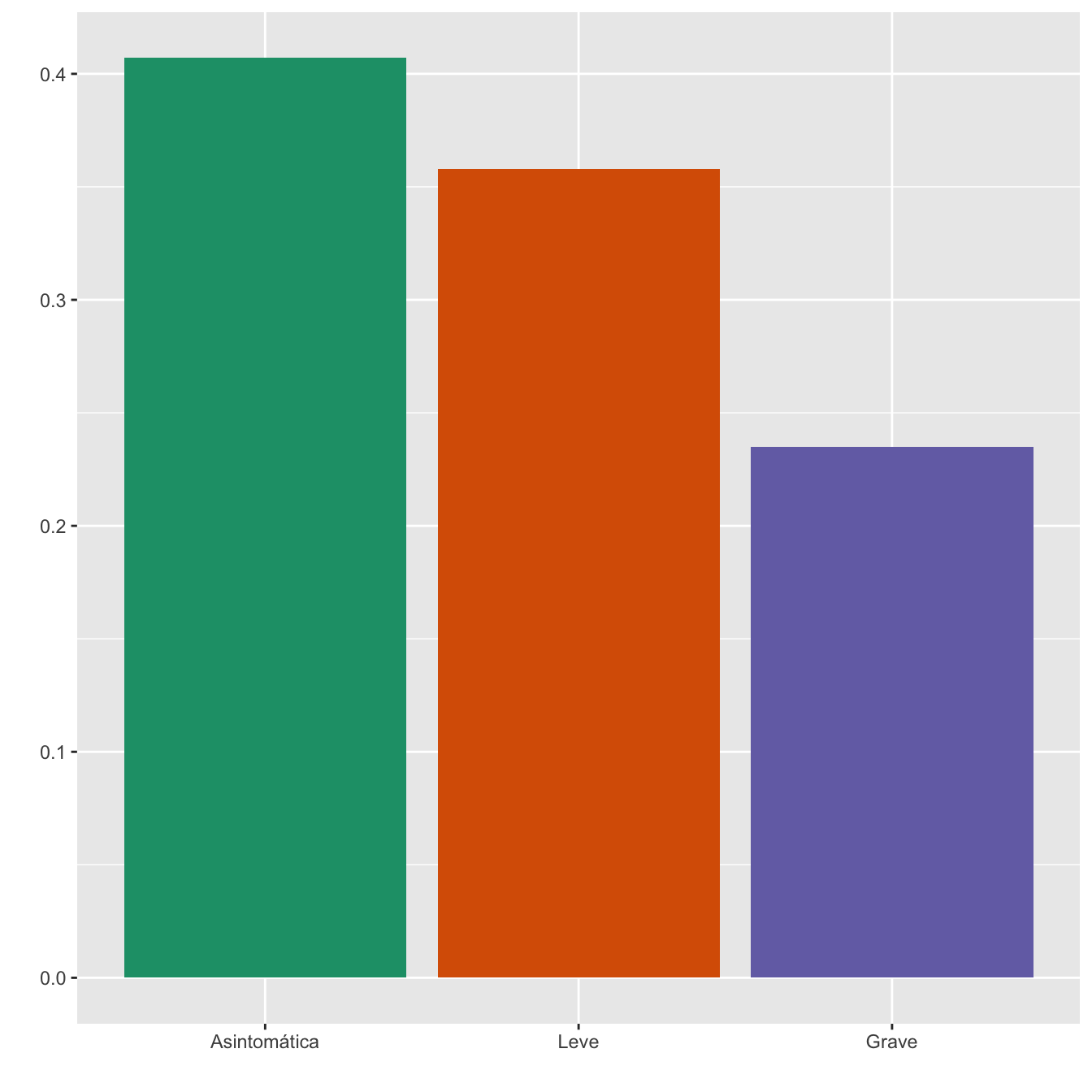
Figura 4.1:
Trim=Casos$Trim.Diag
NA_T=length(Trim[is.na(Trim)])
NA_S=length(Sint[is.na(Sint)])
DF=data.frame(Trimestre=Trim,Síntomas=Sint)
taula=table(DF)[,1:3]
proptaula=round(100*prop.table(table(DF)),2)[,1:3]
EEExt=cbind(c(as.vector(taula[,1]),sum(taula[,1])),
c(as.vector(proptaula[,1]), round(100*sum(taula[,1])/n_I,2)),
c(as.vector(taula[,2]),sum(taula[,2])),
c(as.vector(proptaula[,2]), round(100*sum(taula[,2])/n_I,2)),
c(as.vector(taula[,3]),sum(taula[,3])),
c(as.vector(proptaula[,3]), round(100*sum(taula[,3])/n_I,2))
)
rownames(EEExt)=c("1er trimestre", "2o trimestre", "3er trimestre","Total")
colnames(EEExt)=Columnes.Sint
EEExt %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | |
|---|---|---|---|---|---|---|
| 1er trimestre | 2 | 0.23 | 29 | 3.31 | 10 | 1.14 |
| 2o trimestre | 20 | 2.28 | 94 | 10.72 | 44 | 5.02 |
| 3er trimestre | 335 | 38.20 | 191 | 21.78 | 152 | 17.33 |
| Total | 357 | 21.40 | 314 | 18.82 | 206 | 12.35 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=3),levels=c("Asintomática", "Leve", "Grave"))
Grupo=rep(c("1er trimestre", "2o trimestre", "3er trimestre") , 3)
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("Trimestre de infección")+
ylab("")+
scale_fill_brewer(palette = "Dark2") 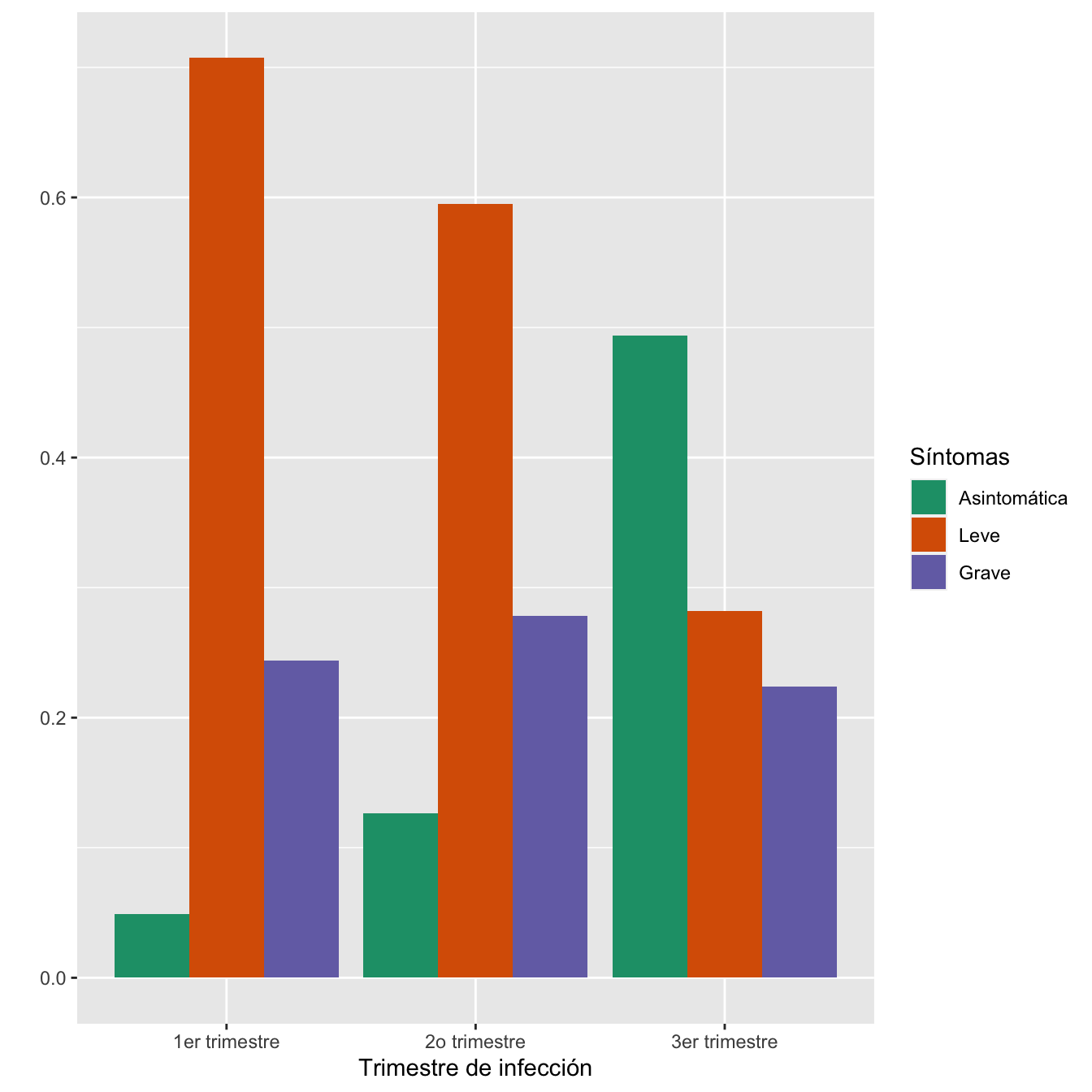
Figura 4.2:
4.1 Antecedentes maternos
4.1.1 Edades
I=Casos$Edad._años_
DF=data.frame(Edad=I,Sint=Casos$SINTOMAS_DIAGNOSTICO)
DF %>%
ggplot( aes(x=Sint, y=Edad, fill=Sint)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title=element_text(size=11)
) +
xlab("")+
ylab("Edades (años)")+
scale_fill_brewer(palette="Dark2")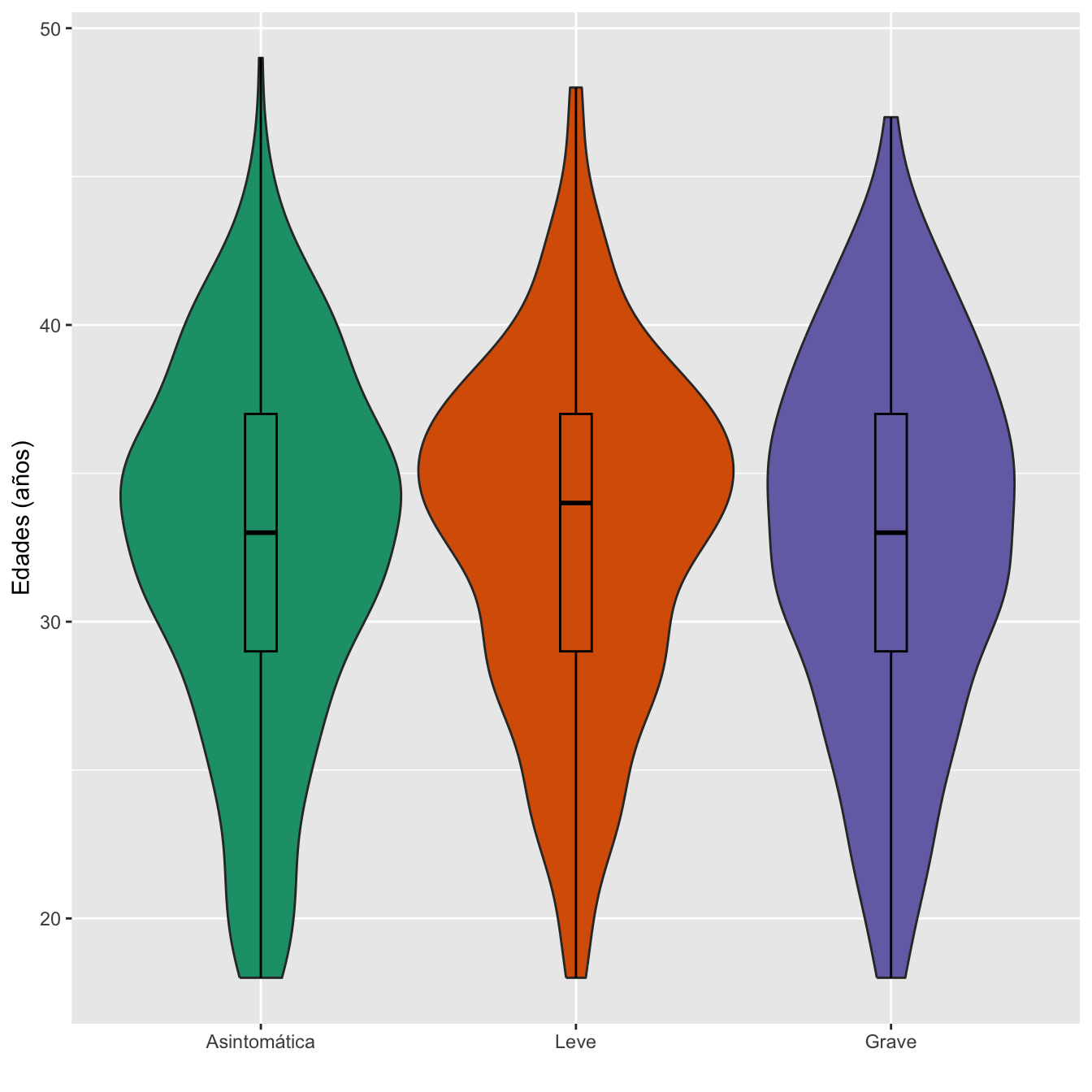
I1=I[Casos$SINTOMAS_DIAGNOSTICO=="Asintomática"]
I2=I[Casos$SINTOMAS_DIAGNOSTICO=="Leve"]
I3=I[Casos$SINTOMAS_DIAGNOSTICO=="Grave"]
Dades=rbind(c(round(min(I1,na.rm=TRUE),1),round(max(I1,na.rm=TRUE),1), round(mean(I1,na.rm=TRUE),1),round(median(I1,na.rm=TRUE),1),round(quantile(I1,c(0.25,0.75),na.rm=TRUE),1), round(sd(I1,na.rm=TRUE),1)),
c(round(min(I2,na.rm=TRUE),1),round(max(I2,na.rm=TRUE),1), round(mean(I2,na.rm=TRUE),1),round(median(I2,na.rm=TRUE),1),round(quantile(I2,c(0.25,0.75),na.rm=TRUE),1), round(sd(I2,na.rm=TRUE),1)),
c(round(min(I3,na.rm=TRUE),1),round(max(I3,na.rm=TRUE),1), round(mean(I3,na.rm=TRUE),1),round(median(I3,na.rm=TRUE),1),round(quantile(I3,c(0.25,0.75),na.rm=TRUE),1), round(sd(I3,na.rm=TRUE),1)))
colnames(Dades)=c("Edad mínima","Edad máxima","Edad media", "Edad mediana", "1er cuartil", "3er cuartil", "Desv. típica")
rownames(Dades)=c("Asintomática", "Leve","Grave")
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Edad mínima | Edad máxima | Edad media | Edad mediana | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Asintomática | 18 | 49 | 32.5 | 33 | 29 | 37 | 6.2 |
| Leve | 18 | 48 | 33.0 | 34 | 29 | 37 | 5.8 |
| Grave | 18 | 47 | 32.7 | 33 | 29 | 37 | 6.2 |
Ajuste de las edades maternas a distribuciones normales: test de Shapiro-Wilks, p-valores \(7\times 10^{-5}\), \(0.003\) y 0.0259, respectivamente
Edades medias: test de Kruskal-Wallis, p-valor 0.7215272
Desviaciones típicas: test de Fligner-Killeen, p-valor 0.32
Contrastes post hoc por parejas
Contraste=c("Asíntomatica vs Leve","Asíntomatica vs Grave", "Leve vs Grave")
pvals=round(3*c(t.test(I1,I2,var.equal=TRUE)$p.value, t.test(I1,I3,var.equal=TRUE)$p.value,
t.test(I2,I3,var.equal=TRUE)$p.value),4)
dades=data.frame(Contraste=Contraste,"p-valor"=pvals)
dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Contraste | p.valor |
|---|---|
| Asíntomatica vs Leve | 0.9155 |
| Asíntomatica vs Grave | 1.9550 |
| Leve vs Grave | 1.9925 |
En tablas como la que sigue:
p-valor: El p-valor del test \(\chi^2\) de si la distribución de la fila correspondiente en casos sintomáticos es la misma que la del global de la muestra de casos, ajustado por Bonferroni cuando haya más de 2 filas
Tipo: El tipo de test \(\chi^2\), paramétrico, siempre que es posible, o Montecarlo, cuando no se dan las condiciones necesarias para que tenga sentido efectuar el test paramétrico
I.cut=cut(I,breaks=c(0,30,40,100),labels=c("18-30","31-40",">40"))
Tabla.DMCasosm(I.cut, c("18-30", "31-40", ">40"))| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| 18-30 | 116 | 32.7 | 103 | 32.9 | 67 | 32.7 | 1 | Paramétrico |
| 31-40 | 211 | 59.4 | 186 | 59.4 | 119 | 58.0 | 1 | Paramétrico |
| >40 | 28 | 7.9 | 24 | 7.7 | 19 | 9.3 | 1 | Paramétrico |
| Datos perdidos | 2 | 1 | 1 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=3),levels=c("Asintomática", "Leve", "Grave"))
Grupo=ordered(rep(c("18-30","31-40",">40") , 3),levels=c("18-30","31-40",">40"))
taula=table(data.frame(Edad=I.cut,Síntomas=Sint))
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("Edad")+
ylab("")+
scale_fill_brewer(palette = "Dark2") 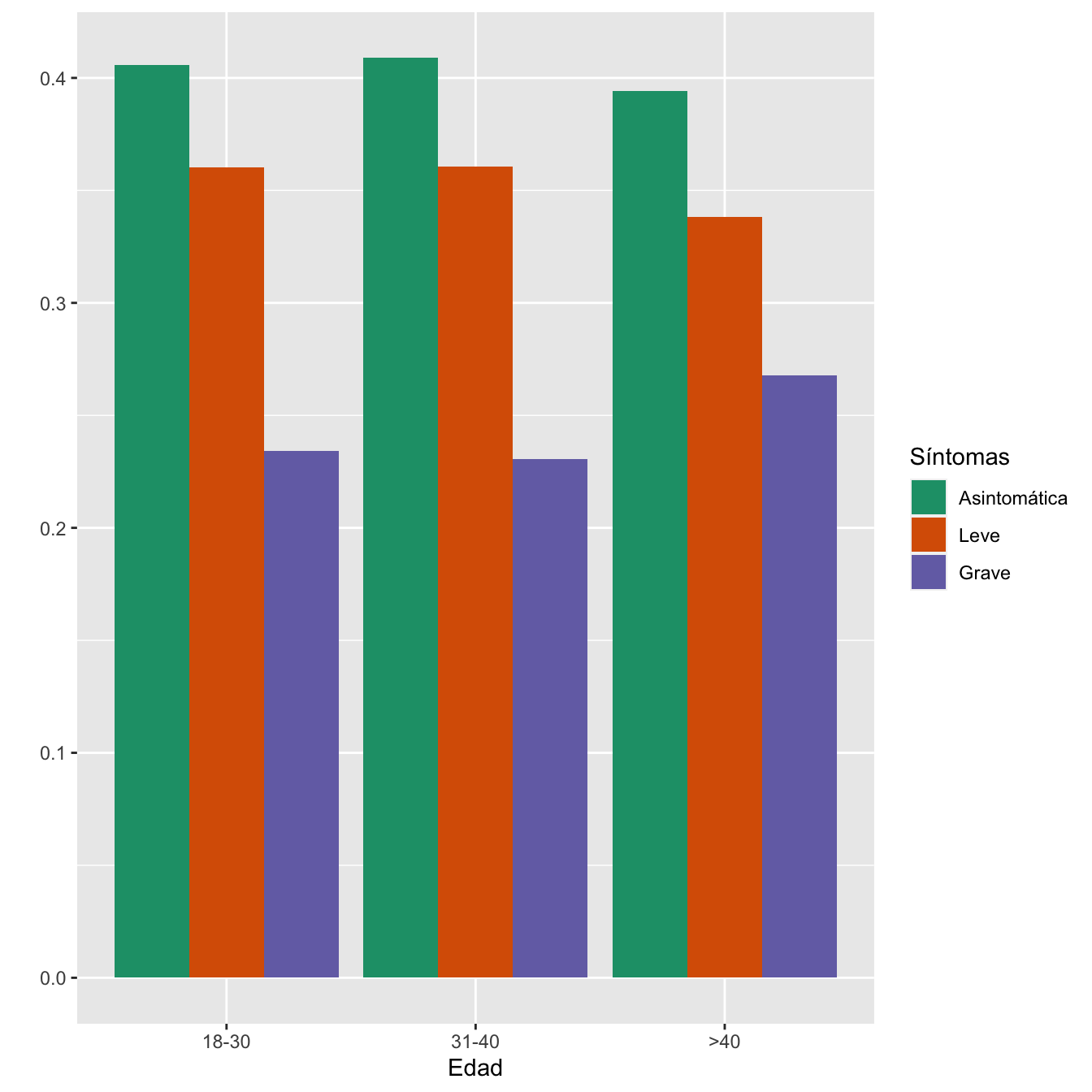
Figura 4.3:
- Asociación entre los grupos de sintomatología y las franjas de edad: test \(\chi^2\), p-valor 0.975
4.1.2 Etnia
I=Casos$Etnia
I[I=="Asia"]="Asiática"
I=factor(I)
Tabla.DMCasosm(I,c("Árabe", "Asiática", "Blanca", "Latinoamericana","Negra"))| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Árabe | 28 | 7.8 | 20 | 6.4 | 6 | 2.9 | 0.332868 | Paramétrico |
| Asiática | 12 | 3.4 | 5 | 1.6 | 7 | 3.4 | 1.000000 | Paramétrico |
| Blanca | 248 | 69.5 | 196 | 62.6 | 93 | 45.6 | 0.000001 | Paramétrico |
| Latinoamericana | 59 | 16.5 | 85 | 27.2 | 96 | 47.1 | 0.000000 | Paramétrico |
| Negra | 10 | 2.8 | 7 | 2.2 | 2 | 1.0 | 1.000000 | Montecarlo |
| Datos perdidos | 0 | 1 | 2 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=5),levels=c("Asintomática", "Leve", "Grave"))
Grupo=ordered(rep(c("Árabe", "Asiática", "Blanca", "Latinoamericana","Negra") , 3),levels=c("Árabe", "Asiática", "Blanca", "Latinoamericana","Negra"))
taula=table(data.frame(Etnia=I,Síntomas=Sint))
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
xlab("Etnia")+
ylab("")+
scale_fill_brewer(palette = "Dark2") 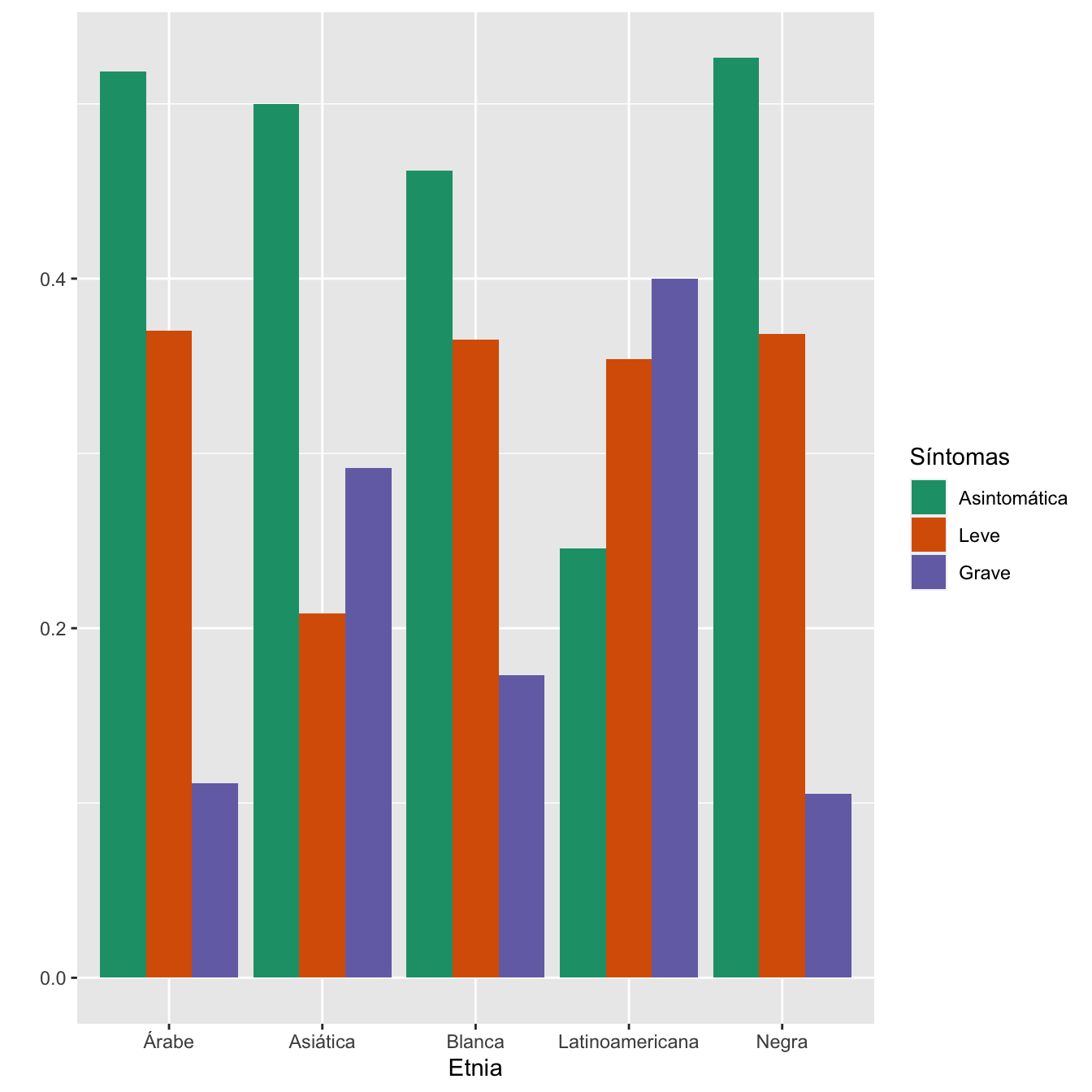
Figura 4.4:
- Asociación entre los grupos de sintomatología y las etnias: test \(\chi^2\), p-valor \(4\times 10^{-11}\)
4.1.3 Hábito tabáquico
taula=table(data.frame(Casos$FUMADORA_CAT,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$FUMADORA_CAT,"Fumadoras", "No fumadoras")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Fumadoras | 38 | 11.1 | 42 | 13.8 | 16 | 8.3 | 0.173771 | Paramétrico |
| No fumadoras | 304 | 88.9 | 263 | 86.2 | 176 | 91.7 | ||
| Datos perdidos | 15 | 9 | 14 |
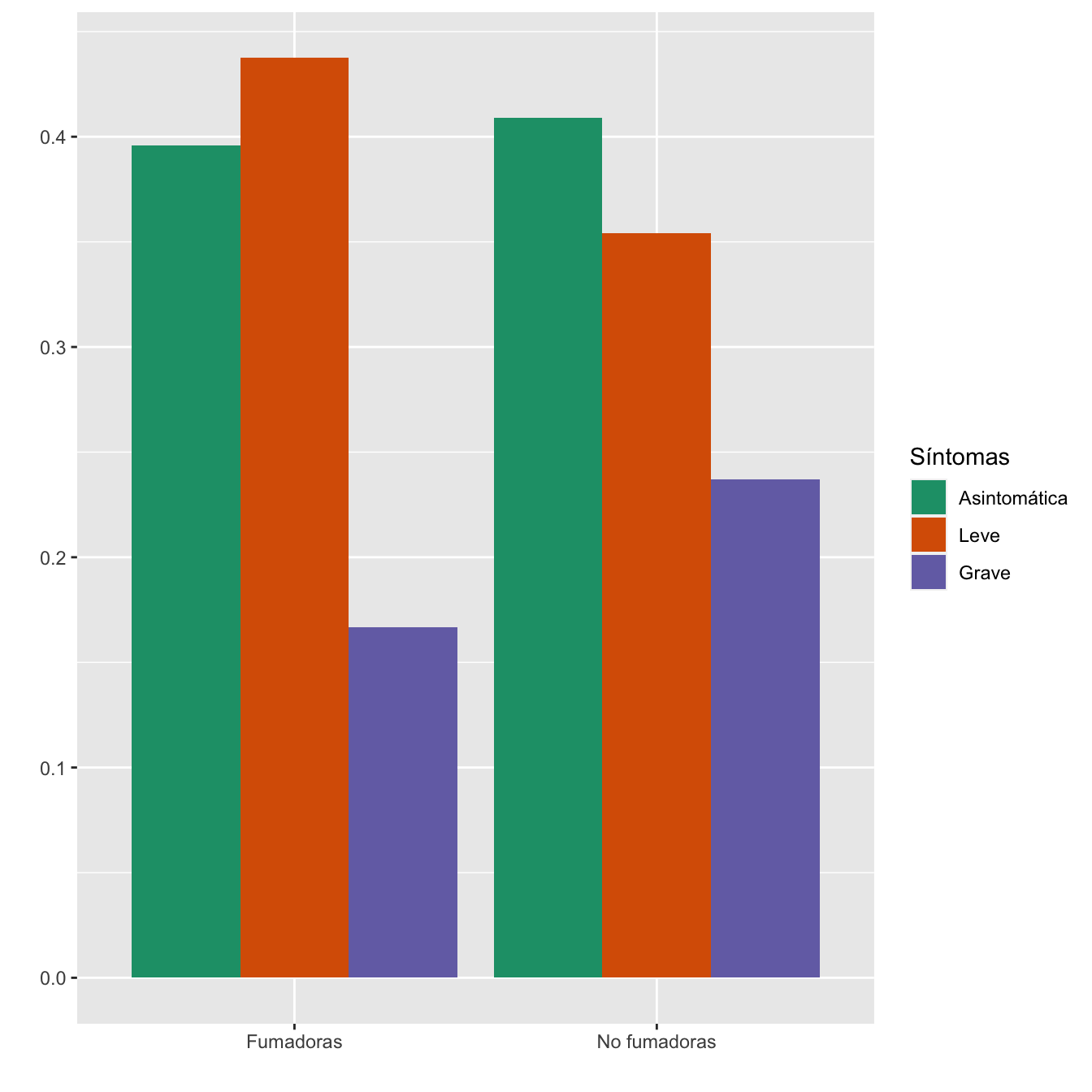
Figura 4.5:
- Potencia del test: 0.369
4.1.4 Obesidad
taula=table(data.frame(Casos$Obesidad,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Obesidad,"Obesas", "No obesas")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Obesas | 48 | 13.4 | 61 | 19.4 | 52 | 25.2 | 0.001928 | Paramétrico |
| No obesas | 309 | 86.6 | 253 | 80.6 | 154 | 74.8 | ||
| Datos perdidos | 0 | 0 | 0 |
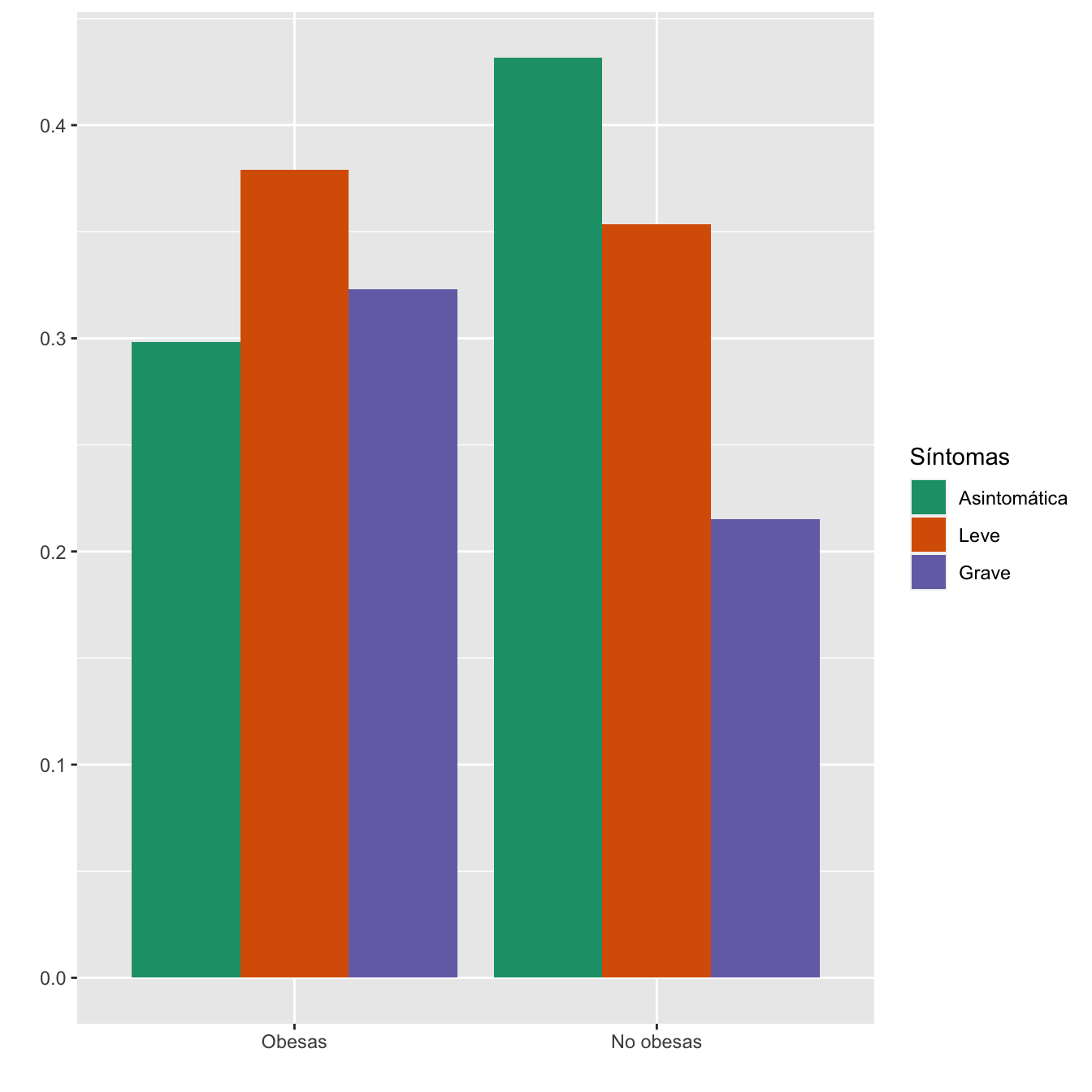
Figura 4.6:
- Potencia del test: 0.896
4.1.5 Hipertensión pregestacional
taula=table(data.frame(Casos$Hipertensión.pregestacional,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Hipertensión.pregestacional,"HTA", "No HTA")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| HTA | 5 | 1.4 | 4 | 1.3 | 5 | 2.4 | 0.615638 | Montecarlo |
| No HTA | 352 | 98.6 | 310 | 98.7 | 201 | 97.6 | ||
| Datos perdidos | 0 | 0 | 0 |
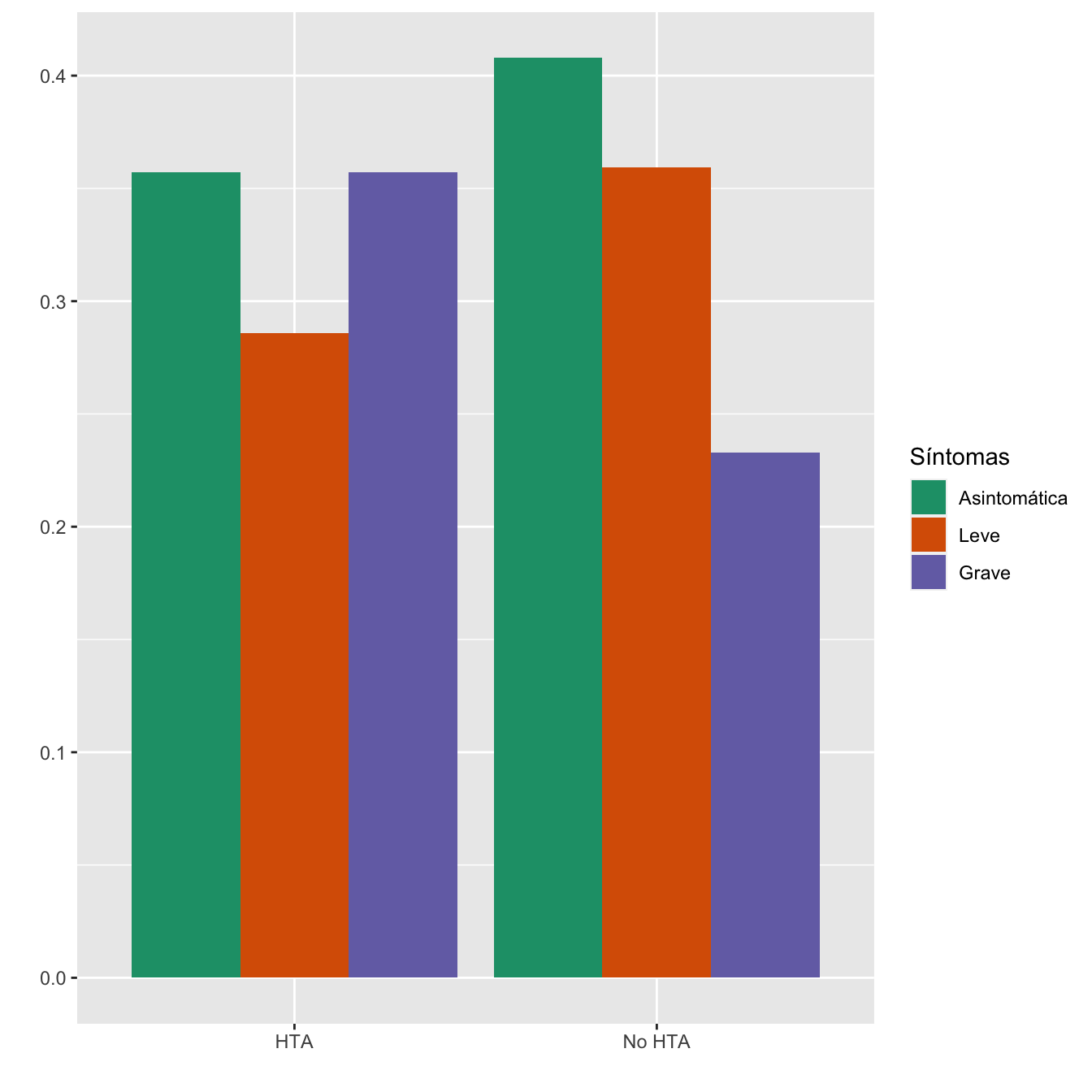
Figura 4.7:
- Potencia del test: 0.151
4.1.6 Diabetes
taula=table(data.frame(Casos$DIABETES,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$DIABETES,"DM", "No DM")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| DM | 2 | 0.6 | 8 | 2.5 | 4 | 1.9 | 0.117688 | Montecarlo |
| No DM | 355 | 99.4 | 306 | 97.5 | 202 | 98.1 | ||
| Datos perdidos | 0 | 0 | 0 |
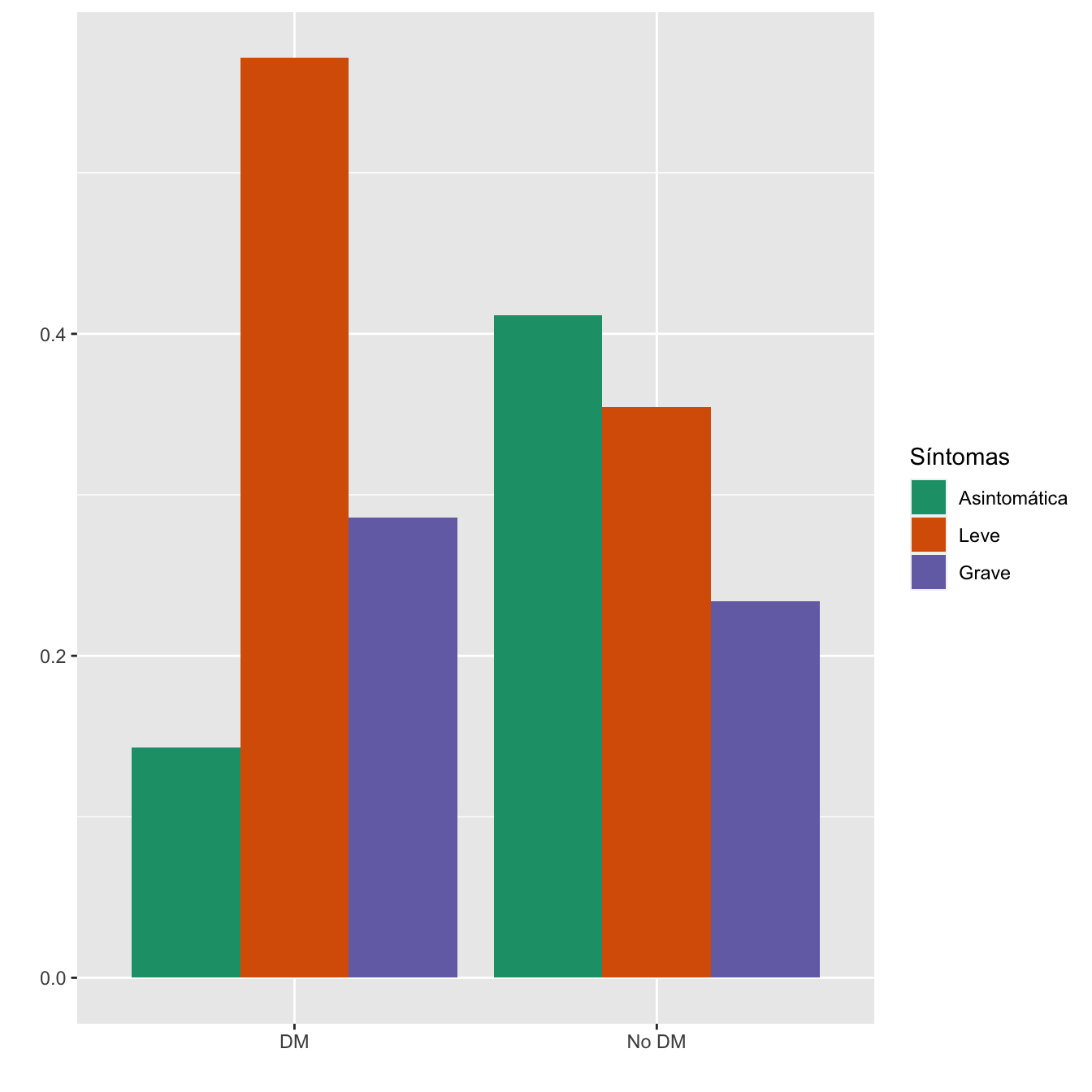
Figura 4.8:
- Potencia del test: 0.452
4.1.7 Cardiopatía
taula=table(data.frame(Casos$Enfermedad.cardiaca.crónica,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Enfermedad.cardiaca.crónica,"ECC", "No ECC")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| ECC | 4 | 1.1 | 3 | 1 | 4 | 1.9 | 0.639936 | Montecarlo |
| No ECC | 353 | 98.9 | 311 | 99 | 202 | 98.1 | ||
| Datos perdidos | 0 | 0 | 0 |
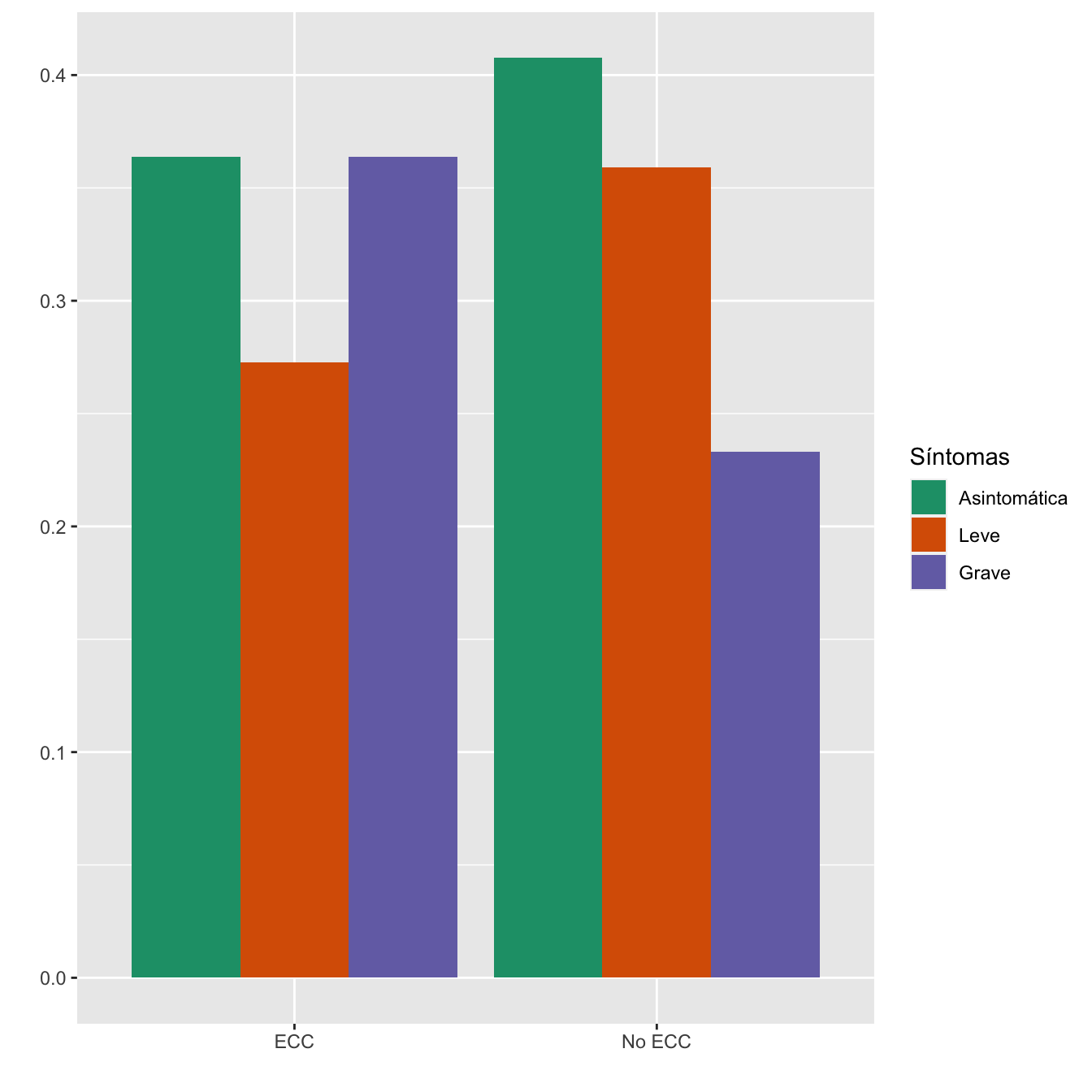
Figura 4.9:
- Potencia del test: 0.138
4.1.8 Enfermedad pulmonar crónica
INA=Casos$Enfermedad.pulmonar.crónica.no.asma
IA=Casos$Diagnóstico.clínico.de.Asma
I.EPC=rep(NA,length(Sint))
for (i in 1:length(Sint)){I.EPC[i]=max(INA[i],IA[i],na.rm=TRUE)}
taula=table(data.frame(I.EPC,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(I.EPC,"EPC", "No EPC")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| EPC | 11 | 3.1 | 18 | 5.7 | 9 | 4.4 | 0.242472 | Paramétrico |
| No EPC | 346 | 96.9 | 296 | 94.3 | 197 | 95.6 | ||
| Datos perdidos | 0 | 0 | 0 |
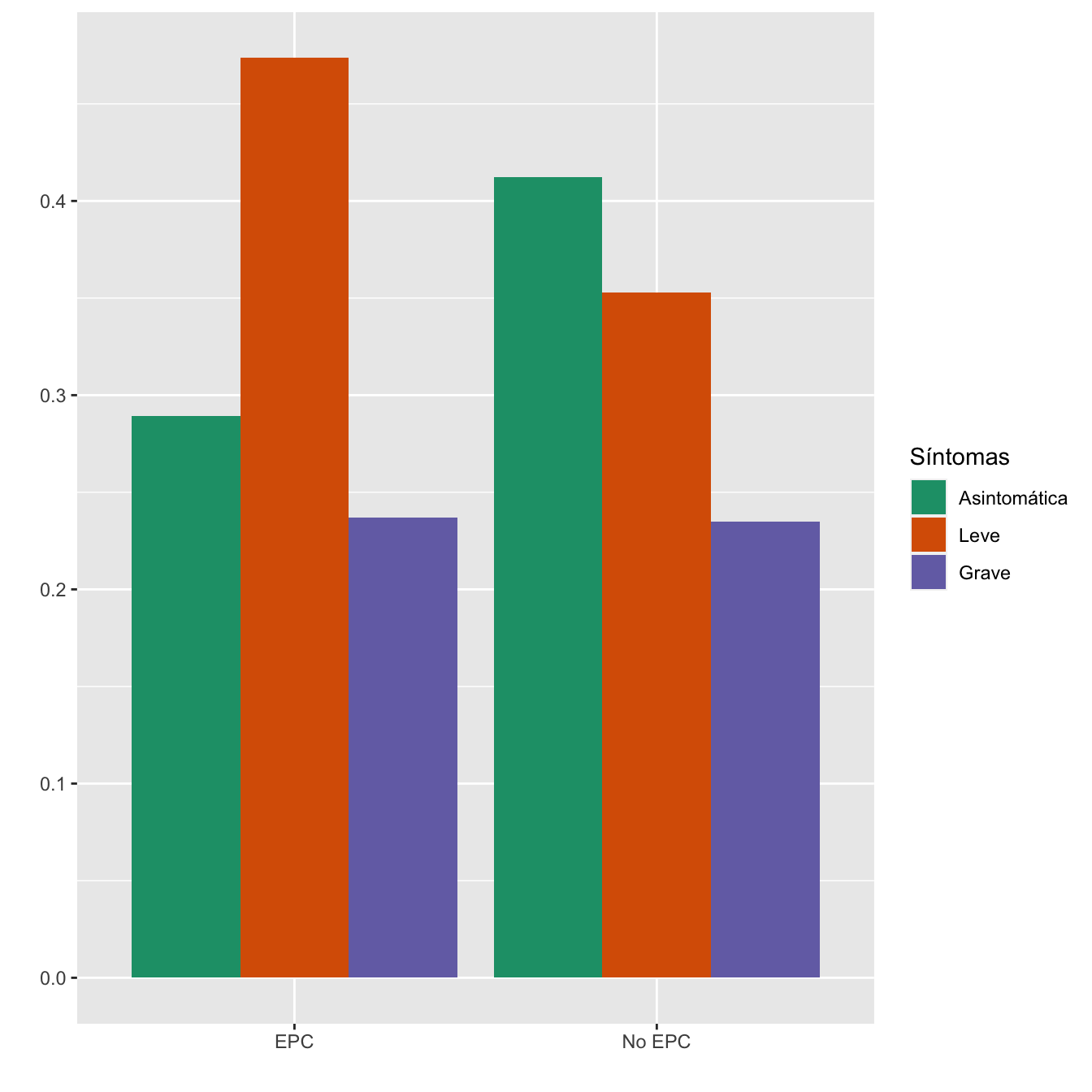
Figura 4.10:
- Potencia del test: 0.306
4.1.9 Gestación múltiple
taula=table(data.frame(Casos$Gestación.Múltiple,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Gestación.Múltiple,"Gestación múltiple", "Gestación única")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Gestación múltiple | 2 | 0.6 | 12 | 3.8 | 4 | 1.9 | 0.008999 | Montecarlo |
| Gestación única | 355 | 99.4 | 302 | 96.2 | 202 | 98.1 | ||
| Datos perdidos | 0 | 0 | 0 |
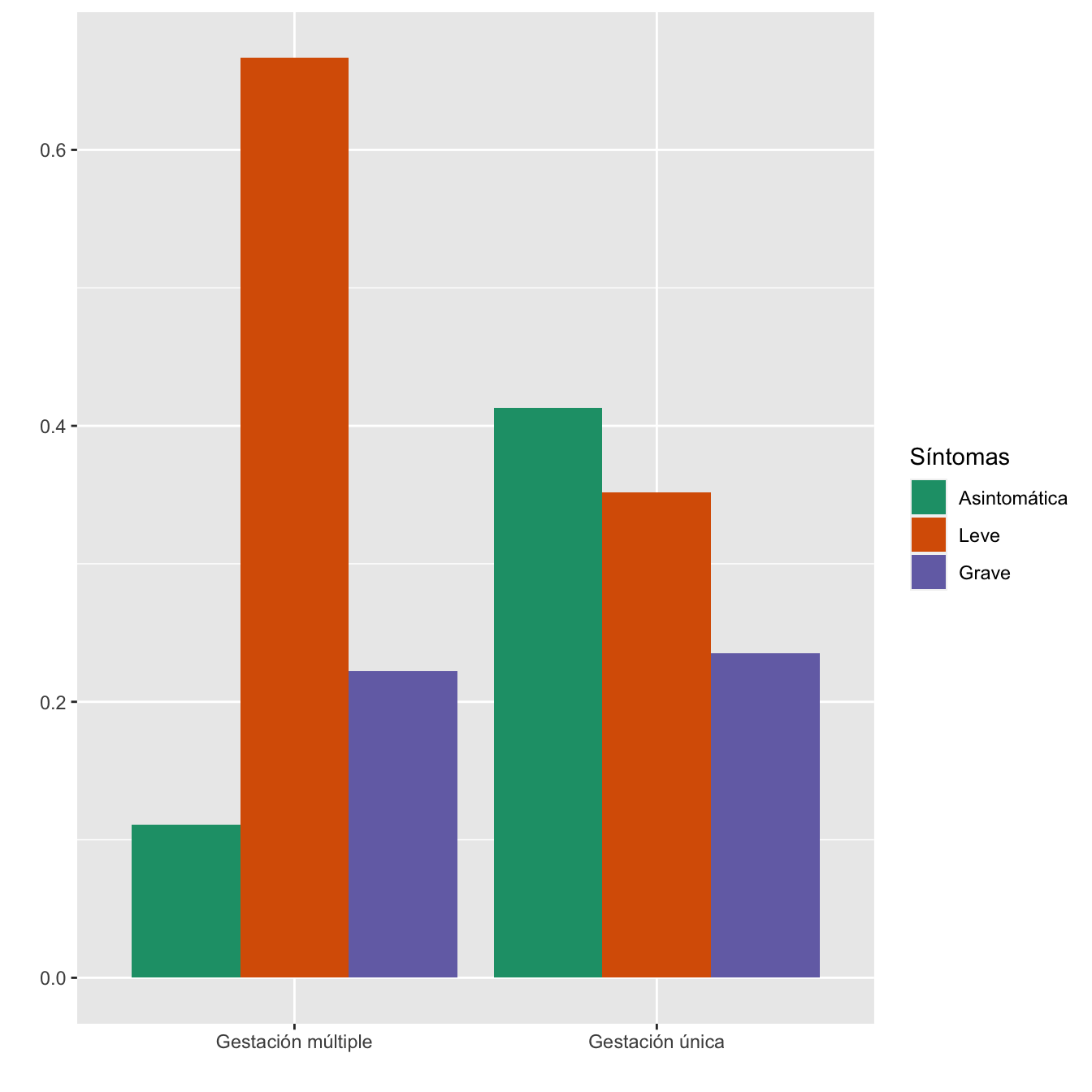
Figura 4.11:
- Potencia del test: 0.764
4.1.10 Paridad
taula=table(data.frame(Casos$NULIPARA,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$NULIPARA, "Nulípara","Multípara")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Nulípara | 130 | 37.5 | 123 | 39.4 | 66 | 32.4 | 0.258148 | Paramétrico |
| Multípara | 217 | 62.5 | 189 | 60.6 | 138 | 67.6 | ||
| Datos perdidos | 10 | 2 | 2 |
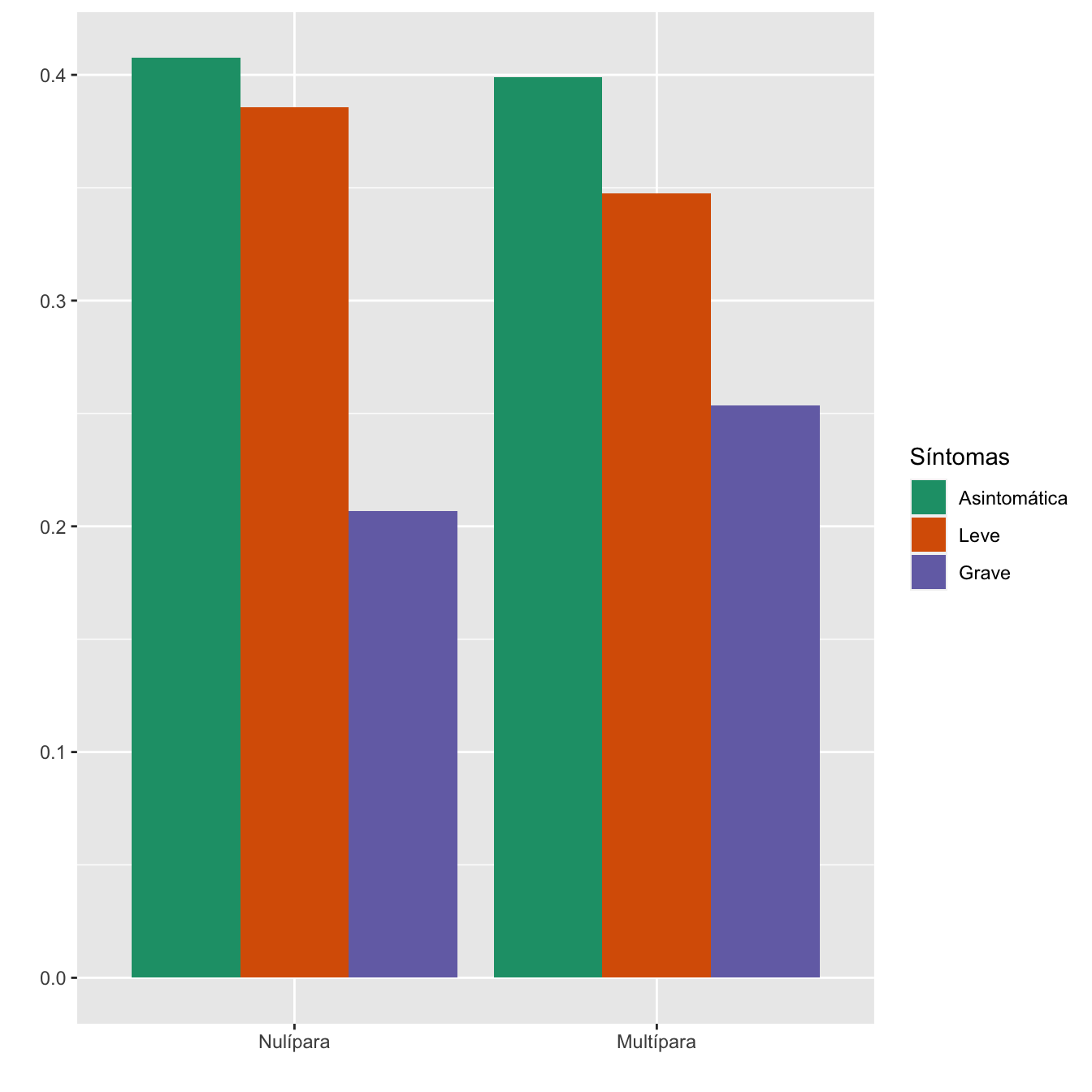
Figura 4.12:
- Potencia del test: 0.294
4.2 Desenlaces
4.2.1 Anomalía congénita
taula=table(data.frame(Casos$Diagnóstico.de.malformación.ecográfica._.semana.20._,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Diagnóstico.de.malformación.ecográfica._.semana.20._,"Anomalías congénitas", "No anomalías congénitas")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Anomalías congénitas | 6 | 1.7 | 6 | 1.9 | 2 | 1.1 | 0.742326 | Montecarlo |
| No anomalías congénitas | 342 | 98.3 | 305 | 98.1 | 188 | 98.9 | ||
| Datos perdidos | 9 | 3 | 16 |
Barplot.DGS(Casos$Diagnóstico.de.malformación.ecográfica._.semana.20._,"Anomalías congénitas", "No anomalías congénitas")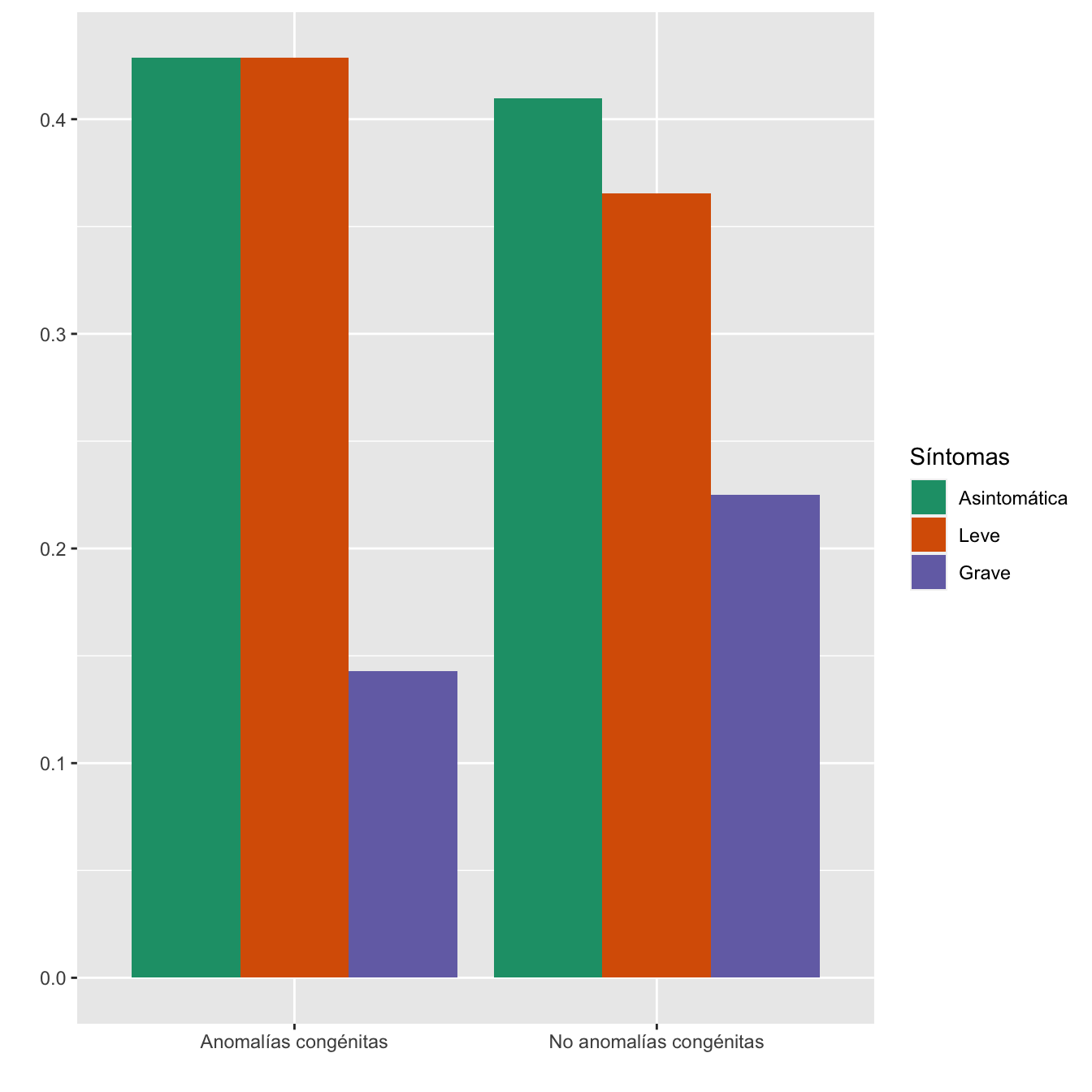
Figura 4.13:
- Potencia del test: 0.096
4.2.2 Retraso del crecimiento intrauterino
taula=table(data.frame(Casos$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Defecto.del.crecimiento.fetal..en.tercer.trimestre._.CIR._.,"RCIU", "No RCIU")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| RCIU | 16 | 4.5 | 10 | 3.2 | 6 | 2.9 | 0.544841 | Paramétrico |
| No RCIU | 341 | 95.5 | 304 | 96.8 | 200 | 97.1 | ||
| Datos perdidos | 0 | 0 | 0 |
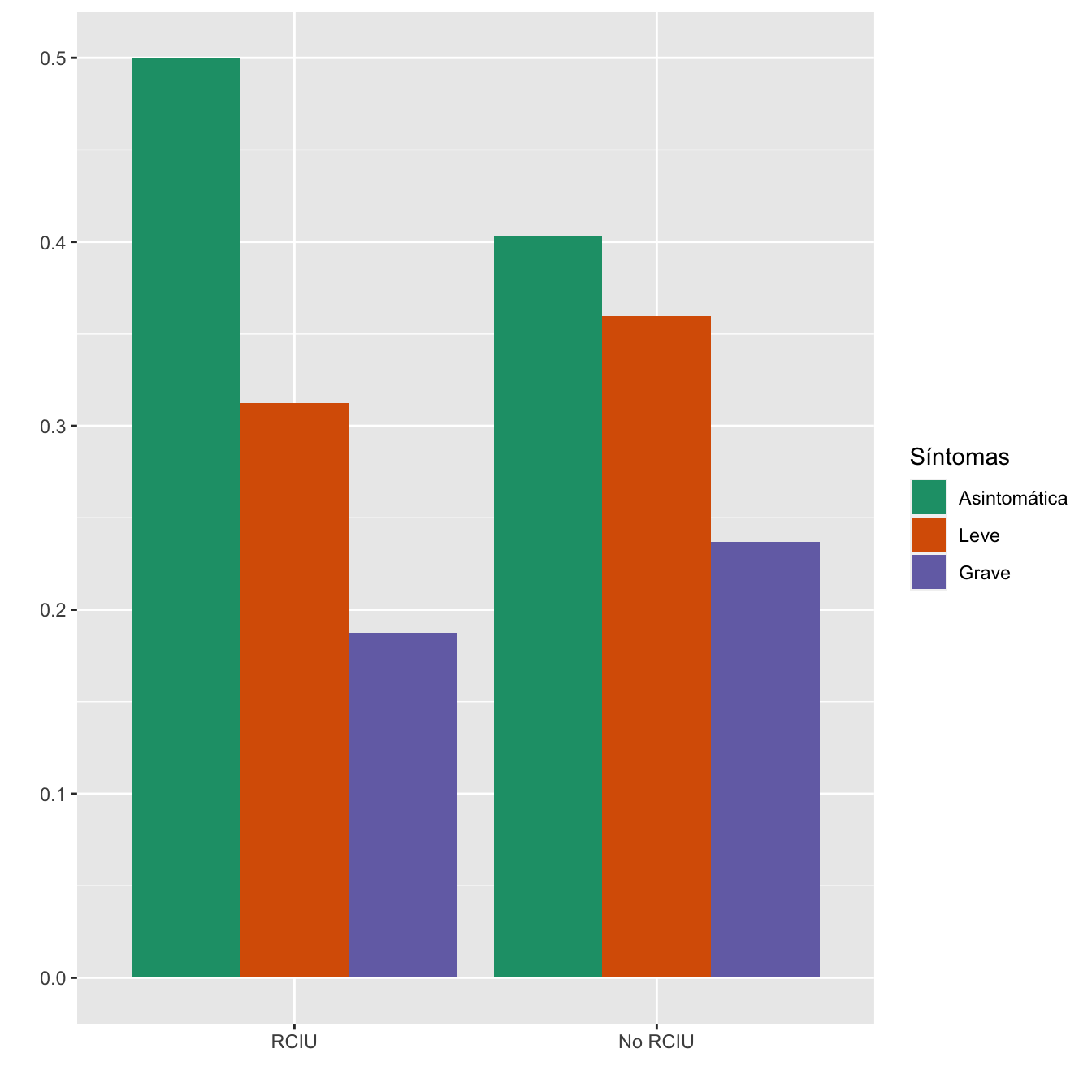
Figura 4.14:
- Potencia del test: 0.152
4.2.3 Diabetes gestacional
taula=table(data.frame(Casos$Diabetes.gestacional,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Diabetes.gestacional,"DG", "No DG")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| DG | 24 | 6.7 | 28 | 8.9 | 14 | 6.8 | 0.506116 | Paramétrico |
| No DG | 333 | 93.3 | 286 | 91.1 | 192 | 93.2 | ||
| Datos perdidos | 0 | 0 | 0 |
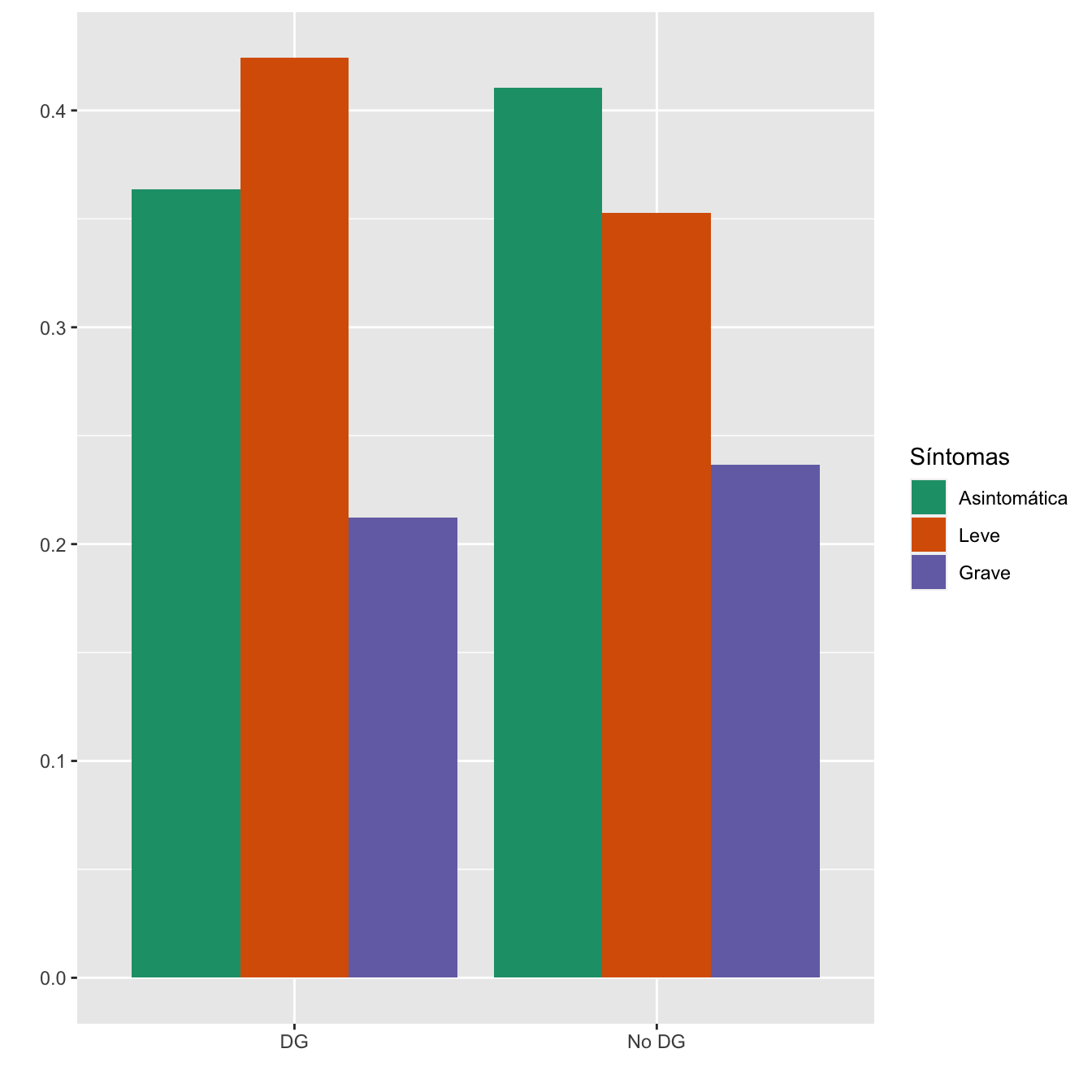
Figura 4.15:
- Potencia del test: 0.166
4.2.4 Hipertensión gestacional
taula=table(data.frame(Casos$Hipertensión.gestacional,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Hipertensión.gestacional,"HG", "No HG")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| HG | 9 | 2.5 | 8 | 2.5 | 3 | 1.5 | 0.684832 | Montecarlo |
| No HG | 348 | 97.5 | 306 | 97.5 | 203 | 98.5 | ||
| Datos perdidos | 0 | 0 | 0 |
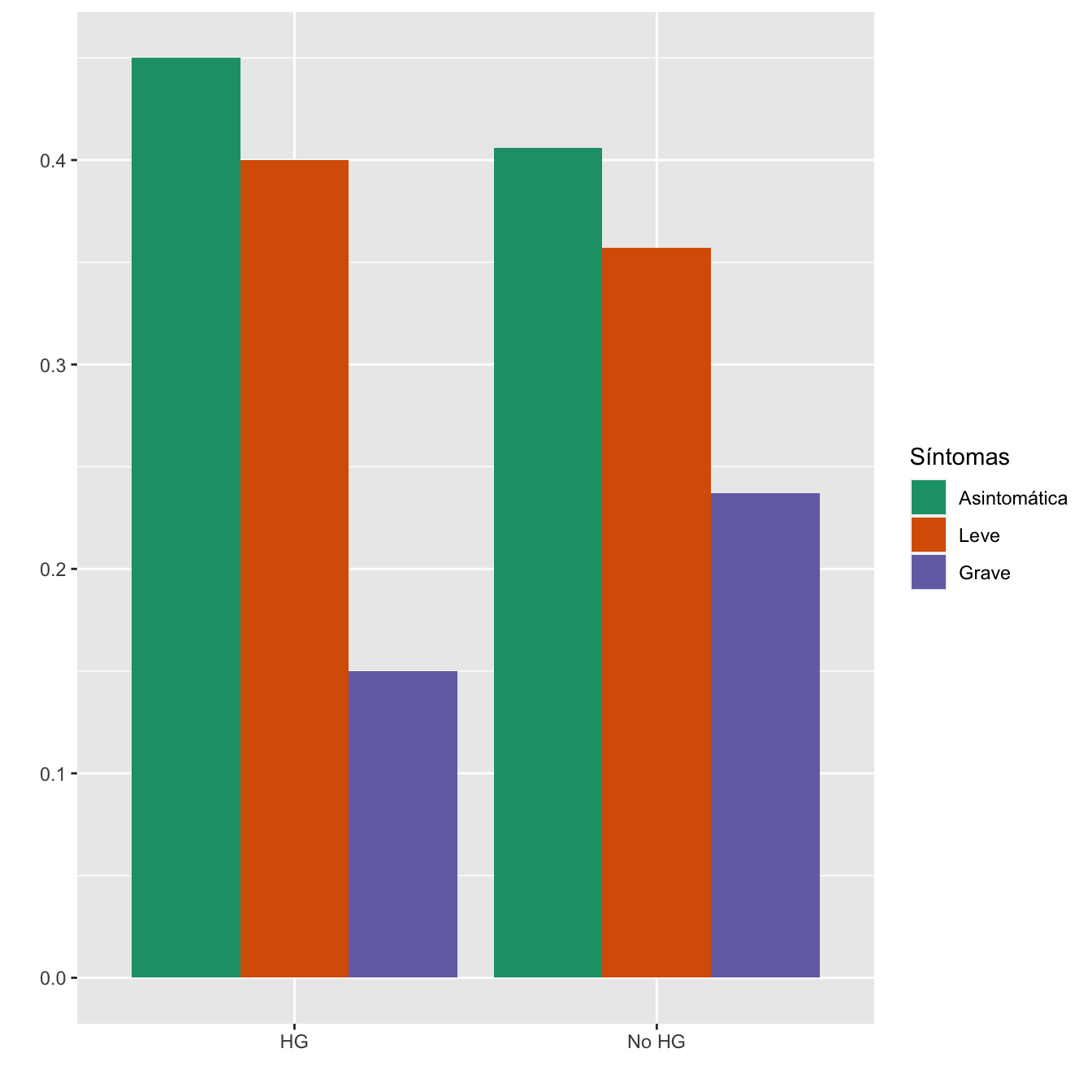
Figura 4.16:
- Potencia del test: 0.117
4.2.5 Rotura prematura de membranas
taula=table(data.frame(Casos$Bolsa.rota.anteparto,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$Bolsa.rota.anteparto,"RPM", "No RPM")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| RPM | 53 | 14.8 | 46 | 14.6 | 25 | 12.1 | 0.639141 | Paramétrico |
| No RPM | 304 | 85.2 | 268 | 85.4 | 181 | 87.9 | ||
| Datos perdidos | 0 | 0 | 0 |
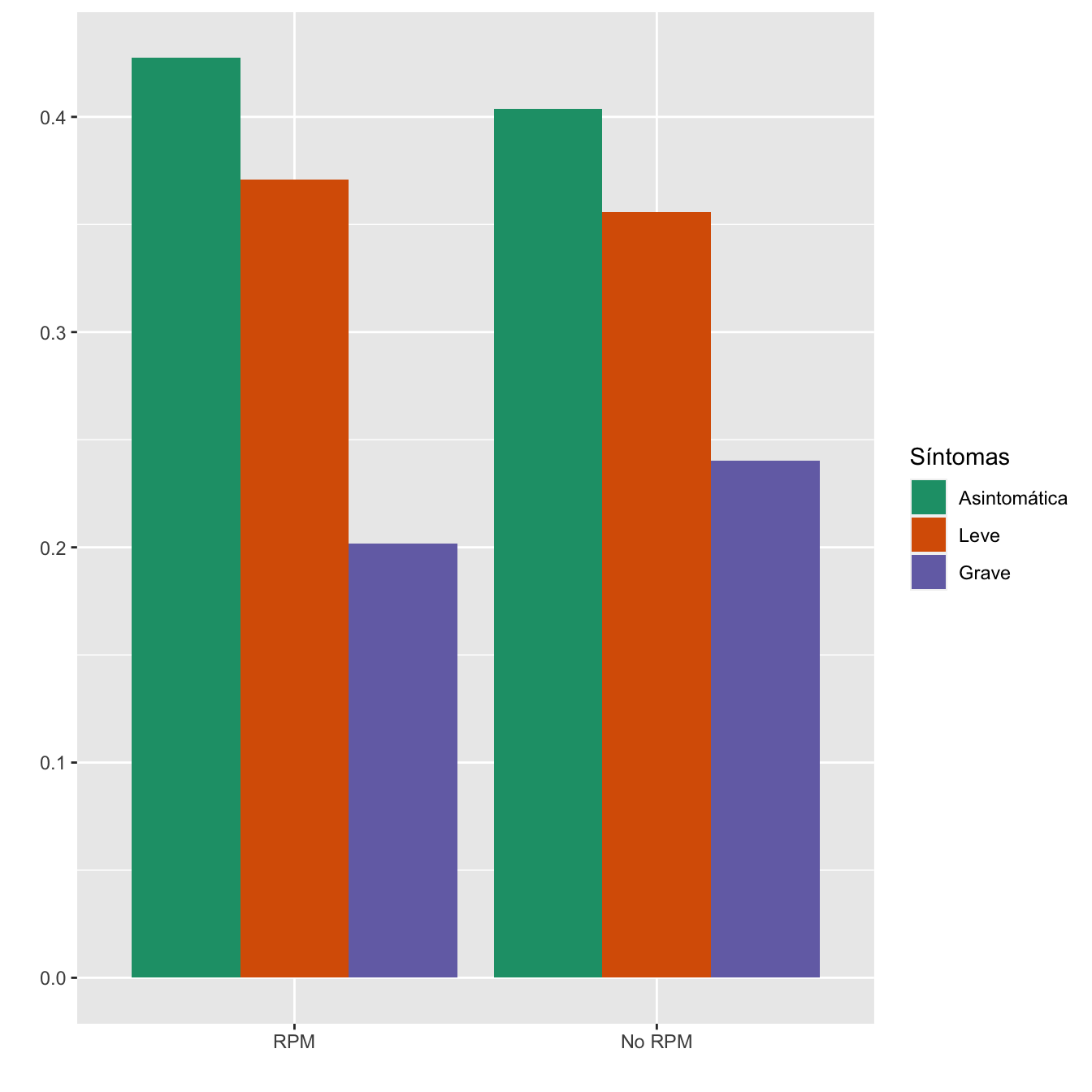
Figura 4.17:
- Potencia del test: 0.123
4.2.6 Preeclampsia
taula=table(data.frame(Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL,"PE", "No PE")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| PE | 19 | 5.3 | 16 | 5.1 | 11 | 5.3 | 0.989005 | Paramétrico |
| No PE | 338 | 94.7 | 298 | 94.9 | 195 | 94.7 | ||
| Datos perdidos | 0 | 0 | 0 |
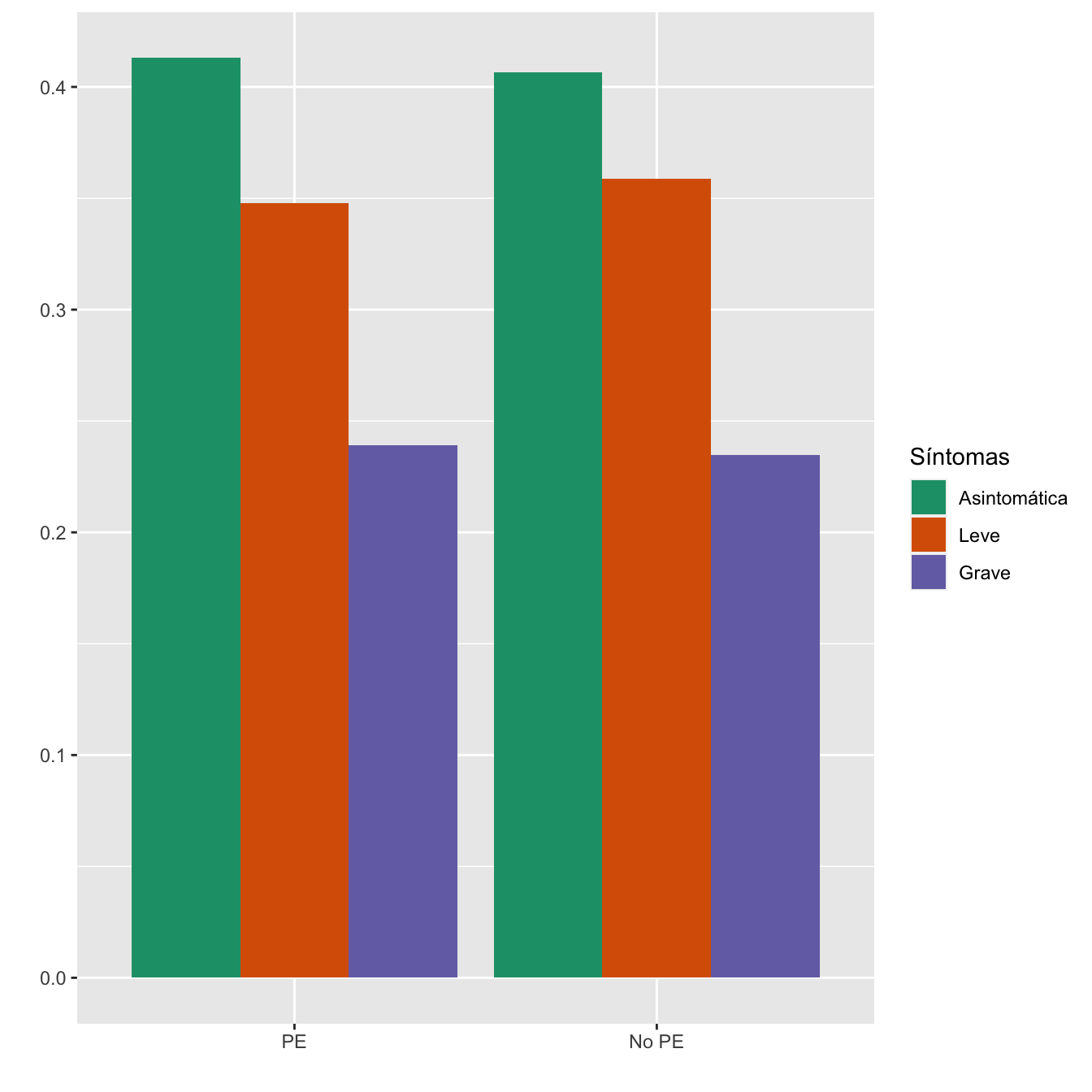
Figura 4.18:
- Potencia del test: 0.052
4.2.7 Preeclampsia con criterios de gravedad
I=Casos[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1,]$Preeclampsia.grave_HELLP_ECLAMPSIA
IA=Casos[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1 & Casos$SINTOMAS_DIAGNOSTICO=="Asintomática",]$Preeclampsia.grave_HELLP_ECLAMPSIA
IL=Casos[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1 & Casos$SINTOMAS_DIAGNOSTICO=="Leve",]$Preeclampsia.grave_HELLP_ECLAMPSIA
IG=Casos[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1 & Casos$SINTOMAS_DIAGNOSTICO=="Grave",]$Preeclampsia.grave_HELLP_ECLAMPSIA
taula=table(data.frame(I,Sint[Casos$PREECLAMPSIA_ECLAMPSIA_TOTAL==1]))
Tabla.DMCasosr(IA,IL,IG,"PE con CG", "PE sin CG")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| PE con CG | 7 | 36.8 | 6 | 37.5 | 6 | 54.5 | 0.636936 | Montecarlo |
| PE sin CG | 12 | 63.2 | 10 | 62.5 | 5 | 45.5 | ||
| Datos perdidos | 0 | 0 | 0 |
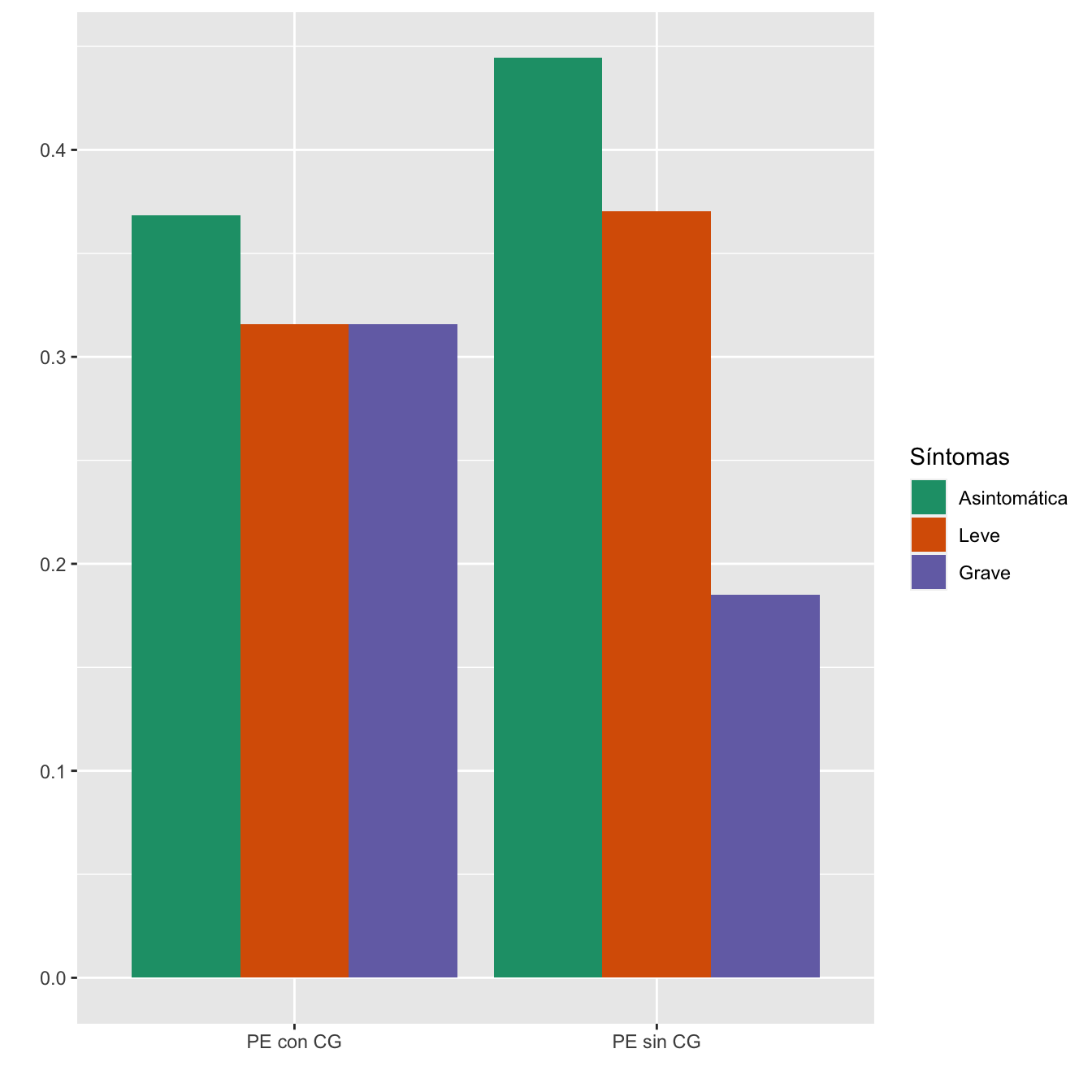
- Potencia del test: 0.137
4.2.8 Eventos hemorrágicos
taula=table(data.frame(Casos$EVENTOS_HEMORRAGICOS_TOTAL,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$EVENTOS_HEMORRAGICOS_TOTAL,"Eventos hemorrágicos", "No eventos hemorrágicos")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Eventos hemorrágicos | 17 | 4.8 | 16 | 5.1 | 18 | 8.7 | 0.120466 | Paramétrico |
| No eventos hemorrágicos | 340 | 95.2 | 298 | 94.9 | 188 | 91.3 | ||
| Datos perdidos | 0 | 0 | 0 |
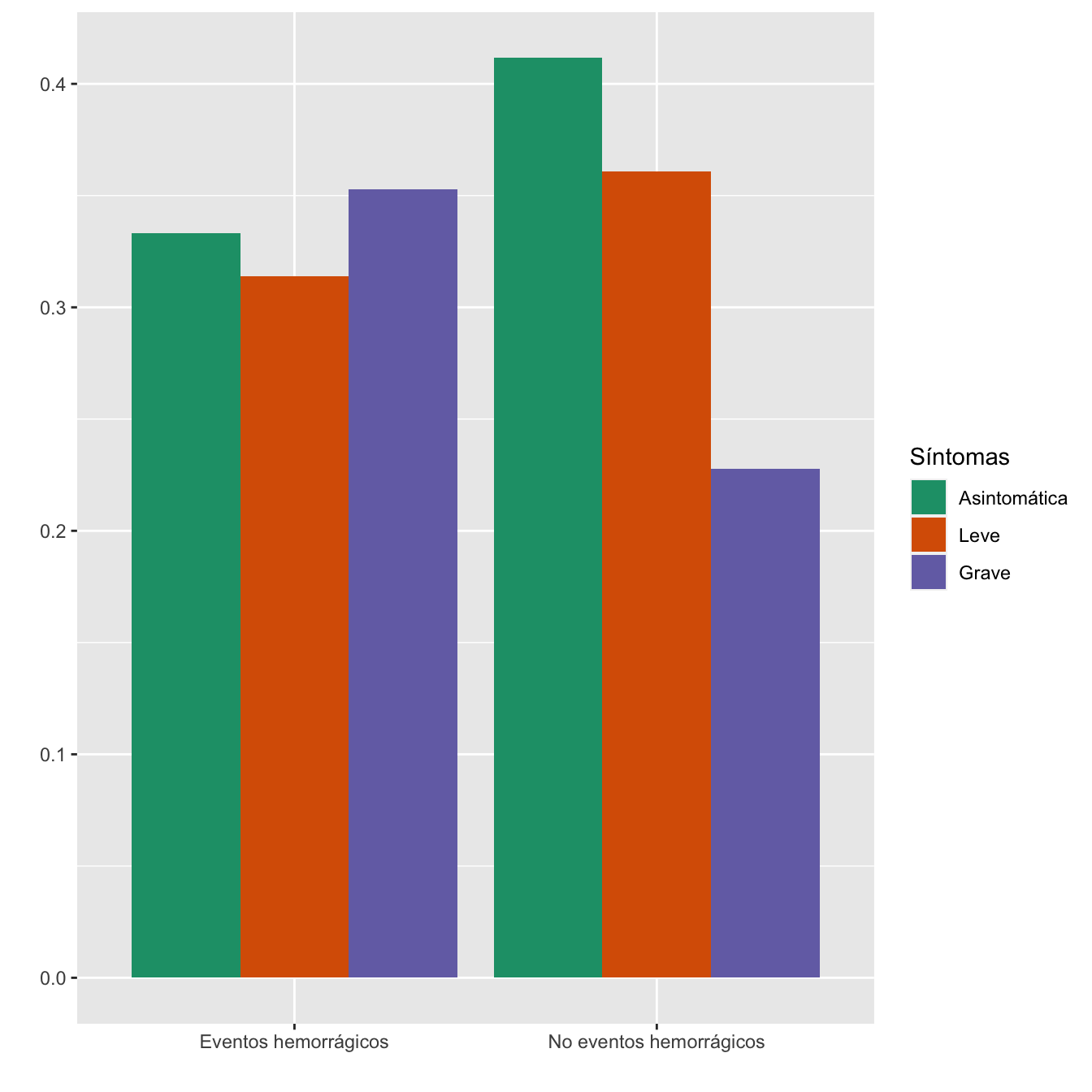
Figura 4.19:
Asociación entre los grupos de sintomatología y evento hemorrágico: test \(\chi^2\), p-valor 0.12
Test de diferencia en la tendencia de la gravedad respecto del global: p-valor 0.1573
Potencia del test: 0.437
4.2.9 Hemorragia postparto
I=Casos$Hemorragia.postparto
I.sino=I
I.sino[!is.na(I.sino)& I.sino!="No"]="Sí"
taula=table(data.frame(I.sino,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(I.sino,"HPP", "No HPP")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| HPP | 13 | 3.7 | 11 | 3.6 | 17 | 8.9 | 0.011451 | Paramétrico |
| No HPP | 341 | 96.3 | 298 | 96.4 | 175 | 91.1 | ||
| Datos perdidos | 3 | 5 | 14 |
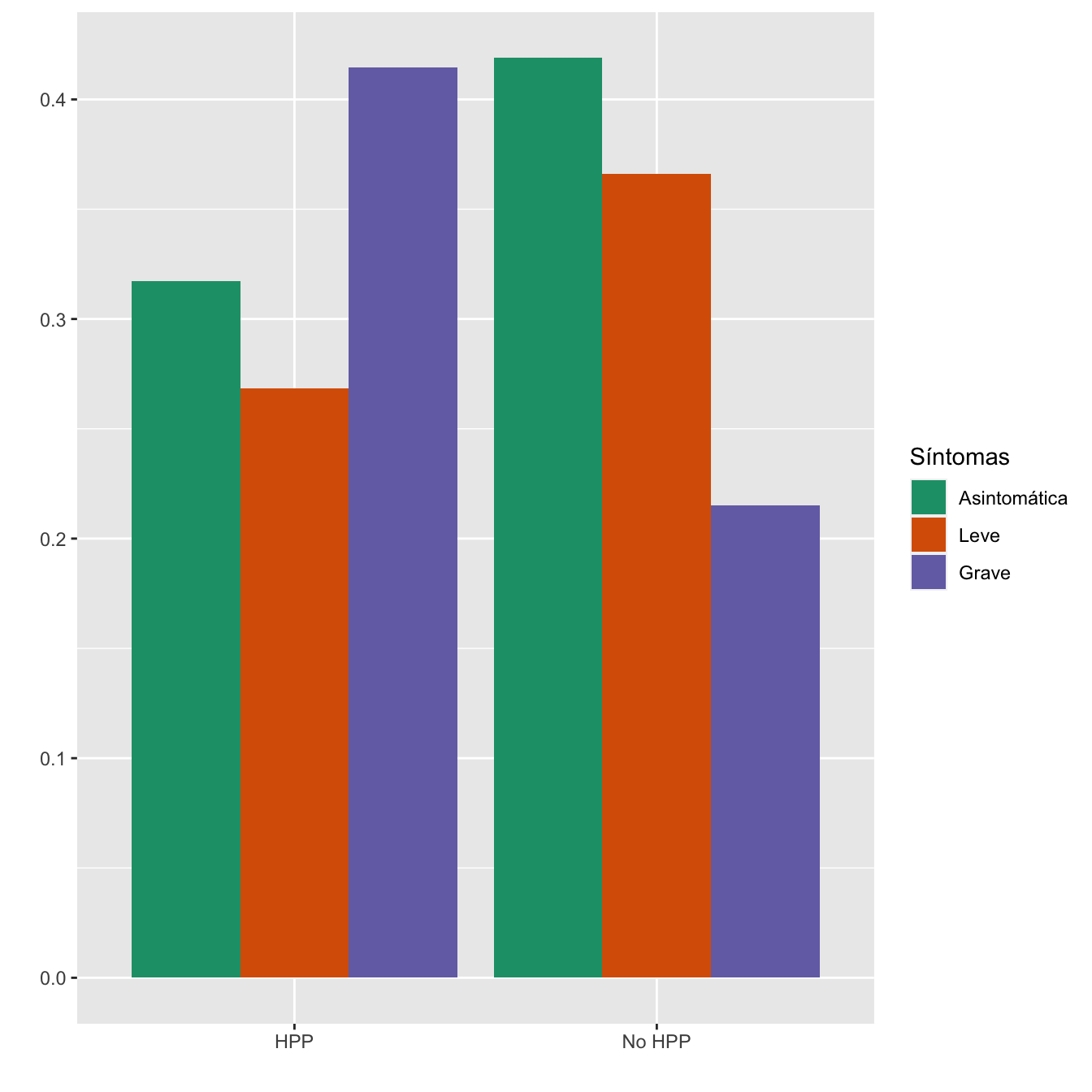
Figura 4.20:
- Potencia del test: 0.768
I=ordered(I,levels=names(table(I))[c(2,3,1,4)])
Tabla.DMCasosm(I,c("Tto. médico",
"Tto. Qx. conservador" , "Histerectomía",
"No") ,r=6)| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Tto. médico | 12 | 3.4 | 8 | 2.6 | 12 | 6.2 | 0.398160 | Paramétrico |
| Tto. Qx. conservador | 1 | 0.3 | 0 | 0.0 | 3 | 1.6 | 0.225977 | Montecarlo |
| Histerectomía | 0 | 0.0 | 3 | 1.0 | 2 | 1.0 | 0.810719 | Montecarlo |
| No | 341 | 96.3 | 298 | 96.4 | 175 | 91.1 | 0.045805 | Paramétrico |
| Datos perdidos | 3 | 5 | 14 |
DF=data.frame(Factor=I,Síntomas=Sint)
taula=table(DF)
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=4),levels=c("Asintomática", "Leve", "Grave"))
Grupo=ordered(rep(c("Tto. médico",
"Tto. Qx. conservador" , "Histerectomía" ,
"No") , 3),levels=c("Tto. médico",
"Tto. Qx. conservador" , "Histerectomía",
"No"))
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
ylab("")+
xlab("")+
scale_fill_brewer(palette = "Dark2") 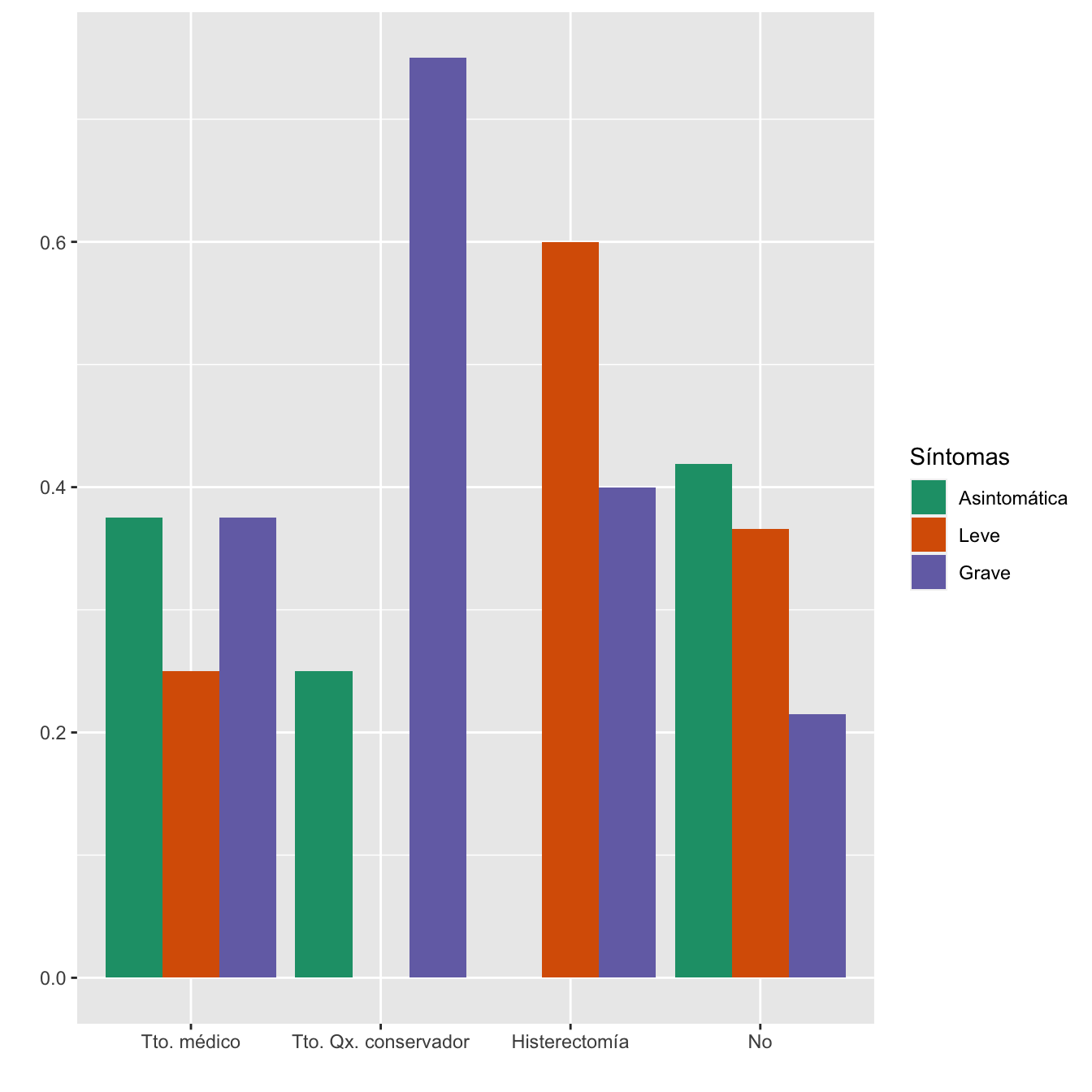
Figura 4.21:
Asociación entre los grupos de sintomatología y el tipo de hemorragia postparto: test \(\chi^2\) de Montecarlo, p-valor 0.172
Potencia del test: 0.495
4.2.10 Eventos trombóticos
taula=table(data.frame(Casos$EVENTOS_TROMBO_TOTALES,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$EVENTOS_TROMBO_TOTALES,"Eventos trombóticos", "No eventos trombóticos")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Eventos trombóticos | 0 | 0 | 0 | 0 | 7 | 3.4 | 1e-04 | Montecarlo |
| No eventos trombóticos | 357 | 100 | 314 | 100 | 199 | 96.6 | ||
| Datos perdidos | 0 | 0 | 0 |
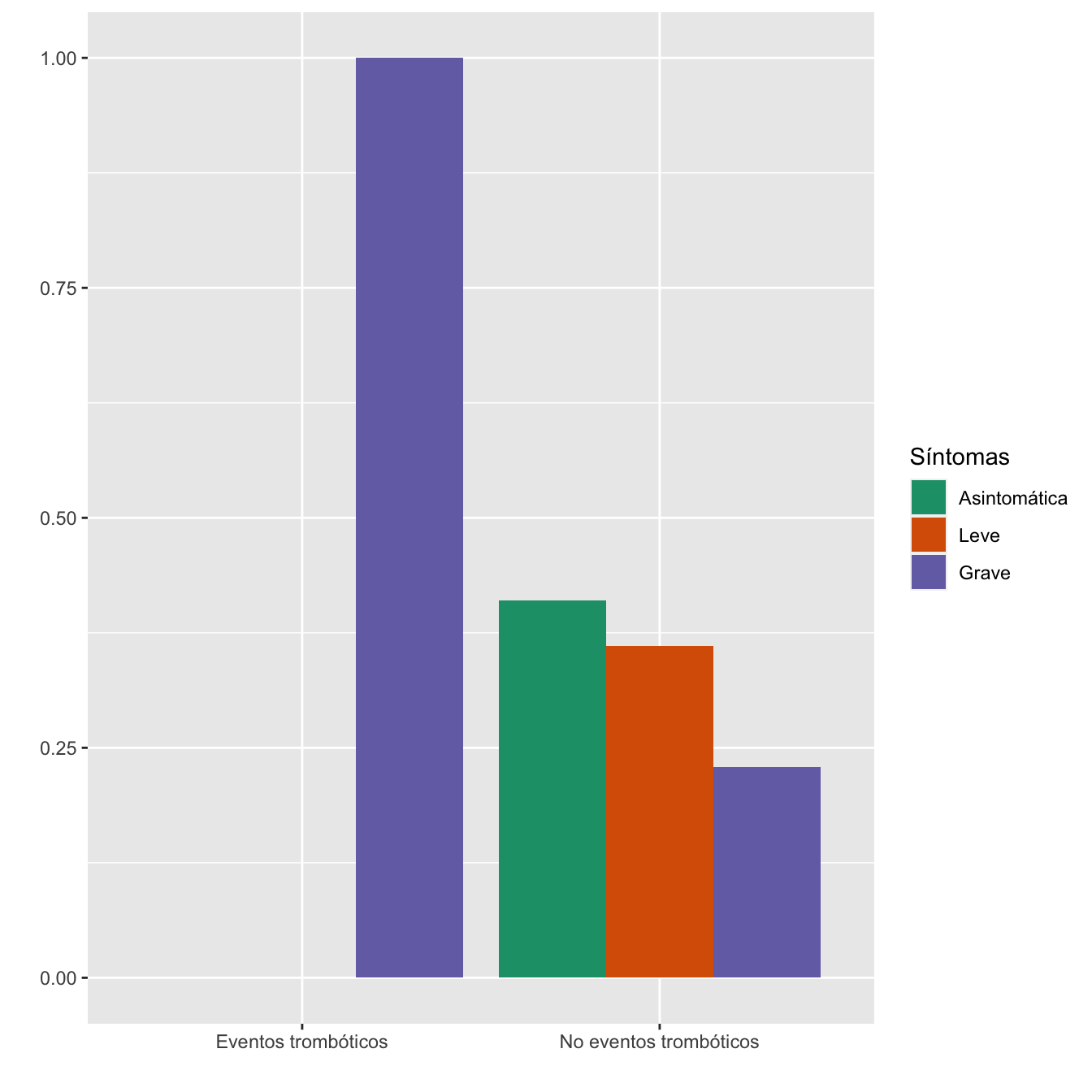
Figura 4.22:
- Potencia del test: 0.994
4.2.11 UCI materna (Sí-No)
| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| UCI | 2 | 0.6 | 5 | 1.6 | 17 | 8.3 | 0 | Paramétrico |
| No UCI | 355 | 99.4 | 309 | 98.4 | 189 | 91.7 | ||
| Datos perdidos | 0 | 0 | 0 |
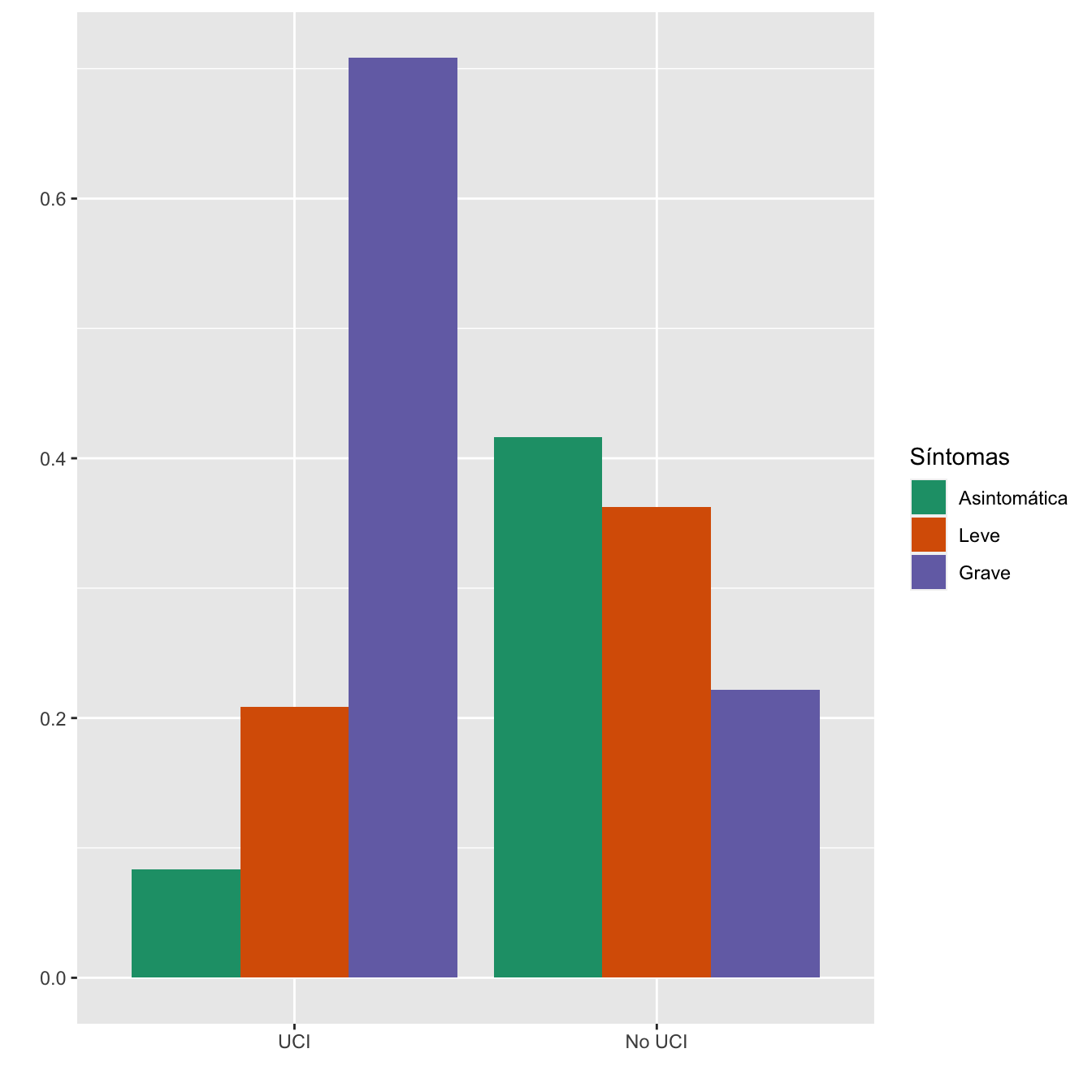
Figura 4.23:
- Potencia del test: 1
4.2.12 UCI materna (antes-después)
I=Casos$UCI_ANTES.DESPUES.DEL.PARTO[Casos$UCI==1]
IA=Casos[Casos$UCI==1 & Casos$SINTOMAS_DIAGNOSTICO=="Asintomática",]$UCI_ANTES.DESPUES.DEL.PARTO
IA=factor(IA,levels=c("ANTES DEL PARTO","DESPUES DEL PARTO"))
IL=Casos[Casos$UCI==1 & Casos$SINTOMAS_DIAGNOSTICO=="Leve",]$UCI_ANTES.DESPUES.DEL.PARTO
IL=factor(IL,levels=c("ANTES DEL PARTO","DESPUES DEL PARTO"))
IG=Casos[Casos$UCI==1 & Casos$SINTOMAS_DIAGNOSTICO=="Grave",]$UCI_ANTES.DESPUES.DEL.PARTO
IG=factor(IG,levels=c("ANTES DEL PARTO","DESPUES DEL PARTO"))
taula=table(data.frame(I,Sint[Casos$UCI==1]))
Tabla.DMCasosr(IA,IL,IG,"UCI antes del parto", "UCI después del parto")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| UCI antes del parto | 2 | 100 | 5 | 100 | 13 | 76.5 | 0.548745 | Montecarlo |
| UCI después del parto | 0 | 0 | 0 | 0 | 4 | 23.5 | ||
| Datos perdidos | 0 | 0 | 0 |
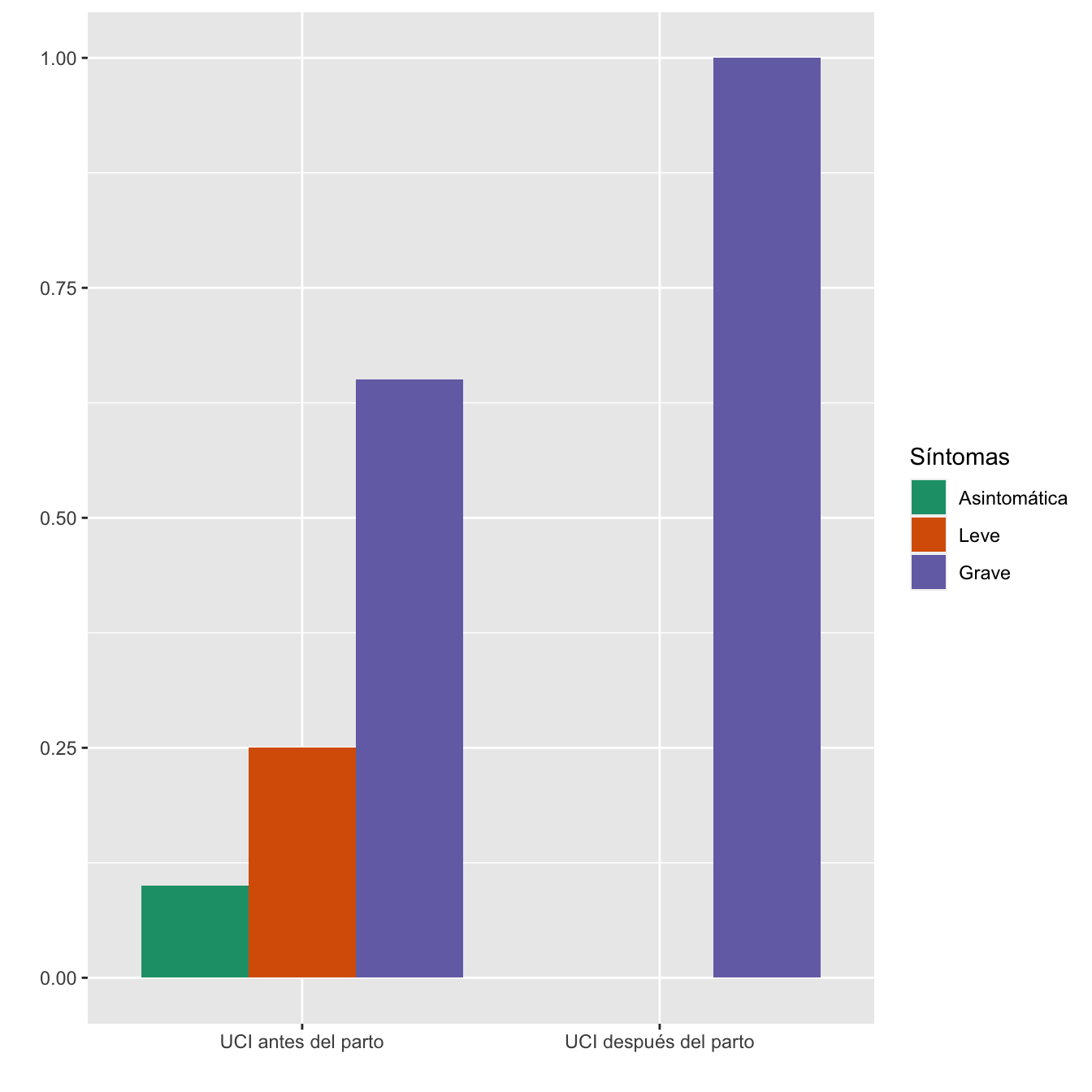
- Potencia del test: 0.223
4.2.13 Prematuridad
taula=table(data.frame(Casos$PREMATURO,Sint))[c(2,1) ,1:3]
Tabla.DMCasos(Casos$PREMATURO,"Prematuro", "A término")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Prematuro | 28 | 7.8 | 37 | 11.8 | 39 | 18.9 | 0.00046 | Paramétrico |
| A término | 329 | 92.2 | 277 | 88.2 | 167 | 81.1 | ||
| Datos perdidos | 0 | 0 | 0 |
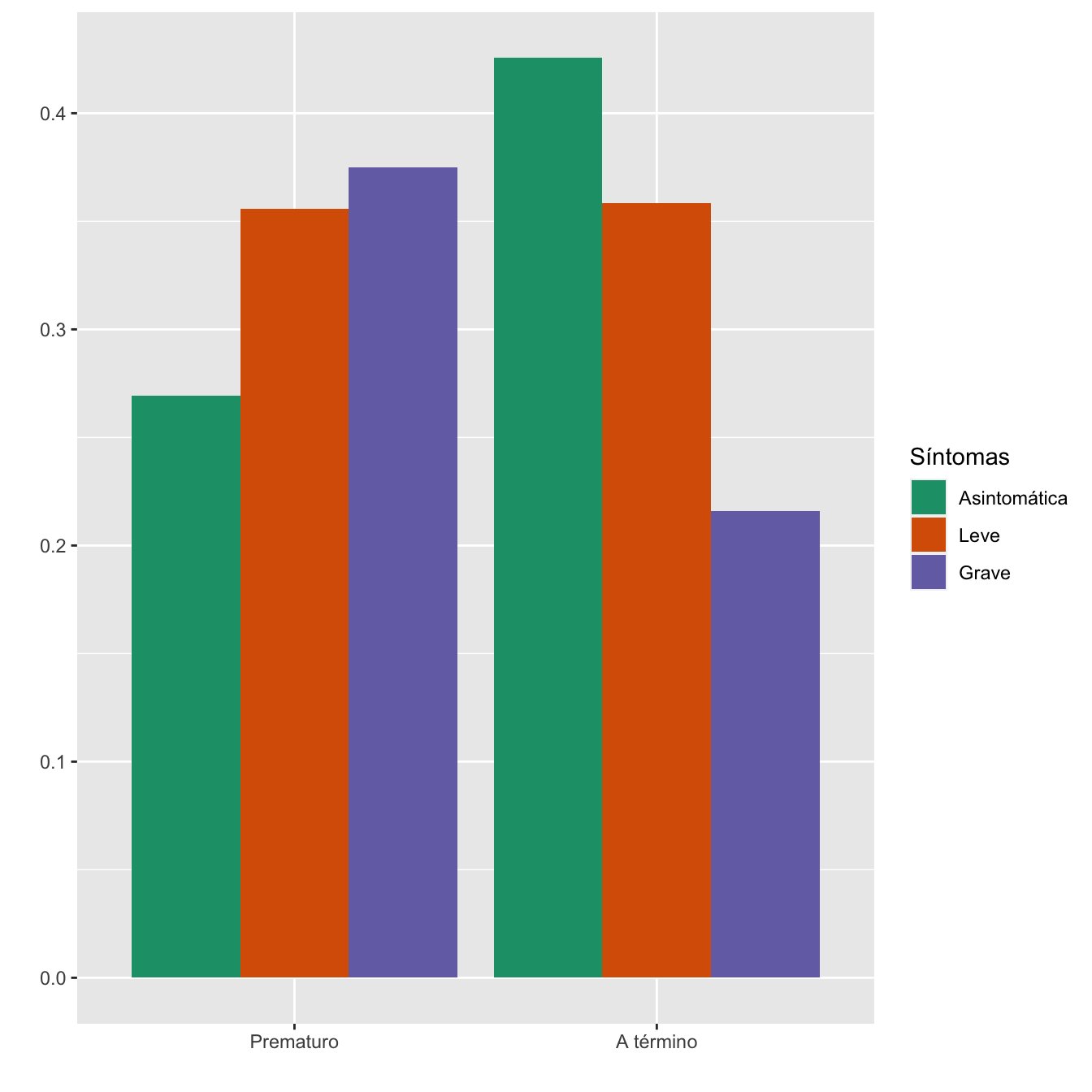
Figura 4.24:
- Potencia del test: 0.949
4.2.14 Inicio de parto
I=Casos$Inicio.de.parto
I=factor(I,levels=c("Espontáneo", "Inducido", "Cesárea"),ordered=TRUE)
DF=data.frame(Factor=I,Síntomas=Sint)
taula=table(DF)
Tabla.DMCasosm(I,c("Espontáneo", "Inducido","Cesárea programada"))| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Espontáneo | 200 | 56.0 | 158 | 50.5 | 84 | 41.0 | 0.008240 | Paramétrico |
| Inducido | 127 | 35.6 | 117 | 37.4 | 81 | 39.5 | 1.000000 | Paramétrico |
| Cesárea programada | 30 | 8.4 | 38 | 12.1 | 40 | 19.5 | 0.001769 | Paramétrico |
| Datos perdidos | 0 | 1 | 1 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=3),levels=c("Asintomática", "Leve", "Grave"))
Grupo=ordered(rep(rownames(EEExt)[1:3] , 3),levels=rownames(EEExt)[1:3])
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
ylab("")+
xlab("Inicio de parto")+
scale_fill_brewer(palette = "Dark2") 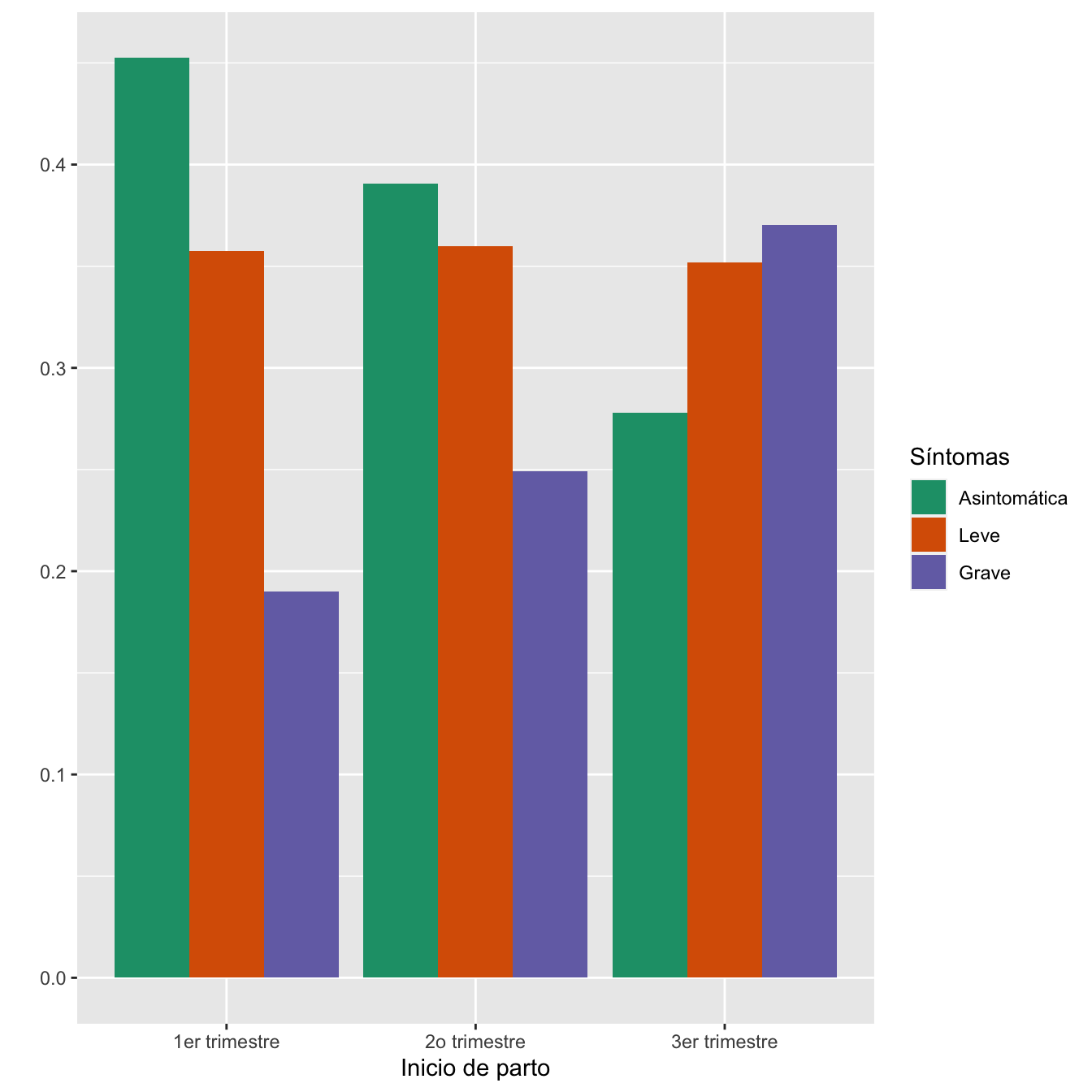
Figura 4.25:
- Asociación entre los grupos de sintomatología y el inicio de parto: test \(\chi^2\), p-valor \(6\times 10^{-4}\)
4.2.15 Tipo de parto
I.In=Casos$Inicio.de.parto
I.In[I.In=="Cesárea"]="Cesárea programada"
I=Casos$Tipo.de.parto
I[I.In=="Cesárea programada"]="Cesárea programada"
I[I=="Cesárea"]="Cesárea urgente"
I=factor(I,levels=c("Eutocico", "Instrumental", "Cesárea programada", "Cesárea urgente"),ordered=TRUE)
DF=data.frame(Factor=I,Síntomas=Sint)
taula=table(DF)
Tabla.DMCasosm(I,c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"))| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Eutócico | 233 | 65.3 | 184 | 58.6 | 111 | 53.9 | 0.089891 | Paramétrico |
| Instrumental | 45 | 12.6 | 39 | 12.4 | 16 | 7.8 | 0.685373 | Paramétrico |
| Cesárea programada | 30 | 8.4 | 38 | 12.1 | 40 | 19.4 | 0.002576 | Paramétrico |
| Cesárea urgente | 49 | 13.7 | 53 | 16.9 | 39 | 18.9 | 0.958557 | Paramétrico |
| Datos perdidos | 0 | 0 | 0 |
Síntomas=ordered(rep(c("Asintomática", "Leve", "Grave"), each=4),levels=c("Asintomática", "Leve", "Grave"))
Grupo=ordered(rep(c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente") , 3),levels=c("Eutócico", "Instrumental", "Cesárea programada", "Cesárea urgente"))
valor=as.vector(prop.table(taula, margin=1))
data <- data.frame(Grupo,Síntomas,valor)
ggplot(data, aes(fill=Síntomas, y=valor, x=Grupo)) +
geom_bar(position="dodge", stat="identity")+
ylab("")+
xlab("Tipo de parto")+
scale_fill_brewer(palette = "Dark2") 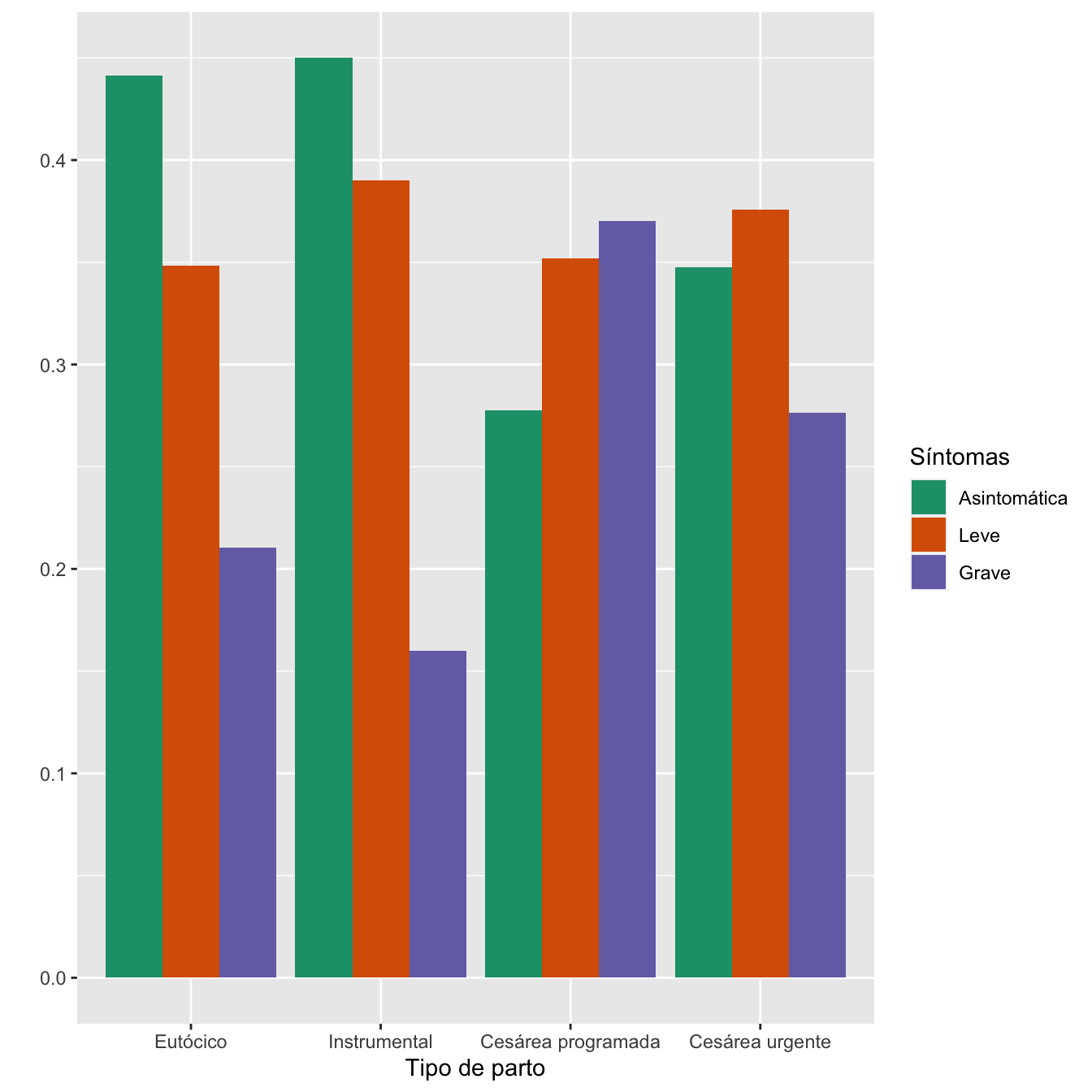
Figura 4.26:
Asociación entre los grupos de sintomatología y tipos de parto: test \(\chi^2\), p-valor \(0.002\)
Asociación entre los grupos de sintomatología y tipos de parto diferentes de la cesárea programada: test \(\chi^2\), p-valor 0.139
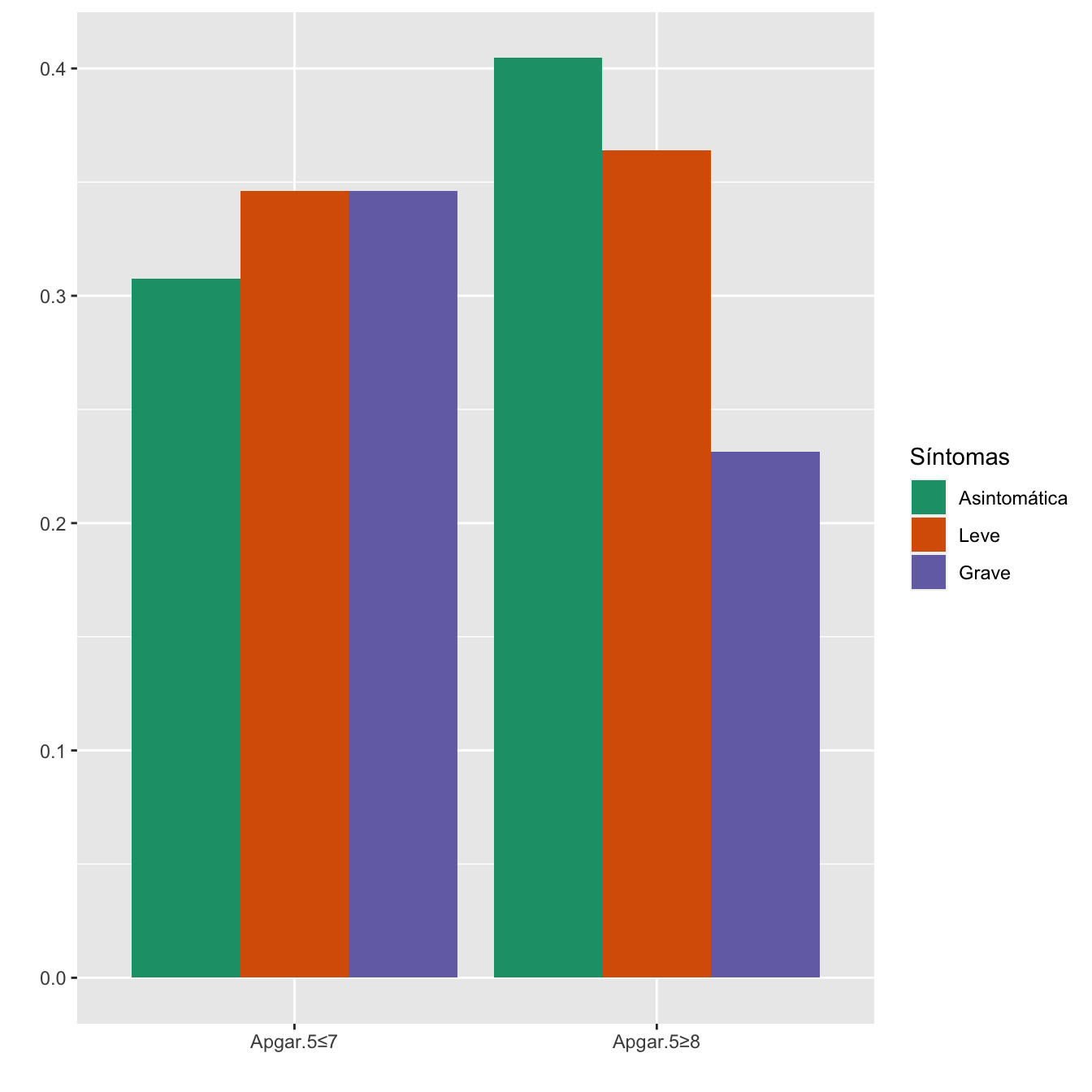
4.2.16 APGAR
I=c(Casos$APGAR.5...126,CasosGM$APGAR.5...150)
I[I==19]=NA
I=cut(I,breaks=c(-1,7,20),labels=c("0-7","8-10"))
I=ordered(I,levels=c("8-10","0-7"))
SintN=c(Casos$SINTOMAS_DIAGNOSTICO,CasosGM$SINTOMAS_DIAGNOSTICO)
taula=table(data.frame(I,SintN))[c(2,1) ,1:3]
Tabla.DMCasos(I,"Apgar.5≤7","Apgar.5≥8")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Apgar.5≤7 | 8 | 2.2 | 9 | 2.8 | 9 | 4.4 | 0.335268 | Paramétrico |
| Apgar.5≥8 | 352 | 97.8 | 312 | 97.2 | 196 | 95.6 | ||
| Datos perdidos | 3 | 4 | 2 |
- Potencia del test: 0.228
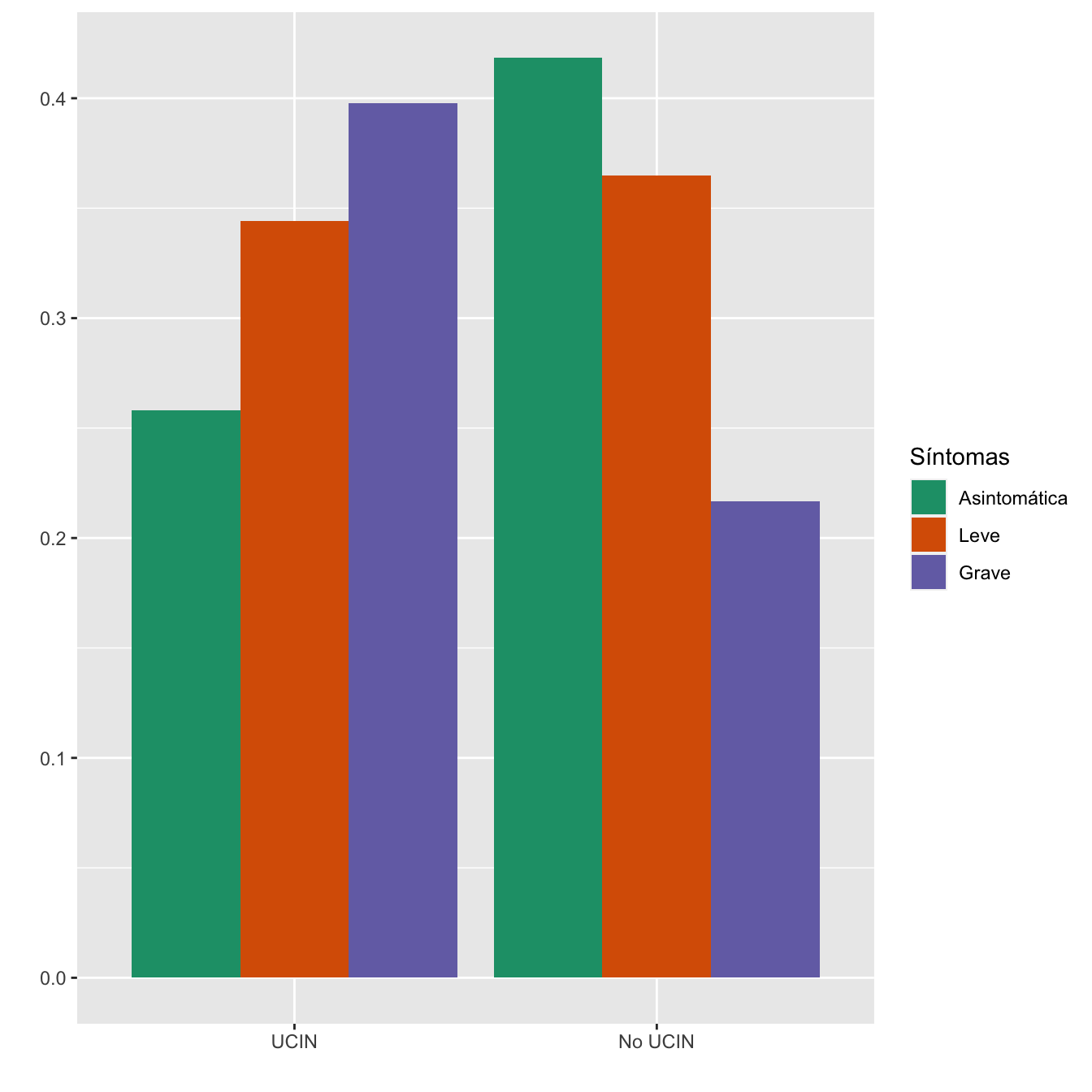
4.2.17 Sintomatología vs UCI neonato
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,CasosGM[CasosGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
IA=c(Casos[Casos$SINTOMAS_DIAGNOSTICO=="Asintomática"&Casos$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,CasosGM[CasosGM$SINTOMAS_DIAGNOSTICO=="Asintomática" & CasosGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
IL=c(Casos[Casos$SINTOMAS_DIAGNOSTICO=="Leve"&Casos$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,CasosGM[CasosGM$SINTOMAS_DIAGNOSTICO=="Leve" & CasosGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
IG=c(Casos[Casos$SINTOMAS_DIAGNOSTICO=="Grave"&Casos$Feto.muerto.intraútero=="No",]$Ingreso.en.UCIN,CasosGM[CasosGM$SINTOMAS_DIAGNOSTICO=="Grave" & CasosGM$Feto.vivo=="Sí",]$Ingreso.en.UCI)
SintN=c(Casos[Casos$Feto.muerto.intraútero=="No",]$SINTOMAS_DIAGNOSTICO,CasosGM[CasosGM$Feto.vivo=="Sí",]$SINTOMAS_DIAGNOSTICO)
taula=table(data.frame(I,SintN))[c(2,1) ,1:3]
Tabla.DMCasosr(IA,IL,IG,"UCIN", "No UCIN")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| UCIN | 24 | 6.8 | 32 | 10.1 | 37 | 17.9 | 0.000207 | Paramétrico |
| No UCIN | 328 | 93.2 | 286 | 89.9 | 170 | 82.1 | ||
| Datos perdidos | 3 | 4 | 2 |
- Potencia del test: 0.966
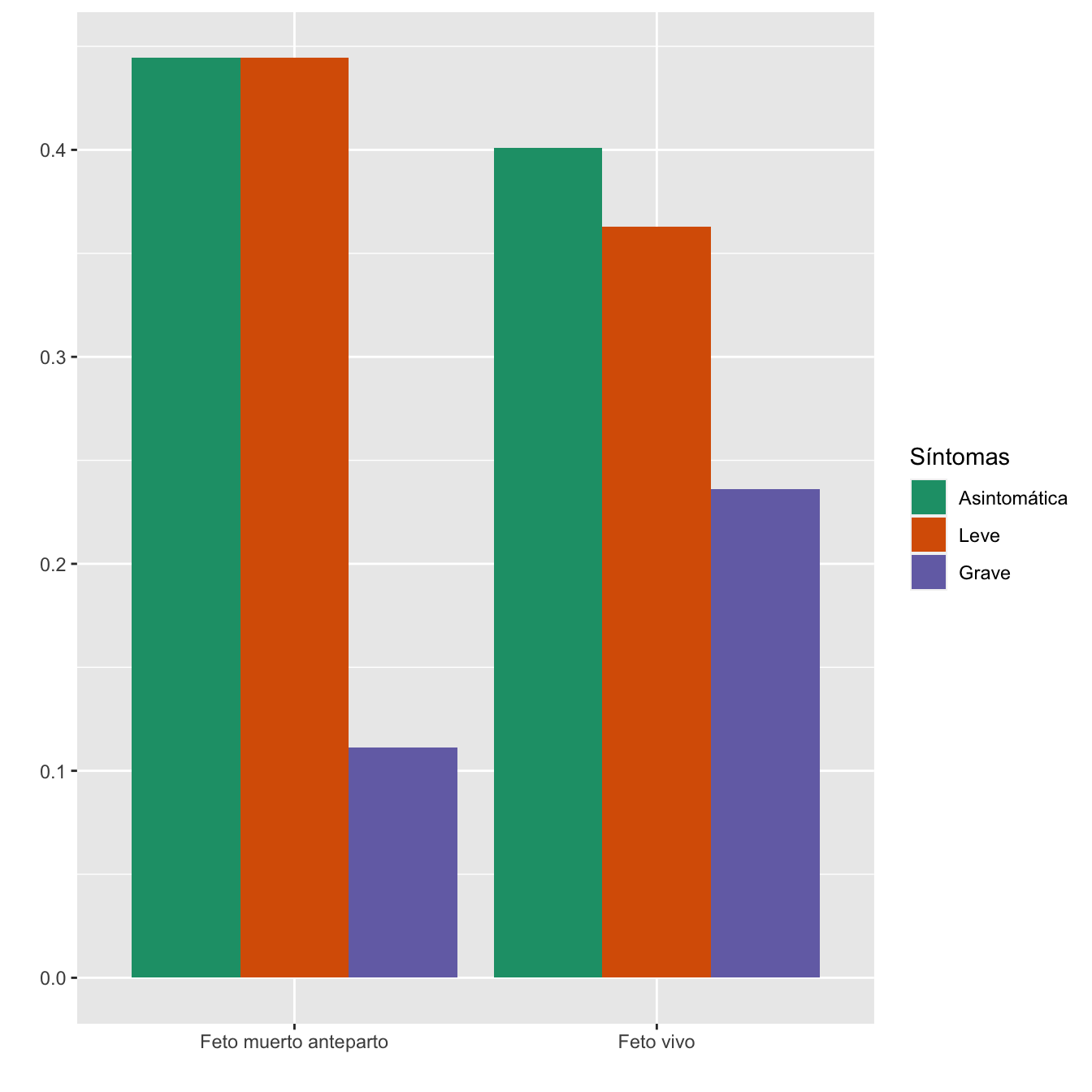
4.2.18 Sintomatología vs Fetos muertos anteparto
I1=Casos$Feto.muerto.intraútero
I1[I1=="Sí"]="Muerto"
I1[I1=="No"]="Vivo"
I1=ordered(I1,levels=c("Vivo","Muerto"))
I1A=I1[Casos$SINTOMAS_DIAGNOSTICO=="Asintomática"]
I1L=I1[Casos$SINTOMAS_DIAGNOSTICO=="Leve"]
I1G=I1[Casos$SINTOMAS_DIAGNOSTICO=="Grave"]
I2=CasosGM$Feto.vivo
I2[I2=="Sí"]="Vivo"
I2[I2=="No"]="Muerto"
I2=ordered(I2,levels=c("Vivo","Muerto"))
I2A=I2[CasosGM$SINTOMAS_DIAGNOSTICO=="Asintomática"]
I2L=I2[CasosGM$SINTOMAS_DIAGNOSTICO=="Leve"]
I2G=I2[CasosGM$SINTOMAS_DIAGNOSTICO=="Grave"]
IA=c(I1A,I2A)
IL=c(I1L,I2L)
IG=c(I1G,I2G)
I=c(I1,I2)
SintN1=Casos$SINTOMAS_DIAGNOSTICO
SintN2=CasosGM$SINTOMAS_DIAGNOSTICO
SintN=c(SintN1,SintN2)
taula=table(data.frame(I,SintN))[c(2,1) ,1:3]
Tabla.DMCasosr(IA,IL,IG,"Feto muerto anteparto", "Feto vivo")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Feto muerto anteparto | 4 | 1.1 | 4 | 1.2 | 1 | 0.5 | 0.765023 | Montecarlo |
| Feto vivo | 355 | 98.9 | 321 | 98.8 | 209 | 99.5 | ||
| Datos perdidos | 0 | 1 | 0 |
- Potencia del test: 0.115
4.2.19 Edades gestacionales
I=Casos$EG_TOTAL_PARTO
Síntomas=ordered(Sint,levels=c("Asintomática" ,"Leve", "Grave" ))
data =data.frame(
Síntomas,
Edades=I
)
data %>%
ggplot( aes(x=Síntomas, y=Edades, fill=Síntomas)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title = element_text(size=11)
) +
xlab("")+
ylab("Edades gestacionales (semanas)")+
scale_fill_brewer(palette = "Dark2") 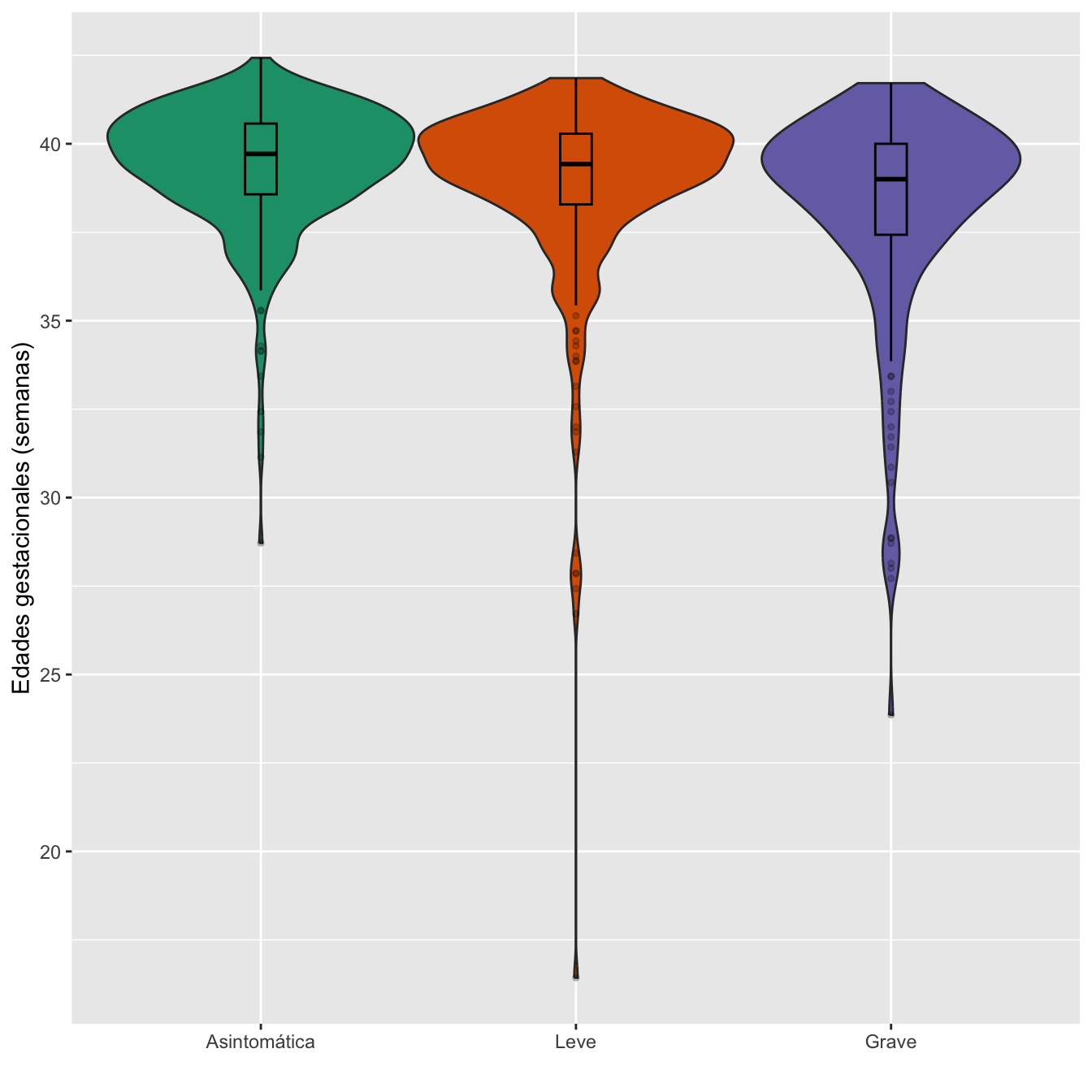
Figura 4.27:
Dades=rbind(c(round(min(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),
round(median(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Asintomática"],c(0.25,0.75),na.rm=TRUE),1),
round(sd(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1)),
c(round(min(I[data$Síntomas=="Leve"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Leve"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Leve"],na.rm=TRUE),1),
round(median(I[data$Síntomas=="Leve"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Leve"],c(0.25,0.75),na.rm=TRUE),1),round(sd(I[data$Síntomas=="Leve"],na.rm=TRUE),1)),
c(round(min(I[data$Síntomas=="Grave"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Grave"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Grave"],na.rm=TRUE),1),round(median(I[data$Síntomas=="Grave"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Grave"],c(0.25,0.75),na.rm=TRUE),1),round(sd(I[data$Síntomas=="Grave"],na.rm=TRUE),1)) )
colnames(Dades)=c("Edad gest. mínima","Edad gest. máxima","Edad gest. media",
"Edad gest. mediana", "1er cuartil", "3er cuartil","Desv. típica")
rownames(Dades)=c("Asintomática" ,"Leve", "Grave" )
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Edad gest. mínima | Edad gest. máxima | Edad gest. media | Edad gest. mediana | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Asintomática | 28.7 | 42.4 | 39.4 | 39.7 | 38.6 | 40.6 | 1.8 |
| Leve | 16.4 | 41.9 | 38.9 | 39.4 | 38.3 | 40.3 | 2.6 |
| Grave | 23.9 | 41.7 | 38.2 | 39.0 | 37.4 | 40.0 | 3.0 |
Ajuste de las edades gestacionales de cada nivel a distribuciones normales: test de Shapiro-Wilks, p-valores \(4\times 10^{-16}\), \(3\times 10^{-23}\), \(2\times 10^{-15}\), respectivamente
Homocedasticidad: Test de Fligner-Killeen, p-valor \(0.001\)
Edades gestacionales medias: test de Kruskal-Wallis, p-valor \(2\times 10^{-6}\)
Contrastes posteriores de edades gestacionales medias por parejas: tests de Mann-Whitney,p-valores ajustados por Bonferroni:
- Asintomática vs Leve: 0.029
- Asintomática vs Grave: \(10^{-6}\)
- Leve vs Grave: \(0.01\)
4.2.20 Pesos hijos infectadas
SintN=c(Casos[Casos$Feto.muerto.intraútero=="No",]$SINTOMAS_DIAGNOSTICO,CasosGM[CasosGM$Feto.vivo=="Sí",]$SINTOMAS_DIAGNOSTICO)
Síntomas=SintN[!is.na(SintN)]
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí",]$Peso._gramos_...148)
I=I[!is.na(SintN)]
data =data.frame(
Síntomas,
Pesos=I
)
data %>%
ggplot( aes(x=Síntomas, y=Pesos, fill=Síntomas)) +
geom_violin(width=1) +
geom_boxplot(width=0.1, color="black", alpha=0.2,outlier.fill="black",
outlier.size=1) +
theme(
legend.position="none",
plot.title = element_text(size=11)
) +
xlab("")+
ylab("Pesos (gramos)")+
scale_fill_brewer(palette = "Dark2") 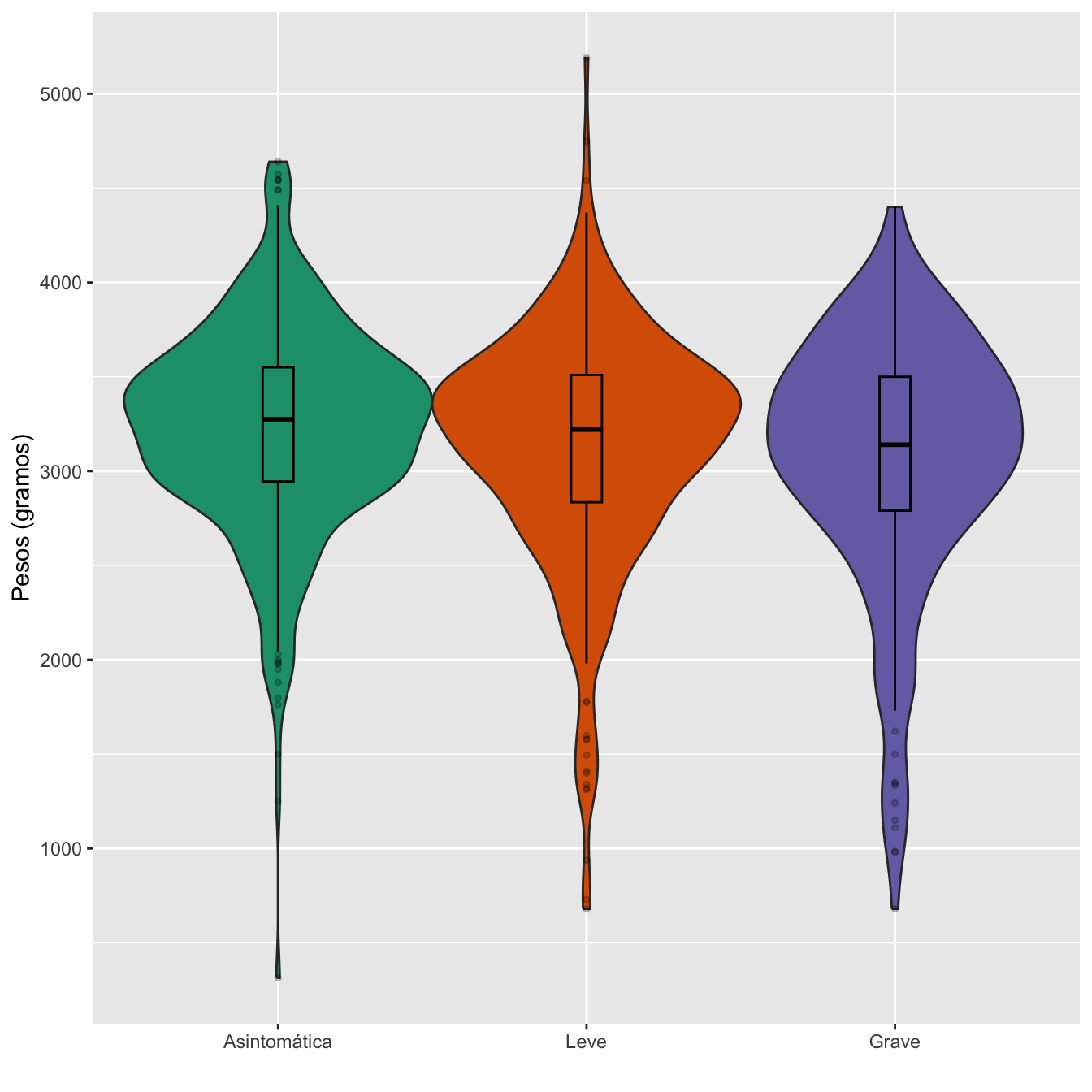
Figura 4.28:
Dades=rbind(c(round(min(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),
round(median(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Asintomática"],c(0.25,0.75),na.rm=TRUE),1),
round(sd(I[data$Síntomas=="Asintomática"],na.rm=TRUE),1)),
c(round(min(I[data$Síntomas=="Leve"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Leve"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Leve"],na.rm=TRUE),1),
round(median(I[data$Síntomas=="Leve"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Leve"],c(0.25,0.75),na.rm=TRUE),1),round(sd(I[data$Síntomas=="Leve"],na.rm=TRUE),1)),
c(round(min(I[data$Síntomas=="Grave"],na.rm=TRUE),1),round(max(I[data$Síntomas=="Grave"],na.rm=TRUE),1), round(mean(I[data$Síntomas=="Grave"],na.rm=TRUE),1),round(median(I[data$Síntomas=="Grave"],na.rm=TRUE),1),
round(quantile(I[data$Síntomas=="Grave"],c(0.25,0.75),na.rm=TRUE),1),round(sd(I[data$Síntomas=="Grave"],na.rm=TRUE),1)) )
colnames(Dades)=c("Peso. mínimo","Peso. máximo","Peso. medio","Peso mediano", "1er cuartil", "3er cuartil","Desv. típica")
rownames(Dades)=c("Asintomática" ,"Leve", "Grave" )
Dades %>%
kbl() %>%
kable_styling() %>%
scroll_box(width="100%", box_css="border: 0px;")| Peso. mínimo | Peso. máximo | Peso. medio | Peso mediano | 1er cuartil | 3er cuartil | Desv. típica | |
|---|---|---|---|---|---|---|---|
| Asintomática | 315 | 4640 | 3231.0 | 3275 | 2945 | 3550 | 553.7 |
| Leve | 680 | 5190 | 3137.3 | 3220 | 2835 | 3510 | 624.7 |
| Grave | 680 | 4400 | 3062.4 | 3140 | 2790 | 3500 | 686.3 |
Ajuste de los pesos de cada nivel a distribuciones normales: test de Shapiro-Wilks, p-valores \(9\times 10^{-10}\), \(2\times 10^{-8}\), \(10^{-7}\), respectivamente
Homocedasticidad: Test de Fligner-Killeen, p-valor 0.06
Pesos medios: test de Kruskal-Wallis, p-valor 0.061
4.2.21 Bajo peso
I=c(Casos[Casos$Feto.muerto.intraútero=="No",]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí",]$Peso._gramos_...148)
I.cut=cut(I,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
I.cut=ordered(I.cut,levels=c(0,1))
IA=c(Casos[Casos$Feto.muerto.intraútero=="No" &Casos$SINTOMAS_DIAGNOSTICO=="Asintomática" ,]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí"&CasosGM$SINTOMAS_DIAGNOSTICO=="Asintomática",]$Peso._gramos_...148)
IA.cut=cut(IA,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
IA.cut=ordered(IA.cut,levels=c(0,1))
IL=c(Casos[Casos$Feto.muerto.intraútero=="No" &Casos$SINTOMAS_DIAGNOSTICO=="Leve" ,]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí"&CasosGM$SINTOMAS_DIAGNOSTICO=="Leve",]$Peso._gramos_...148)
IL.cut=cut(IL,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
IL.cut=ordered(IL.cut,levels=c(0,1))
IG=c(Casos[Casos$Feto.muerto.intraútero=="No" &Casos$SINTOMAS_DIAGNOSTICO=="Grave" ,]$Peso._gramos_...125,CasosGM[CasosGM$Feto.vivo=="Sí"&CasosGM$SINTOMAS_DIAGNOSTICO=="Grave",]$Peso._gramos_...148)
IG.cut=cut(IG,breaks=c(0,2500,10000),labels=c(1,0),right=FALSE)
IG.cut=ordered(IG.cut,levels=c(0,1))
SintN=c(Casos[Casos$Feto.muerto.intraútero=="No",]$SINTOMAS_DIAGNOSTICO,CasosGM[CasosGM$Feto.vivo=="Sí",]$SINTOMAS_DIAGNOSTICO)
taula=table(data.frame(I.cut,SintN))[c(2,1) ,1:3]
Tabla.DMCasosr(IA.cut,IL.cut,IG.cut,"Bajo peso", "No bajo peso")| Asintomáticas (N) | Asintomáticas (%) | Leves (N) | Leves (%) | Graves (N) | Graves (%) | p-valor | Tipo | |
|---|---|---|---|---|---|---|---|---|
| Bajo peso | 30 | 8.6 | 38 | 12.1 | 32 | 15.6 | 0.039705 | Paramétrico |
| No bajo peso | 320 | 91.4 | 277 | 87.9 | 173 | 84.4 | ||
| Datos perdidos | 5 | 7 | 4 |
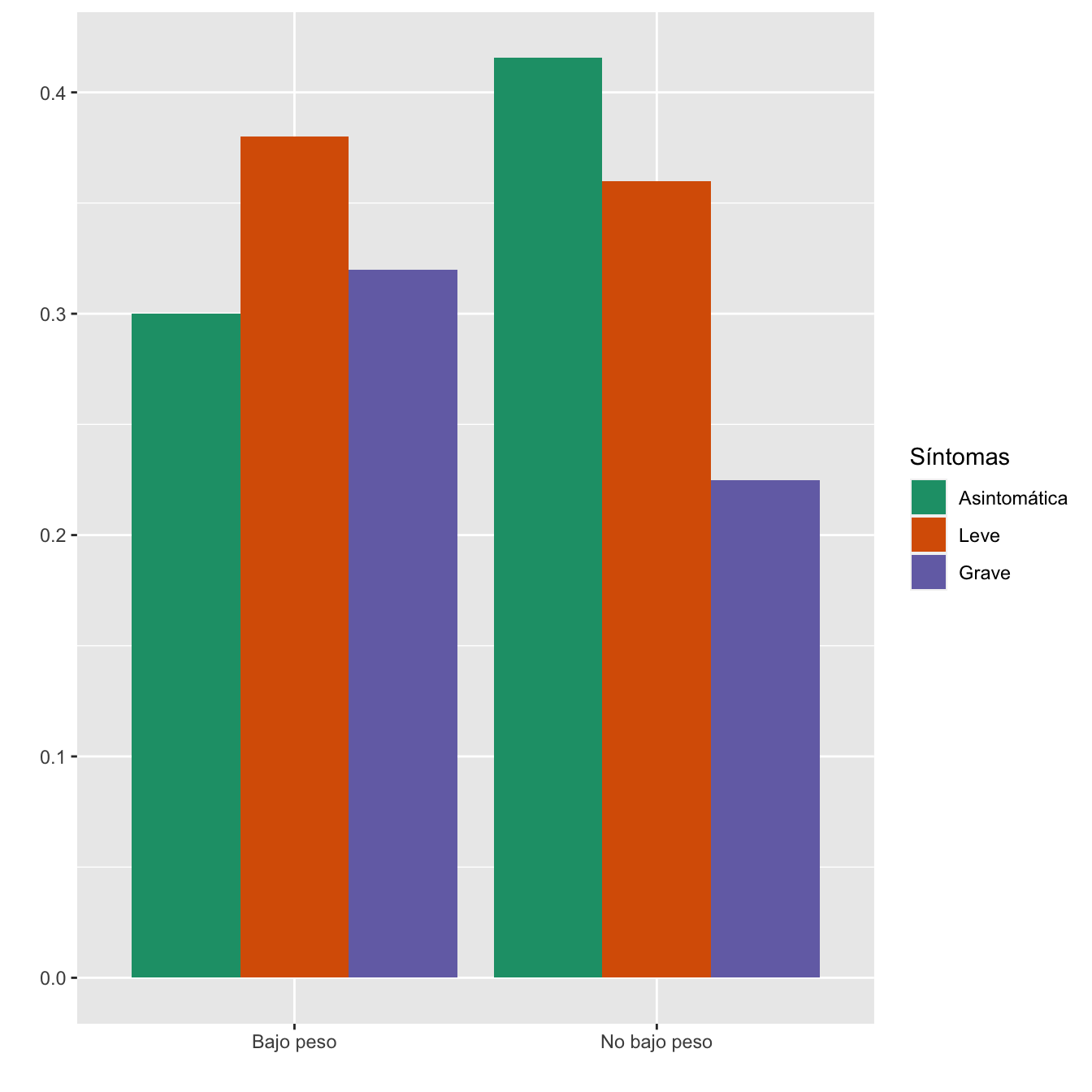
- Potencia del test: 0.617